+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 7wwu | ||||||
|---|---|---|---|---|---|---|---|
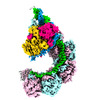
| タイトル | ICP1 Csy complex | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | RNA BINDING PROTEIN/RNA / CRISPR-Cas system / RNA / RNA BINDING PROTEIN / RNA BINDING PROTEIN-RNA complex | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | ||||||
| 生物種 |  Vibrio phage ICP1_2011_A (ファージ) Vibrio phage ICP1_2011_A (ファージ) | ||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.5 Å | ||||||
 データ登録者 データ登録者 | Zhang, M. / Peng, R. | ||||||
| 資金援助 |  中国, 1件 中国, 1件
| ||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2023 ジャーナル: Proc Natl Acad Sci U S A / 年: 2023タイトル: Mechanistic insights into DNA binding and cleavage by a compact type I-F CRISPR-Cas system in bacteriophage. 著者: Manling Zhang / Ruchao Peng / Qi Peng / Sheng Liu / Zhiteng Li / Yuqin Zhang / Hao Song / Jia Yang / Xiao Xing / Peiyi Wang / Jianxun Qi / George F Gao /  要旨: CRISPR-Cas systems are widespread adaptive antiviral systems used in prokaryotes. Some phages, in turn, although have small genomes can economize the use of genetic space to encode compact or ...CRISPR-Cas systems are widespread adaptive antiviral systems used in prokaryotes. Some phages, in turn, although have small genomes can economize the use of genetic space to encode compact or incomplete CRISPR-Cas systems to inhibit the host and establish infection. Phage ICP1, infecting , encodes a compact type I-F CRISPR-Cas system to suppress the antiphage mobile genetic element in the host genome. However, the mechanism by which this compact system recognizes the target DNA and executes interference remains elusive. Here, we present the electron cryo-microscopy (cryo-EM) structures of both apo- and DNA-bound ICP1 surveillance complexes (Aka Csy complex). Unlike most other type I surveillance complexes, the ICP1 Csy complex lacks the Cas11 subunit or a structurally homologous domain, which is crucial for dsDNA binding and Cas3 activation in other type I CRISPR-Cas systems. Structural and functional analyses revealed that the compact ICP1 Csy complex alone is inefficient in binding to dsDNA targets, presumably stalled at a partial R-loop conformation. The presence of Cas2/3 facilitates dsDNA binding and allows effective dsDNA target cleavage. Additionally, we found that Cas2/3 efficiently cleaved the dsDNA target presented by the ICP1 Csy complex, but not vice versa. These findings suggest a unique mechanism for target dsDNA binding and cleavage by the compact phage-derived CRISPR-Cas system. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  7wwu.cif.gz 7wwu.cif.gz | 462 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb7wwu.ent.gz pdb7wwu.ent.gz | 370.2 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  7wwu.json.gz 7wwu.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  7wwu_validation.pdf.gz 7wwu_validation.pdf.gz | 868.2 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  7wwu_full_validation.pdf.gz 7wwu_full_validation.pdf.gz | 894.2 KB | 表示 | |
| XML形式データ |  7wwu_validation.xml.gz 7wwu_validation.xml.gz | 67.1 KB | 表示 | |
| CIF形式データ |  7wwu_validation.cif.gz 7wwu_validation.cif.gz | 106.1 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/ww/7wwu https://data.pdbj.org/pub/pdb/validation_reports/ww/7wwu ftp://data.pdbj.org/pub/pdb/validation_reports/ww/7wwu ftp://data.pdbj.org/pub/pdb/validation_reports/ww/7wwu | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 22841.510 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)  Vibrio phage ICP1_2011_A (ファージ) Vibrio phage ICP1_2011_A (ファージ)遺伝子: csy1, ICP12011A_088 発現宿主:  参照: UniProt: M1R2X3 | ||||
|---|---|---|---|---|---|
| #2: タンパク質 | 分子量: 29833.129 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)  Vibrio phage ICP1_2011_A (ファージ) Vibrio phage ICP1_2011_A (ファージ)遺伝子: csy2, ICP12011A_087 発現宿主:  参照: UniProt: M1QWL5 | ||||
| #3: タンパク質 | 分子量: 35830.867 Da / 分子数: 6 / 由来タイプ: 組換発現 由来: (組換発現)  Vibrio phage ICP1_2011_A (ファージ) Vibrio phage ICP1_2011_A (ファージ)遺伝子: csy3, ICP12011A_086 発現宿主:  参照: UniProt: M1Q7R8 #4: タンパク質 | | 分子量: 21168.271 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)  Vibrio phage ICP1_2011_A (ファージ) Vibrio phage ICP1_2011_A (ファージ)遺伝子: csy4, ICP12011A_085 発現宿主:  参照: UniProt: M1R9H3 #5: RNA鎖 | | 分子量: 19351.408 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)  Vibrio phage ICP1_2011_A (ファージ) Vibrio phage ICP1_2011_A (ファージ)発現宿主:  参照: GenBank: 452118997 |
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: apo-ICP1 Csy complex / タイプ: COMPLEX / Entity ID: all / 由来: RECOMBINANT |
|---|---|
| 由来(天然) | 生物種:  Vibrio phage ICP1_2011_A (ファージ) Vibrio phage ICP1_2011_A (ファージ) |
| 由来(組換発現) | 生物種:  |
| 緩衝液 | pH: 7.5 |
| 試料 | 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES |
| 急速凍結 | 凍結剤: ETHANE |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: SPOT SCAN FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: SPOT SCAN |
| 電子レンズ | モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2500 nm / 最小 デフォーカス(公称値): 1800 nm |
| 撮影 | 電子線照射量: 60 e/Å2 フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) |
- 解析
解析
| ソフトウェア | 名称: PHENIX / バージョン: 1.19.2_4158: / 分類: 精密化 | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||
| 3次元再構成 | 解像度: 3.5 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 463686 / 対称性のタイプ: POINT | ||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー







 PDBj
PDBj





























