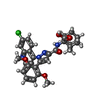
登録情報 データベース : PDB / ID : 7ul2タイトル CryoEM Structure of Inactive NTSR1 Bound to SR48692 and Nb6 Nanobody 6 Neurotensin receptor 1 キーワード / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Homo sapiens (ヒト)Lama glama (ラマ)手法 / / / 解像度 : 2.4 Å データ登録者 Robertson, M.J. / Skiniotis, G. 資金援助 組織 認可番号 国 The G. Harold and Leila Y. Mathers Foundation
ジャーナル : Nat Struct Mol Biol / 年 : 2022タイトル : Structure determination of inactive-state GPCRs with a universal nanobody.著者 : Michael J Robertson / Makaía M Papasergi-Scott / Feng He / Alpay B Seven / Justin G Meyerowitz / Ouliana Panova / Maria Claudia Peroto / Tao Che / Georgios Skiniotis / 要旨 : Cryogenic electron microscopy (cryo-EM) has widened the field of structure-based drug discovery by allowing for routine determination of membrane protein structures previously intractable. Despite ... Cryogenic electron microscopy (cryo-EM) has widened the field of structure-based drug discovery by allowing for routine determination of membrane protein structures previously intractable. Despite representing one of the largest classes of therapeutic targets, most inactive-state G protein-coupled receptors (GPCRs) have remained inaccessible for cryo-EM because their small size and membrane-embedded nature impedes projection alignment for high-resolution map reconstructions. Here we demonstrate that the same single-chain camelid antibody (nanobody) recognizing a grafted intracellular loop can be used to obtain cryo-EM structures of inactive-state GPCRs at resolutions comparable or better than those obtained by X-ray crystallography. Using this approach, we obtained structures of neurotensin 1 receptor bound to antagonist SR48692, μ-opioid receptor bound to alvimopan, apo somatostatin receptor 2 and histamine receptor 2 bound to famotidine. We expect this rapid, straightforward approach to facilitate the broad exploration of GPCR inactive states without the need for extensive engineering and crystallization. 履歴 登録 2022年4月3日 登録サイト / 処理サイト 改定 1.0 2022年6月29日 Provider / タイプ 改定 1.1 2022年11月23日 Group / カテゴリ Item _citation.country / _citation.journal_abbrev ... _citation.country / _citation.journal_abbrev / _citation.journal_id_CSD / _citation.journal_id_ISSN / _citation.pdbx_database_id_DOI / _citation.year 改定 1.2 2022年11月30日 Group / カテゴリ / Item / _citation.title改定 1.3 2022年12月28日 Group / カテゴリ / citation_authorItem _citation.journal_volume / _citation.page_first ... _citation.journal_volume / _citation.page_first / _citation.page_last / _citation_author.identifier_ORCID
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト)
 データ登録者
データ登録者 米国, 1件
米国, 1件  引用
引用 ジャーナル: Nat Struct Mol Biol / 年: 2022
ジャーナル: Nat Struct Mol Biol / 年: 2022
 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 7ul2.cif.gz
7ul2.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb7ul2.ent.gz
pdb7ul2.ent.gz PDB形式
PDB形式 7ul2.json.gz
7ul2.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 7ul2_validation.pdf.gz
7ul2_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 7ul2_full_validation.pdf.gz
7ul2_full_validation.pdf.gz 7ul2_validation.xml.gz
7ul2_validation.xml.gz 7ul2_validation.cif.gz
7ul2_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/ul/7ul2
https://data.pdbj.org/pub/pdb/validation_reports/ul/7ul2 ftp://data.pdbj.org/pub/pdb/validation_reports/ul/7ul2
ftp://data.pdbj.org/pub/pdb/validation_reports/ul/7ul2 リンク
リンク 集合体
集合体
 要素
要素 Homo sapiens (ヒト) / 遺伝子: NTSR1, NTRR, OPRK1, OPRK
Homo sapiens (ヒト) / 遺伝子: NTSR1, NTRR, OPRK1, OPRK


 試料調製
試料調製 電子顕微鏡撮影
電子顕微鏡撮影
 FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: OTHER
FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: OTHER 解析
解析 ムービー
ムービー コントローラー
コントローラー








 PDBj
PDBj