+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 7u5c | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of human CST bound to DNA polymerase alpha-primase in a recruitment state | |||||||||
 要素 要素 |
| |||||||||
 キーワード キーワード | DNA BINDING PROTEIN/DNA / Fill-in / Telomere / Replication / DNA BINDING PROTEIN / DNA BINDING PROTEIN-DNA complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報CST complex / positive regulation of DNA primase activity / DNA primase AEP / ribonucleotide binding / telomerase inhibitor activity / DNA replication initiation / DNA/RNA hybrid binding / telomere maintenance via telomere lengthening / Telomere C-strand synthesis initiation / Inhibition of replication initiation of damaged DNA by RB1/E2F1 ...CST complex / positive regulation of DNA primase activity / DNA primase AEP / ribonucleotide binding / telomerase inhibitor activity / DNA replication initiation / DNA/RNA hybrid binding / telomere maintenance via telomere lengthening / Telomere C-strand synthesis initiation / Inhibition of replication initiation of damaged DNA by RB1/E2F1 / alpha DNA polymerase:primase complex / Polymerase switching / regulation of type I interferon production / Processive synthesis on the lagging strand / DNA primase activity / Removal of the Flap Intermediate / single-stranded telomeric DNA binding / Polymerase switching on the C-strand of the telomere / lagging strand elongation / G-rich strand telomeric DNA binding / DNA replication, synthesis of primer / telomere capping / mitotic DNA replication initiation / bone marrow development / intermediate filament cytoskeleton / hematopoietic stem cell proliferation / DNA strand elongation involved in DNA replication / DNA synthesis involved in DNA repair / G1/S-Specific Transcription / telomeric DNA binding / leading strand elongation / DNA replication origin binding / negative regulation of telomere maintenance via telomerase / replicative senescence / Activation of the pre-replicative complex / DNA replication initiation / spleen development / regulation of G2/M transition of mitotic cell cycle / telomere maintenance / thymus development / positive regulation of DNA replication / Defective pyroptosis / multicellular organism growth / fibrillar center / nuclear matrix / double-strand break repair via nonhomologous end joining / protein import into nucleus / positive regulation of fibroblast proliferation / nuclear envelope / single-stranded DNA binding / 4 iron, 4 sulfur cluster binding / DNA replication / chromosome, telomeric region / DNA-directed DNA polymerase / DNA-directed DNA polymerase activity / intracellular membrane-bounded organelle / DNA repair / nucleotide binding / DNA damage response / chromatin binding / chromatin / nucleolus / protein kinase binding / magnesium ion binding / DNA binding / zinc ion binding / nucleoplasm / membrane / nucleus / metal ion binding / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.6 Å | |||||||||
 データ登録者 データ登録者 | Cai, S.W. / Zinder, J.C. / Svetlov, V. / Bush, M.W. / Nudler, E. / Walz, T. / de Lange, T. | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2022 ジャーナル: Nat Struct Mol Biol / 年: 2022タイトル: Cryo-EM structure of the human CST-Polα/primase complex in a recruitment state. 著者: Sarah W Cai / John C Zinder / Vladimir Svetlov / Martin W Bush / Evgeny Nudler / Thomas Walz / Titia de Lange /  要旨: The CST-Polα/primase complex is essential for telomere maintenance and functions to counteract resection at double-strand breaks. We report a 4.6-Å resolution cryo-EM structure of human CST- ...The CST-Polα/primase complex is essential for telomere maintenance and functions to counteract resection at double-strand breaks. We report a 4.6-Å resolution cryo-EM structure of human CST-Polα/primase, captured prior to catalysis in a recruitment state stabilized by chemical cross-linking. Our structure reveals an evolutionarily conserved interaction between the C-terminal domain of the catalytic POLA1 subunit and an N-terminal expansion in metazoan CTC1. Cross-linking mass spectrometry and negative-stain EM analysis provide insight into CST binding by the flexible POLA1 N-terminus. Finally, Coats plus syndrome disease mutations previously characterized to disrupt formation of the CST-Polα/primase complex map to protein-protein interfaces observed in the recruitment state. Together, our results shed light on the architecture and stoichiometry of the metazoan fill-in machinery. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  7u5c.cif.gz 7u5c.cif.gz | 692.5 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb7u5c.ent.gz pdb7u5c.ent.gz | 545.9 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  7u5c.json.gz 7u5c.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  7u5c_validation.pdf.gz 7u5c_validation.pdf.gz | 1.4 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  7u5c_full_validation.pdf.gz 7u5c_full_validation.pdf.gz | 1.5 MB | 表示 | |
| XML形式データ |  7u5c_validation.xml.gz 7u5c_validation.xml.gz | 125.5 KB | 表示 | |
| CIF形式データ |  7u5c_validation.cif.gz 7u5c_validation.cif.gz | 187.8 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/u5/7u5c https://data.pdbj.org/pub/pdb/validation_reports/u5/7u5c ftp://data.pdbj.org/pub/pdb/validation_reports/u5/7u5c ftp://data.pdbj.org/pub/pdb/validation_reports/u5/7u5c | HTTPS FTP |
-関連構造データ
| 関連構造データ | 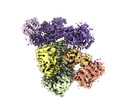 26346MC M: このデータのモデリングに利用したマップデータ C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
| 電子顕微鏡画像生データ |  EMPIAR-11131 (タイトル: Single particle cryo-EM of the human CST•Polα/Primase (POLA1ΔN) complex in a recruitment state EMPIAR-11131 (タイトル: Single particle cryo-EM of the human CST•Polα/Primase (POLA1ΔN) complex in a recruitment stateData size: 869.0 Data #1: Motion corrected micrographs of CST-Pola/Primase deltaN complex in recruitment state [micrographs - single frame]) |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
-タンパク質 , 2種, 2分子 AB
| #1: タンパク質 | 分子量: 49981.012 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: PRIM1 / 発現宿主: Homo sapiens (ヒト) / 遺伝子: PRIM1 / 発現宿主:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ)参照: UniProt: P49642, 転移酵素; リンを含む基を移すもの; 核酸を移すもの |
|---|---|
| #2: タンパク質 | 分子量: 59092.102 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: PRIM2, PRIM2A / 発現宿主: Homo sapiens (ヒト) / 遺伝子: PRIM2, PRIM2A / 発現宿主:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ)参照: UniProt: P49643, 転移酵素; リンを含む基を移すもの; 核酸を移すもの |
-DNA polymerase alpha ... , 2種, 2分子 CD
| #3: タンパク質 | 分子量: 129635.125 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: POLA1, POLA / 発現宿主: Homo sapiens (ヒト) / 遺伝子: POLA1, POLA / 発現宿主:  Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: P09884, DNA-directed DNA polymerase Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: P09884, DNA-directed DNA polymerase |
|---|---|
| #4: タンパク質 | 分子量: 66015.539 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: POLA2 / 発現宿主: Homo sapiens (ヒト) / 遺伝子: POLA2 / 発現宿主:  Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: Q14181 Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: Q14181 |
-CST complex subunit ... , 3種, 3分子 EFG
| #5: タンパク質 | 分子量: 135050.328 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: CTC1, C17orf68 / 発現宿主: Homo sapiens (ヒト) / 遺伝子: CTC1, C17orf68 / 発現宿主:  Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: Q2NKJ3 Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: Q2NKJ3 |
|---|---|
| #6: タンパク質 | 分子量: 42172.949 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: STN1, OBFC1 / 発現宿主: Homo sapiens (ヒト) / 遺伝子: STN1, OBFC1 / 発現宿主:  Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: Q9H668 Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: Q9H668 |
| #7: タンパク質 | 分子量: 13872.013 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: TEN1, C17orf106 / 発現宿主: Homo sapiens (ヒト) / 遺伝子: TEN1, C17orf106 / 発現宿主:  Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: Q86WV5 Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: Q86WV5 |
-DNA鎖 , 1種, 1分子 H
| #8: DNA鎖 | 分子量: 5682.672 Da / 分子数: 1 / 由来タイプ: 合成 / 由来: (合成)  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
-非ポリマー , 2種, 5分子 


| #9: 化合物 | ChemComp-ZN / #10: 化合物 | ChemComp-SF4 / | |
|---|
-詳細
| 研究の焦点であるリガンドがあるか | Y |
|---|
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: Human CST bound to DNA polymerase alpha(POLA1 delta N)-primase in a recruitment state タイプ: COMPLEX / 詳細: Sample was GraFix cross-linked with glutaraldehyde. / Entity ID: #1-#8 / 由来: MULTIPLE SOURCES |
|---|---|
| 分子量 | 値: 0.5 MDa / 実験値: NO |
| 緩衝液 | pH: 7.5 |
| 試料 | 濃度: 0.075 mg/ml / 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES 詳細: The sample was cross-linked with glutaraldehyde (GraFix) |
| 試料支持 | グリッドの材料: GOLD / グリッドのタイプ: Quantifoil R1.2/1.3 |
| 急速凍結 | 装置: FEI VITROBOT MARK IV / 凍結剤: ETHANE / 湿度: 100 % / 凍結前の試料温度: 278 K |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: OTHER FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: OTHER |
| 電子レンズ | モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2200 nm / 最小 デフォーカス(公称値): 1000 nm |
| 撮影 | 電子線照射量: 52 e/Å2 / フィルム・検出器のモデル: GATAN K3 (6k x 4k) |
- 解析
解析
| EMソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| CTF補正 | タイプ: NONE | ||||||||||||||||||||||||||||||||||||||||
| 3次元再構成 | 解像度: 4.6 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 131850 / 対称性のタイプ: POINT | ||||||||||||||||||||||||||||||||||||||||
| 原子モデル構築 |
|
 ムービー
ムービー コントローラー
コントローラー




 PDBj
PDBj
















































