+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-9891 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of spike protein of feline infectious peritonitis virus strain UU4 | ||||||||||||
 マップデータ マップデータ | FIPV-UU4 spike protein (sharpened) | ||||||||||||
 試料 試料 |
| ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報: / endocytosis involved in viral entry into host cell / membrane => GO:0016020 / host cell endoplasmic reticulum-Golgi intermediate compartment membrane / receptor-mediated virion attachment to host cell / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / viral envelope / virion membrane 類似検索 - 分子機能 | ||||||||||||
| 生物種 |  Feline infectious peritonitis virus (ウイルス) Feline infectious peritonitis virus (ウイルス) | ||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.31 Å | ||||||||||||
 データ登録者 データ登録者 | Hsu STD / Yang TJ / Ko TP / Draczkowski P | ||||||||||||
| 資金援助 |  台湾, 3件 台湾, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2020 ジャーナル: Proc Natl Acad Sci U S A / 年: 2020タイトル: Cryo-EM analysis of a feline coronavirus spike protein reveals a unique structure and camouflaging glycans. 著者: Tzu-Jing Yang / Yen-Chen Chang / Tzu-Ping Ko / Piotr Draczkowski / Yu-Chun Chien / Yuan-Chih Chang / Kuen-Phon Wu / Kay-Hooi Khoo / Hui-Wen Chang / Shang-Te Danny Hsu /  要旨: Feline infectious peritonitis virus (FIPV) is an alphacoronavirus that causes a nearly 100% mortality rate without effective treatment. Here we report a 3.3-Å cryoelectron microscopy (cryo-EM) ...Feline infectious peritonitis virus (FIPV) is an alphacoronavirus that causes a nearly 100% mortality rate without effective treatment. Here we report a 3.3-Å cryoelectron microscopy (cryo-EM) structure of the serotype I FIPV spike (S) protein, which is responsible for host recognition and viral entry. Mass spectrometry provided site-specific compositions of densely distributed high-mannose and complex-type Nglycans that account for 1/4 of the total molecular mass; most of the N-glycans could be visualized by cryo-EM. Specifically, the N-glycans that wedge between 2 galectin-like domains within the S1 subunit of FIPV S protein result in a unique propeller-like conformation, underscoring the importance of glycosylation in maintaining protein structures. The cleavage site within the S2 subunit responsible for activation also showed distinct structural features and glycosylation. These structural insights provide a blueprint for a better molecular understanding of the pathogenesis of FIP. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_9891.map.gz emd_9891.map.gz | 301.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-9891-v30.xml emd-9891-v30.xml emd-9891.xml emd-9891.xml | 21.2 KB 21.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_9891.png emd_9891.png | 32.3 KB | ||
| その他 |  emd_9891_additional.map.gz emd_9891_additional.map.gz emd_9891_additional_1.map.gz emd_9891_additional_1.map.gz emd_9891_half_map_1.map.gz emd_9891_half_map_1.map.gz emd_9891_half_map_2.map.gz emd_9891_half_map_2.map.gz | 301.1 MB 301.1 MB 85 MB 85 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-9891 http://ftp.pdbj.org/pub/emdb/structures/EMD-9891 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9891 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9891 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_9891_validation.pdf.gz emd_9891_validation.pdf.gz | 758.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_9891_full_validation.pdf.gz emd_9891_full_validation.pdf.gz | 758.4 KB | 表示 | |
| XML形式データ |  emd_9891_validation.xml.gz emd_9891_validation.xml.gz | 17.4 KB | 表示 | |
| CIF形式データ |  emd_9891_validation.cif.gz emd_9891_validation.cif.gz | 20.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9891 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9891 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9891 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9891 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_9891.map.gz / 形式: CCP4 / 大きさ: 325 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_9891.map.gz / 形式: CCP4 / 大きさ: 325 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | FIPV-UU4 spike protein (sharpened) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.87 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: FIPV-UU4 spike protein (unsharpened)
| ファイル | emd_9891_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | FIPV-UU4 spike protein (unsharpened) | ||||||||||||
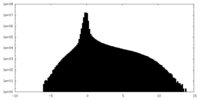
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: FIPV-UU4 spike protein (unsharpened)
| ファイル | emd_9891_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | FIPV-UU4 spike protein (unsharpened) | ||||||||||||
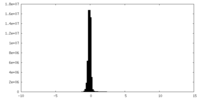
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map 1
| ファイル | emd_9891_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half_map_1 | ||||||||||||
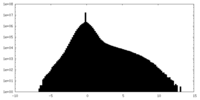
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map 2
| ファイル | emd_9891_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half_map_2 | ||||||||||||
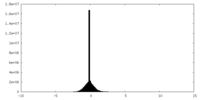
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Feline Infectious Peritonitis Virus Spike Protein
| 全体 | 名称: Feline Infectious Peritonitis Virus Spike Protein |
|---|---|
| 要素 |
|
-超分子 #1: Feline Infectious Peritonitis Virus Spike Protein
| 超分子 | 名称: Feline Infectious Peritonitis Virus Spike Protein / タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Feline infectious peritonitis virus (ウイルス) Feline infectious peritonitis virus (ウイルス) |
| 分子量 | 実験値: 720 kDa/nm |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Feline Infectious Peritonitis Virus Spike Protein
| 分子 | 名称: Feline Infectious Peritonitis Virus Spike Protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Feline infectious peritonitis virus (ウイルス) Feline infectious peritonitis virus (ウイルス) |
| 分子量 | 理論値: 163.829844 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: DAPHGVTLPH FNTSHNNSKF ELNFYNFLQT WDIPPNTETI LGGYLPYCDH EDNCGWYNFV YNNKVGPNAK YSYINTQNLN IPNVHGVYF DVREHNSDGV WDQIDRVGLL IAIHGTSHYS LLMVLQDGVE ASQPHVAVKI CHWNPGNIST YHQFDVNLGD G GQCVFNQR ...文字列: DAPHGVTLPH FNTSHNNSKF ELNFYNFLQT WDIPPNTETI LGGYLPYCDH EDNCGWYNFV YNNKVGPNAK YSYINTQNLN IPNVHGVYF DVREHNSDGV WDQIDRVGLL IAIHGTSHYS LLMVLQDGVE ASQPHVAVKI CHWNPGNIST YHQFDVNLGD G GQCVFNQR FSLDTVLTAN DFYGFQWTDT YVDIYLGGTI TKVWVVNDWS VVEASISSHW NALNYGYYIQ FVNRTTYYAY NS TGGSNYT HLQLTECHTD YCAGYAKNVF VPIDGKIPEG FSFSNWFLLT DKSTLVQGRV LSSQPVFVQC LRPVPTWSNN TAV VHFKND VFCPNVTADV LRFNLNFSDT DVYTDSTTDD QLHFTFEDNT TASITCYSSA NVTDNQPASG SISHTPFVSN SYLC FANFS HSSVSRQFLG ILPPTVREFA FGRDGSIFVN GYKYFSLQPI KSVNFSISSV ENYGFWTIAY TNYTDVMVDV NGTVI TRLF YCDSPLNRIK CQQLKHELPD GFYSASMLVK KDLPKTFVTM PQFYNWMNVT LHVVLNDIEK KADIILAGAP ELASLA DIH FEIAQANGSV VNVTSVCVQA RQLALFYKYT SLQGLYTYSN LVQLQNYDCP FSPQQFNNYL QFETLCFDVS PAVAGCK WS LVHDVKWRTQ FATITVSYKD GAMITTMPKA QLGFQDISNI VKDECTDYNI YGFQGTGIIR STTSRLVAGL YYTSASGD L LGFKISTTGE IFTVVPCDLT AQAAVINDEI VGAITATNQT DLFEFVNHTW SRSARGSSPS TVNTYTMPQF YYITKWNNG TSSNCTSVIT YSSFAICNTG EIKYVNVTHV EIVDDSVGVI KPVSTGNITI PKNFTVAVQA EYVQIQVKPV AVDCAKYVCN GNRHCLNLL TQYTSACQTI ENSLNLGARL ESLMLNDMIT VSDRSLEFAT VDKFNTTALG GEKLGGLYFD GLSSLLPPRV G MRSAVEDL LFNKVVTSGL GTVDDDYKKC SAGTDVADLV CAQYYNGIMV LPGVVDYNKM AMYTASLIGG MALGSITSAV AV PFSMQVQ ARLNYVALQT DVLQENQKIL ANAFNNAIGN ITLALGKVSN AITTVSDGFN SMASALTKIQ SVVNQQGEAL SHL ISQLQK NFQAISSSIA EIYNRLEKVE ADAQVDRLIT GRLAALNAYV AQTLTQYAEV KASRQLAMEK VNECVKSQSD RYGF CGNGT HLFSLVNSAP DGLLFFHTVL LPTEWEEVTA WSGICVNDTY AYLLKDFDHS IFSYNGTYMV TPRNMFQPRK PQMSD FVQI TSCEVTFLNT THTTFQEIVI DYIDINKTIA DMLEQYHSNY TTPELDLQLE IFNQTKLNLT AEIDQLEQRA DNLTNI AHE LQQYIDNLNK TIVDLEWLNR IETYVKWPGS GGYIPEAPRD GQAYVRKDGE WVLLSTFLKG QDNSADIQHS GGRSSLE GP RFEGKPIPNP LLGLDSTRTG HHHHHH |
-分子 #9: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 9 / コピー数: 27 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-分子 #10: alpha-D-mannopyranose
| 分子 | 名称: alpha-D-mannopyranose / タイプ: ligand / ID: 10 / コピー数: 6 / 式: MAN |
|---|---|
| 分子量 | 理論値: 180.156 Da |
| Chemical component information |  ChemComp-MAN: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.2 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 / 詳細: 1X D-PBS |
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 200 / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV 詳細: blot for 3 seconds before plunging; blot force: 0; waiting time: 10s. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: INTEGRATING / 実像数: 2436 / 平均露光時間: 2.5 sec. / 平均電子線量: 1.5 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー













 Z
Z Y
Y X
X