+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-8854 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of the major capsid protein and the capsid stabilizing protein of the marine siphovirus TW1 | |||||||||
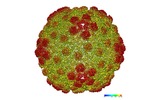
 マップデータ マップデータ | icosahedral reconstruction of marine Siphophage TW1 head | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Major Capsid Protein / Capsid Stabilizing Protein / HK 97 fold / Decoration Protein /  VIRUS (ウイルス) VIRUS (ウイルス) | |||||||||
| 機能・相同性 |  カプシド / Putative coat protein / Uncharacterized protein カプシド / Putative coat protein / Uncharacterized protein 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Pseudoalteromonas phage TW1 (ファージ) Pseudoalteromonas phage TW1 (ファージ) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.6 Å クライオ電子顕微鏡法 / 解像度: 3.6 Å | |||||||||
 データ登録者 データ登録者 | Wang Z / Rossmann MG | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2018 ジャーナル: Structure / 年: 2018タイトル: Structure of the Marine Siphovirus TW1: Evolution of Capsid-Stabilizing Proteins and Tail Spikes. 著者: Zhiqing Wang / Stephen C Hardies / Andrei Fokine / Thomas Klose / Wen Jiang / Byung Cheol Cho / Michael G Rossmann /   要旨: Marine bacteriophage TW1 belongs to the Siphoviridae family and infects Pseudoalteromonas phenolica. Mass spectrometry analysis has identified 16 different proteins in the TW1 virion. Functions of ...Marine bacteriophage TW1 belongs to the Siphoviridae family and infects Pseudoalteromonas phenolica. Mass spectrometry analysis has identified 16 different proteins in the TW1 virion. Functions of most of these proteins have been predicted by bioinformatic methods. A 3.6 Å resolution cryoelectron microscopy map of the icosahedrally averaged TW1 head showed the atomic structures of the major capsid protein, gp57, and the capsid-stabilizing protein, gp56. The gp57 structure is similar to that of the phage HK97 capsid protein. The gp56 protein has two domains, each having folds similar to that of the N-terminal part of phage λ gpD, indicating a common ancestry. The first gp56 domain clamps adjacent capsomers together, whereas the second domain is required for trimerization. A 6-fold-averaged reconstruction of the distal part of the tail showed that TW1 has six tail spikes, which are unusual for siphophages but are similar to the podophages P22 and Sf6, suggesting a common evolutionary origin of these spikes. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_8854.map.gz emd_8854.map.gz | 2.9 GB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-8854-v30.xml emd-8854-v30.xml emd-8854.xml emd-8854.xml | 13.5 KB 13.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_8854.png emd_8854.png | 297.7 KB | ||
| Filedesc metadata |  emd-8854.cif.gz emd-8854.cif.gz | 5.3 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-8854 http://ftp.pdbj.org/pub/emdb/structures/EMD-8854 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8854 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8854 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_8854.map.gz / 形式: CCP4 / 大きさ: 11.1 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_8854.map.gz / 形式: CCP4 / 大きさ: 11.1 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | icosahedral reconstruction of marine Siphophage TW1 head | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
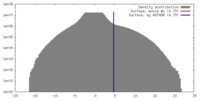
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.667 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Pseudoalteromonas phage TW1
| 全体 | 名称:  Pseudoalteromonas phage TW1 (ファージ) Pseudoalteromonas phage TW1 (ファージ) |
|---|---|
| 要素 |
|
-超分子 #1: Pseudoalteromonas phage TW1
| 超分子 | 名称: Pseudoalteromonas phage TW1 / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all / NCBI-ID: 1366055 / 生物種: Pseudoalteromonas phage TW1 / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|
-分子 #1: Capsid Stabilizing Protein
| 分子 | 名称: Capsid Stabilizing Protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 7 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Pseudoalteromonas phage TW1 (ファージ) Pseudoalteromonas phage TW1 (ファージ) |
| 分子量 | 理論値: 15.017777 KDa |
| 配列 | 文字列: MANSKNSIFV GGAGRVKQTI EGLAQSAFKP GQLLARAAGD AIDVTAKAST TYGNEFLICD DQPQTLGGGT DVAVTAGDTV QAISVLPGQ YVLLSFAATQ NVTTKGAAVA SNGDGNFKLG NPATEQTFAV TEEIINVTTA GTLVLCRAI UniProtKB: Uncharacterized protein |
-分子 #2: Major Capsid Protein
| 分子 | 名称: Major Capsid Protein / タイプ: protein_or_peptide / ID: 2 / コピー数: 7 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Pseudoalteromonas phage TW1 (ファージ) Pseudoalteromonas phage TW1 (ファージ) |
| 分子量 | 理論値: 39.047965 KDa |
| 配列 | 文字列: MSIHFDFKNK QGKELLALNR QWNELSGYRA HAFNESVNML NNVGETFGVN GANAMQTNMQ RVDEMYRLVD STGTGEDRDW GNQTLLGRL LSQAQTVSIG KKVIESRRYS EAGRINRSMS GQTDIDMDKT KSSYQKMVIP VFDGAYGRDF RDYEAMRSEM L PALAEDSE ...文字列: MSIHFDFKNK QGKELLALNR QWNELSGYRA HAFNESVNML NNVGETFGVN GANAMQTNMQ RVDEMYRLVD STGTGEDRDW GNQTLLGRL LSQAQTVSIG KKVIESRRYS EAGRINRSMS GQTDIDMDKT KSSYQKMVIP VFDGAYGRDF RDYEAMRSEM L PALAEDSE EIEFTLLDDV NDYLWNGDAK LKVDTAVWGG LKADASVAAY SLGADLTTAT EAQVVAELLA LLDVLRITNK KS GPFELYI SPQIMSNWQK LAGANTNGFM NIMAAVRALI PEFSVVEADS ALQGNQVLCS VVGTRGLHAK IGMMMSSYQV PRV MHNDPY QFVKWFAAGF QSNNSFSGLK STVYGS UniProtKB: Putative coat protein |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 構成要素:
詳細: 50 mM Tris, pH 7.5, 100 mM NaCl, 8 mM MgSO4 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| グリッド | モデル: Ted Pella Inc, Lacey Carbon / メッシュ: 400 / 支持フィルム - トポロジー: CONTINUOUS / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 75 % / チャンバー内温度: 298 K / 装置: GATAN CRYOPLUNGE 3 |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 22500 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 22500 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 温度 | 最低: 80.0 K / 最高: 80.0 K |
| 撮影 | フィルム・検出器のモデル: DIRECT ELECTRON DE-16 (4k x 4k) 検出モード: COUNTING / 平均電子線量: 20.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 粒子像選択 | 選択した数: 16216 |
|---|---|
| 初期モデル | モデルのタイプ: NONE |
| 初期 角度割当 | タイプ: RANDOM ASSIGNMENT / ソフトウェア - 名称: jspr |
| 最終 角度割当 | タイプ: PROJECTION MATCHING / ソフトウェア - 名称: jspr |
| 最終 再構成 | 想定した対称性 - 点群: I (正20面体型対称 ) / 解像度のタイプ: BY AUTHOR / 解像度: 3.6 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: jspr / 使用した粒子像数: 8663 ) / 解像度のタイプ: BY AUTHOR / 解像度: 3.6 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: jspr / 使用した粒子像数: 8663 |
 ムービー
ムービー コントローラー
コントローラー






 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)