+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-7828 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the mitochondrial calcium uniporter from N. fischeri bound to saposin | ||||||||||||
 マップデータ マップデータ | Cryo-EM structure of MCU from N. fischeri low pass-filtered to 5.0 Angstrom resolution to show saposin molecules | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | Mitochondria / calcium channel / TRANSPORT PROTEIN | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報uniporter activity / uniplex complex / mitochondrial calcium ion homeostasis / calcium import into the mitochondrion / calcium channel activity / protein homotetramerization / mitochondrial inner membrane / metal ion binding 類似検索 - 分子機能 | ||||||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 5.0 Å | ||||||||||||
 データ登録者 データ登録者 | Nguyen NX / Armache J-P | ||||||||||||
| 資金援助 |  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2018 ジャーナル: Nature / 年: 2018タイトル: Cryo-EM structure of a fungal mitochondrial calcium uniporter. 著者: Nam X Nguyen / Jean-Paul Armache / Changkeun Lee / Yi Yang / Weizhong Zeng / Vamsi K Mootha / Yifan Cheng / Xiao-Chen Bai / Youxing Jiang /  要旨: The mitochondrial calcium uniporter (MCU) is a highly selective calcium channel localized to the inner mitochondrial membrane. Here, we describe the structure of an MCU orthologue from the fungus ...The mitochondrial calcium uniporter (MCU) is a highly selective calcium channel localized to the inner mitochondrial membrane. Here, we describe the structure of an MCU orthologue from the fungus Neosartorya fischeri (NfMCU) determined to 3.8 Å resolution by phase-plate cryo-electron microscopy. The channel is a homotetramer with two-fold symmetry in its amino-terminal domain (NTD) that adopts a similar structure to that of human MCU. The NTD assembles as a dimer of dimers to form a tetrameric ring that connects to the transmembrane domain through an elongated coiled-coil domain. The ion-conducting pore domain maintains four-fold symmetry, with the selectivity filter positioned at the start of the pore-forming TM2 helix. The aspartate and glutamate sidechains of the conserved DIME motif are oriented towards the central axis and separated by one helical turn. The structure of NfMCU offers insights into channel assembly, selective calcium permeation, and inhibitor binding. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_7828.map.gz emd_7828.map.gz | 36.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-7828-v30.xml emd-7828-v30.xml emd-7828.xml emd-7828.xml | 21.8 KB 21.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_7828.png emd_7828.png | 553.2 KB | ||
| Filedesc metadata |  emd-7828.cif.gz emd-7828.cif.gz | 6.8 KB | ||
| その他 |  emd_7828_additional.map.gz emd_7828_additional.map.gz emd_7828_additional_1.map.gz emd_7828_additional_1.map.gz | 37.2 MB 37.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-7828 http://ftp.pdbj.org/pub/emdb/structures/EMD-7828 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7828 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7828 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_7828.map.gz / 形式: CCP4 / 大きさ: 40.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_7828.map.gz / 形式: CCP4 / 大きさ: 40.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
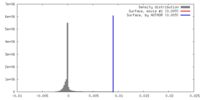
| 注釈 | Cryo-EM structure of MCU from N. fischeri low pass-filtered to 5.0 Angstrom resolution to show saposin molecules | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.84 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: Cryo-EM structure of MCU from N. fischeri at 3.8 Angstrom resolution
| ファイル | emd_7828_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
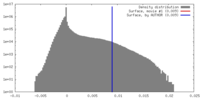
| 注釈 | Cryo-EM structure of MCU from N. fischeri at 3.8 Angstrom resolution | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
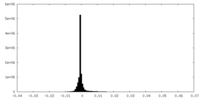
| 密度ヒストグラム |
-追加マップ: Cryo-EM structure of MCU from N. fischeri at 3.8 Angstrom resolution
| ファイル | emd_7828_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM structure of MCU from N. fischeri at 3.8 Angstrom resolution | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
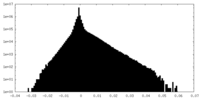
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Mitochondrial calcium uniporter / saposin complex
| 全体 | 名称: Mitochondrial calcium uniporter / saposin complex |
|---|---|
| 要素 |
|
-超分子 #1: Mitochondrial calcium uniporter / saposin complex
| 超分子 | 名称: Mitochondrial calcium uniporter / saposin complex / タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|
-超分子 #3: Mitochondrial calcium uniporter
| 超分子 | 名称: Mitochondrial calcium uniporter / タイプ: organelle_or_cellular_component / ID: 3 / 親要素: 1 / 含まれる分子: #2 |
|---|---|
| 由来(天然) | 生物種:  |
-超分子 #2: saposin
| 超分子 | 名称: saposin / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Saposin A
| 分子 | 名称: Saposin A / タイプ: protein_or_peptide / ID: 1 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 6.911511 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK) ...文字列: (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK) (UNK) |
-分子 #2: Mitochondrial calcium uniporter
| 分子 | 名称: Mitochondrial calcium uniporter / タイプ: protein_or_peptide / ID: 2 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 47.928004 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GSDKRGPQSA EPDPLERLEV KKVQQQHENE KDDSGRDTKS GGKVAKAMTK GDTIAGKLLT TPSRLFKLLI PLTTINRKDI EQIAILIHP QQPLSHLERL IQSEVPPIED ENGQKRPPFV SFIALQLEQD AIRPKRGMYE GTDAEIHRVE GGKDDATVAK R GEDFQEVD ...文字列: GSDKRGPQSA EPDPLERLEV KKVQQQHENE KDDSGRDTKS GGKVAKAMTK GDTIAGKLLT TPSRLFKLLI PLTTINRKDI EQIAILIHP QQPLSHLERL IQSEVPPIED ENGQKRPPFV SFIALQLEQD AIRPKRGMYE GTDAEIHRVE GGKDDATVAK R GEDFQEVD ETFSYLRRPG PGQGDKEQRF IRWSQSTEIG DFIRDAARAK EFIVTIEGAP AGLEQIHVAV PSFDERTYFL RM RLRKISR RIQGLAEIKH ECDALAHRGA QRVALGGFGI LAFWWYIVYK LTFETDLGWD TMEPVTYLVS LSTLMGGYLW FLY HNREIS YRSALDFTIN ARQKKLYQMK GIDLQVWESL IDEANAIRRE IKNIAAEYDV DWDERKDEQD DRVTEALKKE RRLK NGSQK EERPKDDRDD D UniProtKB: Calcium uniporter protein, mitochondrial |
-分子 #3: CALCIUM ION
| 分子 | 名称: CALCIUM ION / タイプ: ligand / ID: 3 / コピー数: 1 / 式: CA |
|---|---|
| 分子量 | 理論値: 40.078 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.6 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 詳細: 20 mM HEPES, pH 7.5, 300 mM sodium chloride, 1 mM calcium chloride, 2% glycerol |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
| 詳細 | N. fischeri MCU was reconstituted into lipid using E. coli total lipids and human saposin A as the membrane scaffolding protein. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | 位相板: VOLTA PHASE PLATE |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)