+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-6975 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | 3.3 angstrom structure of mouse TRPM7 with EDTA | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | CryoEM / Truncated mouse TRPM7 / MEMBRANE PROTEIN / TRANSFERASE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報intracellular magnesium ion homeostasis / calcium-dependent cell-matrix adhesion / varicosity / TRP channels / actomyosin structure organization / myosin binding / monoatomic cation transmembrane transport / necroptotic process / monoatomic cation channel activity / ruffle ...intracellular magnesium ion homeostasis / calcium-dependent cell-matrix adhesion / varicosity / TRP channels / actomyosin structure organization / myosin binding / monoatomic cation transmembrane transport / necroptotic process / monoatomic cation channel activity / ruffle / protein tetramerization / calcium channel activity / memory / synaptic vesicle membrane / calcium ion transport / kinase activity / actin binding / non-specific serine/threonine protein kinase / protein kinase activity / positive regulation of apoptotic process / phosphorylation / protein serine kinase activity / protein serine/threonine kinase activity / neuronal cell body / ATP binding / metal ion binding / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
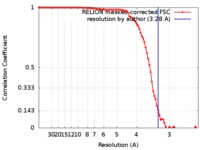
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.28 Å | |||||||||
 データ登録者 データ登録者 | Zhang J / Li Z | |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2018 ジャーナル: Proc Natl Acad Sci U S A / 年: 2018タイトル: Structure of the mammalian TRPM7, a magnesium channel required during embryonic development. 著者: Jingjing Duan / Zongli Li / Jian Li / Raymond E Hulse / Ana Santa-Cruz / William C Valinsky / Sunday A Abiria / Grigory Krapivinsky / Jin Zhang / David E Clapham /   要旨: The transient receptor potential ion channel subfamily M, member 7 (TRPM7), is a ubiquitously expressed protein that is required for mouse embryonic development. TRPM7 contains both an ion channel ...The transient receptor potential ion channel subfamily M, member 7 (TRPM7), is a ubiquitously expressed protein that is required for mouse embryonic development. TRPM7 contains both an ion channel and an α-kinase. The channel domain comprises a nonselective cation channel with notable permeability to Mg and Zn Here, we report the closed state structures of the mouse TRPM7 channel domain in three different ionic conditions to overall resolutions of 3.3, 3.7, and 4.1 Å. The structures reveal key residues for an ion binding site in the selectivity filter, with proposed partially hydrated Mg ions occupying the center of the conduction pore. In high [Mg], a prominent external disulfide bond is found in the pore helix, which is essential for ion channel function. Our results provide a structural framework for understanding the TRPM1/3/6/7 subfamily and extend the knowledge base upon which to study the diversity and evolution of TRP channels. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_6975.map.gz emd_6975.map.gz | 6.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-6975-v30.xml emd-6975-v30.xml emd-6975.xml emd-6975.xml | 11.1 KB 11.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_6975_fsc.xml emd_6975_fsc.xml | 9.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_6975.png emd_6975.png | 189.5 KB | ||
| Filedesc metadata |  emd-6975.cif.gz emd-6975.cif.gz | 6.3 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-6975 http://ftp.pdbj.org/pub/emdb/structures/EMD-6975 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6975 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6975 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_6975_validation.pdf.gz emd_6975_validation.pdf.gz | 423.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_6975_full_validation.pdf.gz emd_6975_full_validation.pdf.gz | 422.7 KB | 表示 | |
| XML形式データ |  emd_6975_validation.xml.gz emd_6975_validation.xml.gz | 10.9 KB | 表示 | |
| CIF形式データ |  emd_6975_validation.cif.gz emd_6975_validation.cif.gz | 14.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6975 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6975 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6975 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6975 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_6975.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_6975.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.23 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Membrane Protein, Transient Receptor Potential ion channel
| 全体 | 名称: Membrane Protein, Transient Receptor Potential ion channel |
|---|---|
| 要素 |
|
-超分子 #1: Membrane Protein, Transient Receptor Potential ion channel
| 超分子 | 名称: Membrane Protein, Transient Receptor Potential ion channel タイプ: cell / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Transient receptor potential cation channel subfamily M member 7
| 分子 | 名称: Transient receptor potential cation channel subfamily M member 7 タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO / EC番号: non-specific serine/threonine protein kinase |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 141.417719 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: SQKSWIESTL TKRECVYIIP SSKDPHRCLP GCQICQQLVR CFCGRLVKQH ACFTASLAMK YSDVKLGEHF NQAIEEWSVE KHTEQSPTD AYGVINFQGG SHSYRAKYVR LSYDTKPEII LQLLLKEWQM ELPKLVISVH GGMQKFELHP RIKQLLGKGL I KAAVTTGA ...文字列: SQKSWIESTL TKRECVYIIP SSKDPHRCLP GCQICQQLVR CFCGRLVKQH ACFTASLAMK YSDVKLGEHF NQAIEEWSVE KHTEQSPTD AYGVINFQGG SHSYRAKYVR LSYDTKPEII LQLLLKEWQM ELPKLVISVH GGMQKFELHP RIKQLLGKGL I KAAVTTGA WILTGGVNTG VAKHVGDALK EHASRSSRKI CTIGIAPWGV IENRNDLVGR DVVAPYQTLL NPLSKLNVLN NL HSHFILV DDGTVGKYGA EVRLRRELEK TINQQRIHAR IGQGVPVVAL IFEGGPNVIL TVLEYLQESP PVPVVVCEGT GRA ADLLAY IHKQTEEGGN LPDAAEPDII STIKKTFNFG QSEAVHLFQT MMECMKKKEL ITVFHIGSED HQDIDVAILT ALLK GTNAS AFDQLILTLA WDRVDIAKNH VFVYGQQWLV GSLEQAMLDA LVMDRVSFVK LLIENGVSMH KFLTIPRLEE LYNTK QGPT NPMLFHLIRD VKQGNLPPGY KITLIDIGLV IEYLMGGTYR CTYTRKRFRL IYNSLGGNNR RSGRNTSSST PQLRKS HET FGNRADKKEK MRHNHFIKTA QPYRPKMDAS MEEGKKKRTK DEIVDIDDPE TKRFPYPLNE LLIWACLMKR QVMARFL WQ HGEESMAKAL VACKIYRSMA YEAKQSDLVD DTSEELKQYS NDFGQLAVEL LEQSFRQDET MAMKLLTYEL KNWSNSTC L KLAVSSRLRP FVAHTCTQML LSDMWMGRLN MRKNSWYKVI LSILVPPAIL MLEYKTKAEM SHIPQSQDAH QMTMEDSEN NFHNITEEIP MEVFKEVKIL DSSDGKNEME IHIKSKKLPI TRKFYAFYHA PIVKFWFNTL AYLGFLMLYT FVVLVKMEQL PSVQEWIVI AYIFTYAIEK VREVFMSEAG KISQKIKVWF SDYFNVSDTI AIISFFVGFG LRFGAKWNYI NAYDNHVFVA G RLIYCLNI IFWYVRLLDF LAVNQQAGPY VMMIGKMVAN MFYIVVIMAL VLLSFGVPRK AILYPHEEPS WSLAKDIVFH PY WMIFGEV YAYEIDVCAN DSTLPTICGP GTWLTPFLQA VYLFVQYIIM VNLLIAFFNN VYLQVKAISN IVWKYQRYHF IMA YHEKPV LPPPLIILSH IVSLFCCVCK RRKKDKTSDG PKLFLTEEDQ KKLHDFEEQC VEMYFDEKDD KFNSGSEERI RVTF ERVEQ MSIQIKEVGD RVNYIKRSLQ SLDS UniProtKB: Transient receptor potential cation channel subfamily M member 7 |
-分子 #2: CHOLESTEROL HEMISUCCINATE
| 分子 | 名称: CHOLESTEROL HEMISUCCINATE / タイプ: ligand / ID: 2 / コピー数: 12 / 式: Y01 |
|---|---|
| 分子量 | 理論値: 486.726 Da |
| Chemical component information |  ChemComp-Y01: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE / 装置: FEI VITROBOT MARK I |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI POLARA 300 |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 BASE (4k x 4k) 平均電子線量: 56.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Tecnai Polara / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)