[English] 日本語
 Yorodumi
Yorodumi- EMDB-6664: The structure of rabbit skeletal muscle actomyosin rigor complex ... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: EMDB / ID: EMD-6664 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Title | The structure of rabbit skeletal muscle actomyosin rigor complex at 5.2 angstrom. | |||||||||
 Map data Map data | ||||||||||
 Sample Sample |
| |||||||||
 Keywords Keywords | Actin / Myosin / Muscle / rigor complex / MOTOR PROTEIN | |||||||||
| Function / homology |  Function and homology information Function and homology informationcontractile muscle fiber / myosin filament / myosin complex / myosin II complex / structural constituent of muscle / cytoskeletal motor activator activity / myosin heavy chain binding / microfilament motor activity / tropomyosin binding / actin filament bundle ...contractile muscle fiber / myosin filament / myosin complex / myosin II complex / structural constituent of muscle / cytoskeletal motor activator activity / myosin heavy chain binding / microfilament motor activity / tropomyosin binding / actin filament bundle / troponin I binding / filamentous actin / mesenchyme migration / myofibril / skeletal muscle myofibril / actin filament bundle assembly / striated muscle thin filament / skeletal muscle thin filament assembly / actin monomer binding / skeletal muscle fiber development / stress fiber / titin binding / actin filament polymerization / muscle contraction / cellular response to starvation / actin filament / filopodium / Hydrolases; Acting on acid anhydrides; Acting on acid anhydrides to facilitate cellular and subcellular movement / calcium-dependent protein binding / actin filament binding / lamellipodium / cell body / protein domain specific binding / hydrolase activity / calcium ion binding / positive regulation of gene expression / magnesium ion binding / ATP binding / identical protein binding / cytoplasm Similarity search - Function | |||||||||
| Biological species |  | |||||||||
| Method | helical reconstruction / cryo EM / Resolution: 5.2 Å | |||||||||
 Authors Authors | Fujii T / Namba K | |||||||||
 Citation Citation |  Journal: Nat Commun / Year: 2017 Journal: Nat Commun / Year: 2017Title: Structure of actomyosin rigour complex at 5.2 Å resolution and insights into the ATPase cycle mechanism. Authors: Takashi Fujii / Keiichi Namba /  Abstract: Muscle contraction is driven by cyclic association and dissociation of myosin head of the thick filament with thin actin filament coupled with ATP binding and hydrolysis by myosin. However, because ...Muscle contraction is driven by cyclic association and dissociation of myosin head of the thick filament with thin actin filament coupled with ATP binding and hydrolysis by myosin. However, because of the absence of actomyosin rigour structure at high resolution, it still remains unclear how the strong binding of myosin to actin filament triggers the release of hydrolysis products and how ATP binding causes their dissociation. Here we report the structure of mammalian skeletal muscle actomyosin rigour complex at 5.2 Å resolution by electron cryomicroscopy. Comparison with the structures of myosin in various states shows a distinctly large conformational change, providing insights into the ATPase-coupled reaction cycle of actomyosin. Based on our observations, we hypothesize that asymmetric binding along the actin filament could function as a Brownian ratchet by favouring directionally biased thermal motions of myosin and actin. | |||||||||
| History |
|
- Structure visualization
Structure visualization
| Movie |
 Movie viewer Movie viewer |
|---|---|
| Structure viewer | EM map:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| Supplemental images |
- Downloads & links
Downloads & links
-EMDB archive
| Map data |  emd_6664.map.gz emd_6664.map.gz | 55.7 MB |  EMDB map data format EMDB map data format | |
|---|---|---|---|---|
| Header (meta data) |  emd-6664-v30.xml emd-6664-v30.xml emd-6664.xml emd-6664.xml | 14.9 KB 14.9 KB | Display Display |  EMDB header EMDB header |
| Images |  emd_6664.png emd_6664.png | 71.5 KB | ||
| Filedesc metadata |  emd-6664.cif.gz emd-6664.cif.gz | 6.2 KB | ||
| Archive directory |  http://ftp.pdbj.org/pub/emdb/structures/EMD-6664 http://ftp.pdbj.org/pub/emdb/structures/EMD-6664 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6664 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6664 | HTTPS FTP |
-Related structure data
| Related structure data |  5h53MC M: atomic model generated by this map C: citing same article ( |
|---|---|
| Similar structure data |
- Links
Links
| EMDB pages |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| Related items in Molecule of the Month |
- Map
Map
| File |  Download / File: emd_6664.map.gz / Format: CCP4 / Size: 59.6 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) Download / File: emd_6664.map.gz / Format: CCP4 / Size: 59.6 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Projections & slices | Image control
Images are generated by Spider. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Voxel size | X=Y=Z: 1.35 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
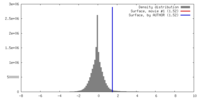
| Density |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Symmetry | Space group: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Details | EMDB XML:
CCP4 map header:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-Supplemental data
- Sample components
Sample components
-Entire : Actomyosin rigor complex
| Entire | Name: Actomyosin rigor complex |
|---|---|
| Components |
|
-Supramolecule #1: Actomyosin rigor complex
| Supramolecule | Name: Actomyosin rigor complex / type: complex / ID: 1 / Parent: 0 / Macromolecule list: #1-#4 |
|---|---|
| Source (natural) | Organism:  |
-Macromolecule #1: Skeletal muscle myosin heavy chain MyHC-EO/IIL
| Macromolecule | Name: Skeletal muscle myosin heavy chain MyHC-EO/IIL / type: protein_or_peptide / ID: 1 / Number of copies: 1 / Enantiomer: LEVO |
|---|---|
| Source (natural) | Organism:  |
| Molecular weight | Theoretical: 96.770133 KDa |
| Sequence | String: MSSDAEMAIF GEAAPYLRKP EKERIEAQNR PFDSKKACFA VDDKEMYVKG MIQSRENDKV TVKTLDDRTL TLNSDQVFPM NPPKFDKIE DMAMMTHLHE PAVLYNLKER YAAWMIYTYS GLFCVTVNPY KWLPVYNPEV VTAYRGKKRQ EAPPHIFSIS D NAYQFMLT ...String: MSSDAEMAIF GEAAPYLRKP EKERIEAQNR PFDSKKACFA VDDKEMYVKG MIQSRENDKV TVKTLDDRTL TLNSDQVFPM NPPKFDKIE DMAMMTHLHE PAVLYNLKER YAAWMIYTYS GLFCVTVNPY KWLPVYNPEV VTAYRGKKRQ EAPPHIFSIS D NAYQFMLT DRDNQSILIT GESGAGKTVN TKRVIQYFAT IAVTGDKKKE QQPGKMQGTL EDQIIQANPL LEAFGNAKTV RN DNSSRFG KFIRIHFGAT GKLASADIET YLLEKSRVTF QLSSERSYHI FYQIMSNKKP ELIDLLLIST NPFDFPFVSQ GEV TVASID DSEELLATDN AIDILGFSSE EKVGIYKLTG AVMHYGNMKF KQKQREEQAE PDGTEVADKA GYLMGLNSAE MLKG LCCPR VKVGNEYVTK GQNVQQVTNS VGALAKAVYE KMFLWMVTRI NQQLDTKQPR QYFIGVLDIA GFEIFDFNSL EQLCI NFTN EKLQQFFNHH MFVLEQEEYK KEGIEWEFID FGMDLAACIE LIEKPMGIFS ILEEECMFPK ATDTSFKNKL YDQHLG KSN NFQKPKPAKG KAEAHFSLVH YAGTVDYNIA GWLDKNKDPL NETVVGLYQK SALKLLSFLF SNYAGAEAGD SGGSKKG GK KKGSSFQTVS AVFRENLNKL MTNLRSTHPH FVRCLIPNET KTPGVMDHYL VMHQLRCNGV LEGIRICRKG FPSRILYA D FKQRYRILNA SAIPEGQFID SKNASEKLLN SIDVDREQYR FGHTKVFFKA GLLGLLEEMR DEKLVTLMTR TQALCRGYL MRVEFKKMME RRDSIFCIQY NIRAFMNVKH WPWMNLFFKI KPLLK UniProtKB: Myosin heavy chain 13 |
-Macromolecule #2: Myosin regulatory light chain 2, skeletal muscle isoform type 1
| Macromolecule | Name: Myosin regulatory light chain 2, skeletal muscle isoform type 1 type: protein_or_peptide / ID: 2 / Number of copies: 1 / Enantiomer: LEVO |
|---|---|
| Source (natural) | Organism:  |
| Molecular weight | Theoretical: 16.507588 KDa |
| Sequence | String: QTQIQEFKEA FTVIDQNRDG IIDKEDLRDT FAAMGRLNVK NEELDAMMKE ASGPINFTVF LTMFGEKLKG ADPEDVITGA FKVLDPEGK GTIKKQFLEE LLITQCDRFS QEEIKNMWAA FSPDVGGNVD YKNICYVITH GDAKDQE UniProtKB: Myosin regulatory light chain 2, skeletal muscle isoform type 1 |
-Macromolecule #3: Myosin light chain 1/3, skeletal muscle isoform
| Macromolecule | Name: Myosin light chain 1/3, skeletal muscle isoform / type: protein_or_peptide / ID: 3 / Number of copies: 1 / Enantiomer: LEVO |
|---|---|
| Source (natural) | Organism:  |
| Molecular weight | Theoretical: 17.187293 KDa |
| Sequence | String: IKIEFSKEQQ DEFKEAFLLY DRTGDSKITL SQVGDVLRAL GTNPTNAEVK KVLGNPSNEE MNAKKIEFEQ FLPMLQAISN NKDQGTYED FVEGLRVFDK EGNGTVMGAE LRHVLATLGE KMKEEEVEAL MAGQEDSNGC INYEAFVKHI MSID UniProtKB: Myosin light chain 1/3, skeletal muscle isoform |
-Macromolecule #4: Actin, alpha skeletal muscle
| Macromolecule | Name: Actin, alpha skeletal muscle / type: protein_or_peptide / ID: 4 / Number of copies: 2 / Enantiomer: LEVO |
|---|---|
| Source (natural) | Organism:  |
| Molecular weight | Theoretical: 41.875633 KDa |
| Sequence | String: DEDETTALVC DNGSGLVKAG FAGDDAPRAV FPSIVGRPRH QGVMVGMGQK DSYVGDEAQS KRGILTLKYP IE(HIC)GII TNW DDMEKIWHHT FYNELRVAPE EHPTLLTEAP LNPKANREKM TQIMFETFNV PAMYVAIQAV LSLYASGRTT GIVLDSG DG VTHNVPIYEG ...String: DEDETTALVC DNGSGLVKAG FAGDDAPRAV FPSIVGRPRH QGVMVGMGQK DSYVGDEAQS KRGILTLKYP IE(HIC)GII TNW DDMEKIWHHT FYNELRVAPE EHPTLLTEAP LNPKANREKM TQIMFETFNV PAMYVAIQAV LSLYASGRTT GIVLDSG DG VTHNVPIYEG YALPHAIMRL DLAGRDLTDY LMKILTERGY SFVTTAEREI VRDIKEKLCY VALDFENEMA TAASSSSL E KSYELPDGQV ITIGNERFRC PETLFQPSFI GMESAGIHET TYNSIMKCDI DIRKDLYANN VMSGGTTMYP GIADRMQKE ITALAPSTMK IKIIAPPERK YSVWIGGSIL ASLSTFQQMW ITKQEYDEAG PSIVHRKCF UniProtKB: Actin, alpha skeletal muscle |
-Macromolecule #5: ADENOSINE-5'-DIPHOSPHATE
| Macromolecule | Name: ADENOSINE-5'-DIPHOSPHATE / type: ligand / ID: 5 / Number of copies: 2 / Formula: ADP |
|---|---|
| Molecular weight | Theoretical: 427.201 Da |
| Chemical component information |  ChemComp-ADP: |
-Experimental details
-Structure determination
| Method | cryo EM |
|---|---|
 Processing Processing | helical reconstruction |
| Aggregation state | filament |
- Sample preparation
Sample preparation
| Buffer | pH: 7.5 |
|---|---|
| Vitrification | Cryogen name: ETHANE |
- Electron microscopy
Electron microscopy
| Microscope | JEOL 3200FSC |
|---|---|
| Image recording | Film or detector model: TVIPS TEMCAM-F415 (4k x 4k) / Average electron dose: 20.0 e/Å2 |
| Electron beam | Acceleration voltage: 200 kV / Electron source:  FIELD EMISSION GUN FIELD EMISSION GUN |
| Electron optics | Illumination mode: FLOOD BEAM / Imaging mode: BRIGHT FIELD |
- Image processing
Image processing
| Final reconstruction | Applied symmetry - Helical parameters - Δz: 27.6 Å Applied symmetry - Helical parameters - Δ&Phi: 166.7 ° Applied symmetry - Helical parameters - Axial symmetry: C1 (asymmetric) Resolution.type: BY AUTHOR / Resolution: 5.2 Å / Resolution method: FSC 0.143 CUT-OFF / Number images used: 31535 |
|---|---|
| Startup model | Type of model: NONE |
| Final angle assignment | Type: NOT APPLICABLE |
 Movie
Movie Controller
Controller
















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)