+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-4571 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Elongator catalytic subcomplex Elp123 lobe | |||||||||
 マップデータ マップデータ | Postprocessed map of Elongator catalytic subcomplex Elp123 lobe from yeast at 3.3 A resolution. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Elongator / yeast / tRNA modification / Elp123 / TRANSLATION | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報tRNA carboxymethyluridine synthase / tRNA uridine(34) acetyltransferase activity / elongator holoenzyme complex / tRNA wobble base 5-methoxycarbonylmethyl-2-thiouridinylation / protein urmylation / tRNA wobble uridine modification / : / protein transport / regulation of translation / 4 iron, 4 sulfur cluster binding ...tRNA carboxymethyluridine synthase / tRNA uridine(34) acetyltransferase activity / elongator holoenzyme complex / tRNA wobble base 5-methoxycarbonylmethyl-2-thiouridinylation / protein urmylation / tRNA wobble uridine modification / : / protein transport / regulation of translation / 4 iron, 4 sulfur cluster binding / microtubule binding / tRNA binding / regulation of transcription by RNA polymerase II / nucleoplasm / identical protein binding / nucleus / metal ion binding / cytoplasm / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |   | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.3 Å | |||||||||
 データ登録者 データ登録者 | Dauden MI / Weis F | |||||||||
| 資金援助 |  ドイツ, ドイツ,  ポーランド, 2件 ポーランド, 2件
| |||||||||
 引用 引用 |  ジャーナル: Sci Adv / 年: 2019 ジャーナル: Sci Adv / 年: 2019タイトル: Molecular basis of tRNA recognition by the Elongator complex. 著者: Maria I Dauden / Marcin Jaciuk / Felix Weis / Ting-Yu Lin / Carolin Kleindienst / Nour El Hana Abbassi / Heena Khatter / Rościsław Krutyhołowa / Karin D Breunig / Jan Kosinski / Christoph ...著者: Maria I Dauden / Marcin Jaciuk / Felix Weis / Ting-Yu Lin / Carolin Kleindienst / Nour El Hana Abbassi / Heena Khatter / Rościsław Krutyhołowa / Karin D Breunig / Jan Kosinski / Christoph W Müller / Sebastian Glatt /   要旨: The highly conserved Elongator complex modifies transfer RNAs (tRNAs) in their wobble base position, thereby regulating protein synthesis and ensuring proteome stability. The precise mechanisms of ...The highly conserved Elongator complex modifies transfer RNAs (tRNAs) in their wobble base position, thereby regulating protein synthesis and ensuring proteome stability. The precise mechanisms of tRNA recognition and its modification reaction remain elusive. Here, we show cryo-electron microscopy structures of the catalytic subcomplex of Elongator and its tRNA-bound state at resolutions of 3.3 and 4.4 Å. The structures resolve details of the catalytic site, including the substrate tRNA, the iron-sulfur cluster, and a SAM molecule, which are all validated by mutational analyses in vitro and in vivo. tRNA binding induces conformational rearrangements, which precisely position the targeted anticodon base in the active site. Our results provide the molecular basis for substrate recognition of Elongator, essential to understand its cellular function and role in neurodegenerative diseases and cancer. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_4571.map.gz emd_4571.map.gz | 1.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-4571-v30.xml emd-4571-v30.xml emd-4571.xml emd-4571.xml | 29.6 KB 29.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_4571_fsc.xml emd_4571_fsc.xml | 13 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_4571.png emd_4571.png | 187.7 KB | ||
| Filedesc metadata |  emd-4571.cif.gz emd-4571.cif.gz | 8.8 KB | ||
| その他 |  emd_4571_additional_1.map.gz emd_4571_additional_1.map.gz emd_4571_additional_2.map.gz emd_4571_additional_2.map.gz emd_4571_half_map_1.map.gz emd_4571_half_map_1.map.gz emd_4571_half_map_2.map.gz emd_4571_half_map_2.map.gz | 2.6 MB 1.6 MB 85.7 MB 85.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-4571 http://ftp.pdbj.org/pub/emdb/structures/EMD-4571 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4571 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4571 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_4571_validation.pdf.gz emd_4571_validation.pdf.gz | 385.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_4571_full_validation.pdf.gz emd_4571_full_validation.pdf.gz | 385 KB | 表示 | |
| XML形式データ |  emd_4571_validation.xml.gz emd_4571_validation.xml.gz | 16.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4571 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4571 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4571 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4571 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_4571.map.gz / 形式: CCP4 / 大きさ: 93 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_4571.map.gz / 形式: CCP4 / 大きさ: 93 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Postprocessed map of Elongator catalytic subcomplex Elp123 lobe from yeast at 3.3 A resolution. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.35 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: LocScale map of Elongator catalytic subcomplex Elp123 lobe...
| ファイル | emd_4571_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | LocScale map of Elongator catalytic subcomplex Elp123 lobe including the dimerization domain (DD) of Elp1, used to build the atomic model of the C-terminal domains of Elp1. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
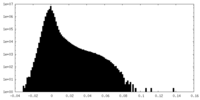
| 密度ヒストグラム |
-追加マップ: LocScale map of Elongator catalytic subcomplex Elp123 lobe...
| ファイル | emd_4571_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | LocScale map of Elongator catalytic subcomplex Elp123 lobe used to build the atomic model. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
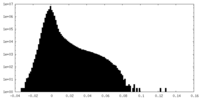
| 密度ヒストグラム |
-ハーフマップ: Half map of Elongator catalytic subcomplex Elp123 lobe from yeast.
| ファイル | emd_4571_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map of Elongator catalytic subcomplex Elp123 lobe from yeast. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map of Elongator catalytic subcomplex Elp123 lobe from yeast.
| ファイル | emd_4571_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map of Elongator catalytic subcomplex Elp123 lobe from yeast. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Elongator catalytic subcomplex Elp123
| 全体 | 名称: Elongator catalytic subcomplex Elp123 |
|---|---|
| 要素 |
|
-超分子 #1: Elongator catalytic subcomplex Elp123
| 超分子 | 名称: Elongator catalytic subcomplex Elp123 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 詳細: The EM map corresponds to one lobe of the Elp123 complex, that includes one copy of Elp1, Elp2 and Elp3, and the C-terminal part of a second copy of Elp1. |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 621 KDa |
-分子 #1: Elongator complex protein 1
| 分子 | 名称: Elongator complex protein 1 / タイプ: protein_or_peptide / ID: 1 詳細: Chain D corresponds to the C-terminal domain of Elp1 コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  株: ATCC 204508 / S288c |
| 分子量 | 理論値: 153.166266 KDa |
| 配列 | 文字列: MVEHDKSGSK RQELRSNMRN LITLNKGKFK PTASTAEGDE DDLSFTLLDS VFDTLSDSIT CVLGSTDIGA IEVQQFMKDG SRNVLASFN IQTFDDKLLS FVHFADINQL VFVFEQGDII TATYDPVSLD PAETLIEIMG TIDNGIAAAQ WSYDEETLAM V TKDRNVVV ...文字列: MVEHDKSGSK RQELRSNMRN LITLNKGKFK PTASTAEGDE DDLSFTLLDS VFDTLSDSIT CVLGSTDIGA IEVQQFMKDG SRNVLASFN IQTFDDKLLS FVHFADINQL VFVFEQGDII TATYDPVSLD PAETLIEIMG TIDNGIAAAQ WSYDEETLAM V TKDRNVVV LSKLFEPISE YHLEVDDLKI SKHVTVGWGK KETQFRGKGA RAMEREALAS LKASGLVGNQ LRDPTMPYMV DT GDVTALD SHEITISWRG DCDYFAVSSV EEVPDEDDET KSIKRRAFRV FSREGQLDSA SEPVTGMEHQ LSWKPQGSLI ASI QRKTDL GEEDSVDVIF FERNGLRHGE FDTRLPLDEK VESVCWNSNS EALAVVLANR IQLWTSKNYH WYLKQELYAS DISY VKWHP EKDFTLMFSD AGFINIVDFA YKMAQGPTLE PFDNGTSLVV DGRTVNITPL ALANVPPPMY YRDFETPGNV LDVAC SFSN EIYAAINKDV LIFAAVPSIE EMKKGKHPSI VCEFPKSEFT SEVDSLRQVA FINDSIVGVL LDTDNLSRIA LLDIQD ITQ PTLITIVEVY DKIVLLRSDF DYNHLVYETR DGTVCQLDAE GQLMEITKFP QLVRDFRVKR VHNTSAEDDD NWSAESS EL VAFGITNNGK LFANQVLLAS AVTSLEITDS FLLFTTAQHN LQFVHLNSTD FKPLPLVEEG VEDERVRAIE RGSILVSV I PSKSSVVLQA TRGNLETIYP RIMVLAEVRK NIMAKRYKEA FIVCRTHRIN LDILHDYAPE LFIENLEVFI NQIGRVDYL NLFISCLSED DVTKTKYKET LYSGISKSFG MEPAPLTEMQ IYMKKKMFDP KTSKVNKICD AVLNVLLSNP EYKKKYLQTI ITAYASQNP QNLSAALKLI SELENSEEKD SCVTYLCFLQ DVNVVYKSAL SLYDVSLALL VAQKSQMDPR EYLPFLQELQ D NEPLRRKF LIDDYLGNYE KALEHLSEID KDGNVSEEVI DYVESHDLYK HGLALYRYDS EKQNVIYNIY AKHLSSNQMY TD AAVAYEM LGKLKEAMGA YQSAKRWREA MSIAVQKFPE EVESVAEELI SSLTFEHRYV DAADIQLEYL DNVKEAVALY CKA YRYDIA SLVAIKAKKD ELLEEVVDPG LGEGFGIIAE LLADCKGQIN SQLRRLRELR AKKEENPYAF YGQETEQADD VSVA PSETS TQESFFTRYT GKTGGTAKTG ASRRTAKNKR REERKRARGK KGTIYEEEYL VQSVGRLIER LNQTKPDAVR VVEGL CRRN MREQAHQIQK NFVEVLDLLK ANVKEIYSIS EKDRERVNEN GEVYYIPEIP VPEIHDFPKS HIVDF UniProtKB: Elongator complex protein 1 |
-分子 #2: Elongator complex protein 2
| 分子 | 名称: Elongator complex protein 2 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  株: ATCC 204508 / S288c |
| 分子量 | 理論値: 89.51943 KDa |
| 配列 | 文字列: MVECITPEAI FIGANKQTQV SDIHKVKKIV AFGAGKTIAL WDPIEPNNKG VYATLKGHEA EVTCVRFVPD SDFMVSASED HHVKIWKFT DYSHLQCIQT IQHYSKTIVA LSALPSLISV GCADGTISIW RQNIQNDEFG LAHEFTIKKG FFYPLCLSLS K VEEKKYLL ...文字列: MVECITPEAI FIGANKQTQV SDIHKVKKIV AFGAGKTIAL WDPIEPNNKG VYATLKGHEA EVTCVRFVPD SDFMVSASED HHVKIWKFT DYSHLQCIQT IQHYSKTIVA LSALPSLISV GCADGTISIW RQNIQNDEFG LAHEFTIKKG FFYPLCLSLS K VEEKKYLL AIGGTNVNVF IASFILSDSG IEKCRVVAEL EGHEDWVKSL AFRHQETPGD YLLCSGSQDR YIRLWRIRIN DL IDDSEED SKKLTLLSNK QYKFQIDDEL RVGINFEALI MGHDDWISSL QWHESRLQLL AATADTSLMV WEPDETSGIW VCS LRLGEM SSKGASTATG SSGGFWSCLW FTHERMDFFL TNGKTGSWRM WATKDNIICD QRLGISGATK DVTDIAWSPS GEYL LATSL DQTTRLFAPW IYDASGRKRE IATWHEFSRP QIHGYDMICV ETVTDTRFVS GGDEKILRSF DLPKGVAGML QKFVG IQFE EKSEMPDSAT VPVLGLSNKA GEDDANEDDE EEEGGNKETP DITDPLSLLE CPPMEDQLQR HLLWPEVEKL YGHGFE ITC LDISPDQKLI ASACRSNNVQ NAVIRIFSTE NWLEIKPALP FHSLTITRLK FSKDGKFLLS VCRDRKWALW ERNMEDN TF ELRFKNEKPH TRIIWDADWA PLEFGNVFVT ASRDKTVKVW RHQKEPADDY VLEASIKHTK AVTAISIHDS MIREKILI S VGLENGEIYL YSYTLGKFEL ITQLNEDITP ADKITRLRWS HLKRNGKLFL GVGSSDLSTR IYSLAYE UniProtKB: Elongator complex protein 2 |
-分子 #3: Elongator complex protein 3
| 分子 | 名称: Elongator complex protein 3 / タイプ: protein_or_peptide / ID: 3 / 詳細: FeS cluster and 5DA / コピー数: 1 / 光学異性体: LEVO / EC番号: histone acetyltransferase |
|---|---|
| 由来(天然) | 生物種:  株: ATCC 204508 / S288c |
| 分子量 | 理論値: 63.755059 KDa |
| 配列 | 文字列: MARHGKGPKT NKKKLAPEKE RFIQCCADIT LELTDSLTSG TTREINLNGL ITKYSKKYKL KQQPRLTDII NSIPDQYKKY LLPKLKAKP VRTASGIAVV AVMCKPHRCP HIAYTGNICV YCPGGPDSDF EYSTQSYTGY EPTSMRAIRA RYDPYEQARG R VEQLKQLG ...文字列: MARHGKGPKT NKKKLAPEKE RFIQCCADIT LELTDSLTSG TTREINLNGL ITKYSKKYKL KQQPRLTDII NSIPDQYKKY LLPKLKAKP VRTASGIAVV AVMCKPHRCP HIAYTGNICV YCPGGPDSDF EYSTQSYTGY EPTSMRAIRA RYDPYEQARG R VEQLKQLG HSIDKVEYVL MGGTFMSLPK EYREDFIVKL HNALSGFNGN DIDEAILYSQ QSLTKCVGIT IETRPDYCTQ TH LDDMLKY GCTRLEIGVQ SLYEDVARDT NRGHTVRSVC ETFAVSKDAG YKVVSHMMPD LPNVGMERDI EQFKEYFENP DFR TDGLKI YPTLVIRGTG LYELWKTGRY KSYSANALVD LVARILALVP PWTRIYRVQR DIPMPLVTSG VDNGNLRELA LARM KDLGT TCRDVRTREV GIQEVHHKVQ PDQVELIRRD YYANGGWETF LSYEDPKKDI LIGLLRLRKA SKKYTYRKEF TSQRT SIVR ELHVYGSVVP LHSRDPRKFQ HQGFGTLLME EAERIAKEEH GSEKISVISG VGVRNYYGKL GYELDGPYMS KRI UniProtKB: Elongator complex protein 3 |
-分子 #4: IRON/SULFUR CLUSTER
| 分子 | 名称: IRON/SULFUR CLUSTER / タイプ: ligand / ID: 4 / コピー数: 1 / 式: SF4 |
|---|---|
| 分子量 | 理論値: 351.64 Da |
| Chemical component information |  ChemComp-FS1: |
-分子 #5: 5'-DEOXYADENOSINE
| 分子 | 名称: 5'-DEOXYADENOSINE / タイプ: ligand / ID: 5 / コピー数: 1 / 式: 5AD |
|---|---|
| 分子量 | 理論値: 251.242 Da |
| Chemical component information |  ChemComp-5AD: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.4 mg/mL | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||||||||
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 200 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. / 詳細: Pelco EasyGlow glow discharger, 20 mA | ||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV 詳細: 2.5 ul of sample, blotting parameters: wait time 15 s, blot force 5, blot time 5-8 s.. | ||||||||||||||||||
| 詳細 | The sample was cross-linked with 0.01% glutaraldehyde, quenched and then plunged. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 詳細 | Gatan Quantum energy filter and a K2 Summit direct detector |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 実像数: 4614 / 平均電子線量: 43.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 105000 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル |
| ||||||
|---|---|---|---|---|---|---|---|
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT | ||||||
| 得られたモデル |  PDB-6qk7: |
 ムービー
ムービー コントローラー
コントローラー















 Z
Z Y
Y X
X