+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | TRRAP module of the human TIP60 complex | |||||||||||||||
 マップデータ マップデータ | ||||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード | Chromatin Modification / Gene Regulation | |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報transcription factor TFTC complex / Swr1 complex / protein antigen binding / regulation of double-strand break repair / SAGA complex / Formation of Senescence-Associated Heterochromatin Foci (SAHF) / DNA repair-dependent chromatin remodeling / NuA4 histone acetyltransferase complex / regulation of RNA splicing / positive regulation of double-strand break repair via homologous recombination ...transcription factor TFTC complex / Swr1 complex / protein antigen binding / regulation of double-strand break repair / SAGA complex / Formation of Senescence-Associated Heterochromatin Foci (SAHF) / DNA repair-dependent chromatin remodeling / NuA4 histone acetyltransferase complex / regulation of RNA splicing / positive regulation of double-strand break repair via homologous recombination / regulation of DNA repair / transcription coregulator activity / Formation of the beta-catenin:TCF transactivating complex / helicase activity / DNA Damage/Telomere Stress Induced Senescence / 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 / nucleosome / HATs acetylate histones / chromatin organization / regulation of apoptotic process / hydrolase activity / Ub-specific processing proteases / regulation of cell cycle / nuclear speck / DNA repair / chromatin binding / regulation of transcription by RNA polymerase II / regulation of DNA-templated transcription / positive regulation of DNA-templated transcription / Golgi apparatus / DNA binding / nucleoplasm / ATP binding / nucleus 類似検索 - 分子機能 | |||||||||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.4 Å | |||||||||||||||
 データ登録者 データ登録者 | Yang Z / Mameri A / Florez Ariza AJ / Cote J / Nogales E | |||||||||||||||
| 資金援助 |  米国, 米国,  カナダ, 4件 カナダ, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Science / 年: 2024 ジャーナル: Science / 年: 2024タイトル: Structural insights into the human NuA4/TIP60 acetyltransferase and chromatin remodeling complex. 著者: Zhenlin Yang / Amel Mameri / Claudia Cattoglio / Catherine Lachance / Alfredo Jose Florez Ariza / Jie Luo / Jonathan Humbert / Deepthi Sudarshan / Arul Banerjea / Maxime Galloy / Amélie ...著者: Zhenlin Yang / Amel Mameri / Claudia Cattoglio / Catherine Lachance / Alfredo Jose Florez Ariza / Jie Luo / Jonathan Humbert / Deepthi Sudarshan / Arul Banerjea / Maxime Galloy / Amélie Fradet-Turcotte / Jean-Philippe Lambert / Jeff A Ranish / Jacques Côté / Eva Nogales /   要旨: The human nucleosome acetyltransferase of histone H4 (NuA4)/Tat-interactive protein, 60 kilodalton (TIP60) coactivator complex, a fusion of the yeast switch/sucrose nonfermentable related 1 (SWR1) ...The human nucleosome acetyltransferase of histone H4 (NuA4)/Tat-interactive protein, 60 kilodalton (TIP60) coactivator complex, a fusion of the yeast switch/sucrose nonfermentable related 1 (SWR1) and NuA4 complexes, both incorporates the histone variant H2A.Z into nucleosomes and acetylates histones H4, H2A, and H2A.Z to regulate gene expression and maintain genome stability. Our cryo-electron microscopy studies show that, within the NuA4/TIP60 complex, the E1A binding protein P400 (EP400) subunit serves as a scaffold holding the different functional modules in specific positions, creating a distinct arrangement of the actin-related protein (ARP) module. EP400 interacts with the transformation/transcription domain-associated protein (TRRAP) subunit by using a footprint that overlaps with that of the Spt-Ada-Gcn5 acetyltransferase (SAGA) complex, preventing the formation of a hybrid complex. Loss of the TRRAP subunit leads to mislocalization of NuA4/TIP60, resulting in the redistribution of H2A.Z and its acetylation across the genome, emphasizing the dual functionality of NuA4/TIP60 as a single macromolecular assembly. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_45176.map.gz emd_45176.map.gz | 168.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-45176-v30.xml emd-45176-v30.xml emd-45176.xml emd-45176.xml | 22 KB 22 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_45176.png emd_45176.png | 115.7 KB | ||
| Filedesc metadata |  emd-45176.cif.gz emd-45176.cif.gz | 9.9 KB | ||
| その他 |  emd_45176_half_map_1.map.gz emd_45176_half_map_1.map.gz emd_45176_half_map_2.map.gz emd_45176_half_map_2.map.gz | 165.3 MB 165.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-45176 http://ftp.pdbj.org/pub/emdb/structures/EMD-45176 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-45176 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-45176 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_45176_validation.pdf.gz emd_45176_validation.pdf.gz | 1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_45176_full_validation.pdf.gz emd_45176_full_validation.pdf.gz | 1 MB | 表示 | |
| XML形式データ |  emd_45176_validation.xml.gz emd_45176_validation.xml.gz | 14.9 KB | 表示 | |
| CIF形式データ |  emd_45176_validation.cif.gz emd_45176_validation.cif.gz | 17.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-45176 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-45176 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-45176 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-45176 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_45176.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_45176.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.05 Å | ||||||||||||||||||||||||||||||||||||
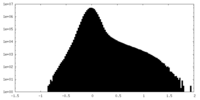
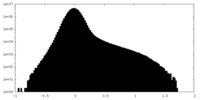
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_45176_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
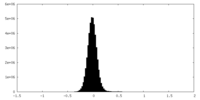
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_45176_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
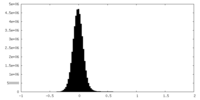
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : TRRAP module of human TIP60 complex
| 全体 | 名称: TRRAP module of human TIP60 complex |
|---|---|
| 要素 |
|
-超分子 #1: TRRAP module of human TIP60 complex
| 超分子 | 名称: TRRAP module of human TIP60 complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #2, #1 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Transformation/transcription domain-associated protein
| 分子 | 名称: Transformation/transcription domain-associated protein タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 438.139531 KDa |
| 配列 | 文字列: MAFVATQGAT VVDQTTLMKK YLQFVAALTD VNTPDETKLK MMQEVSENFE NVTSSPQYST FLEHIIPRFL TFLQDGEVQF LQEKPAQQL RKLVLEIIHR IPTNEHLRPH TKNVLSVMFR FLETENEENV LICLRIIIEL HKQFRPPITQ EIHHFLDFVK Q IYKELPKV ...文字列: MAFVATQGAT VVDQTTLMKK YLQFVAALTD VNTPDETKLK MMQEVSENFE NVTSSPQYST FLEHIIPRFL TFLQDGEVQF LQEKPAQQL RKLVLEIIHR IPTNEHLRPH TKNVLSVMFR FLETENEENV LICLRIIIEL HKQFRPPITQ EIHHFLDFVK Q IYKELPKV VNRYFENPQV IPENTVPPPE MVGMITTIAV KVNPEREDSE TRTHSIIPRG SLSLKVLAEL PIIVVLMYQL YK LNIHNVV AEFVPLIMNT IAIQVSAQAR QHKLYNKELY ADFIAAQIKT LSFLAYIIRI YQELVTKYSQ QMVKGMLQLL SNC PAETAH LRKELLIAAK HILTTELRNQ FIPCMDKLFD ESILIGSGYT ARETLRPLAY STLADLVHHV RQHLPLSDLS LAVQ LFAKN IDDESLPSSI QTMSCKLLLN LVDCIRSKSE QESGNGRDVL MRMLEVFVLK FHTIARYQLS AIFKKCKPQS ELGAV EAAL PGVPTAPAAP GPAPSPAPVP APPPPPPPPP PATPVTPAPV PPFEKQGEKD KEDKQTFQVT DCRSLVKTLV CGVKTI TWG ITSCKAPGEA QFIPNKQLQP KETQIYIKLV KYAMQALDIY QVQIAGNGQT YIRVANCQTV RMKEEKEVLE HFAGVFT MM NPLTFKEIFQ TTVPYMVERI SKNYALQIVA NSFLANPTTS ALFATILVEY LLDRLPEMGS NVELSNLYLK LFKLVFGS V SLFAAENEQM LKPHLHKIVN SSMELAQTAK EPYNYFLLLR ALFRSIGGGS HDLLYQEFLP LLPNLLQGLN MLQSGLHKQ HMKDLFVELC LTVPVRLSSL LPYLPMLMDP LVSALNGSQT LVSQGLRTLE LCVDNLQPDF LYDHIQPVRA ELMQALWRTL RNPADSISH VAYRVLGKFG GSNRKMLKES QKLHYVVTEV QGPSITVEFS DCKASLQLPM EKAIETALDC LKSANTEPYY R RQAWEVIK CFLVAMMSLE DNKHALYQLL AHPNFTEKTI PNVIISHRYK AQDTPARKTF EQALTGAFMS AVIKDLRPSA LP FVASLIR HYTMVAVAQQ CGPFLLPCYQ VGSQPSTAMF HSEENGSKGM DPLVLIDAIA ICMAYEEKEL CKIGEVALAV IFD VASIIL GSKERACQLP LFSYIVERLC ACCYEQAWYA KLGGVVSIKF LMERLPLTWV LQNQQTFLKA LLFVMMDLTG EVSN GAVAM AKTTLEQLLM RCATPLKDEE RAEEIVAAQE KSFHHVTHDL VREVTSPNST VRKQAMHSLQ VLAQVTGKSV TVIME PHKE VLQDMVPPKK HLLRHQPANA QIGLMEGNTF CTTLQPRLFT MDLNVVEHKV FYTELLNLCE AEDSALTKLP CYKSLP SLV PLRIAALNAL AACNYLPQSR EKIIAALFKA LNSTNSELQE AGEACMRKFL EGATIEVDQI HTHMRPLLMM LGDYRSL TL NVVNRLTSVT RLFPNSFNDK FCDQMMQHLR KWMEVVVITH KGGQRSDGNE SISECGRCPL SPFCQFEEMK ICSAIINL F HLIPAAPQTL VKPLLEVVMK TERAMLIEAG SPFREPLIKF LTRHPSQTVE LFMMEATLND PQWSRMFMSF LKHKDARPL RDVLAANPNR FITLLLPGGA QTAVRPGSPS TSTMRLDLQF QAIKIISIIV KNDDSWLASQ HSLVSQLRRV WVSENFQERH RKENMAATN WKEPKLLAYC LLNYCKRNYG DIELLFQLLR AFTGRFLCNM TFLKEYMEEE IPKNYSIAQK RALFFRFVDF N DPNFGDEL KAKVLQHILN PAFLYSFEKG EGEQLLGPPN PEGDNPESIT SVFITKVLDP EKQADMLDSL RIYLLQYATL LV EHAPHHI HDNNKNRNSK LRRLMTFAWP CLLSKACVDP ACKYSGHLLL AHIIAKFAIH KKIVLQVFHS LLKAHAMEAR AIV RQAMAI LTPAVPARME DGHQMLTHWT RKIIVEEGHT VPQLVHILHL IVQHFKVYYP VRHHLVQHMV SAMQRLGFTP SVTI EQRRL AVDLSEVVIK WELQRIKDQQ PDSDMDPNSS GEGVNSVSSS IKRGLSVDSA QEVKRFRTAT GAISAVFGRS QSLPG ADSL LAKPIDKQHT DTVVNFLIRV ACQVNDNTNT AGSPGEVLSR RCVNLLKTAL RPDMWPKSEL KLQWFDKLLM TVEQPN QVN YGNICTGLEV LSFLLTVLQS PAILSSFKPL QRGIAACMTC GNTKVLRAVH SLLSRLMSIF PTEPSTSSVA SKYEELE CL YAAVGKVIYE GLTNYEKATN ANPSQLFGTL MILKSACSNN PSYIDRLISV FMRSLQKMVR EHLNPQAASG STEATSGT S ELVMLSLELV KTRLAVMSME MRKNFIQAIL TSLIEKSPDA KILRAVVKIV EEWVKNNSPM AANQTPTLRE KSILLVKMM TYIEKRFPED LELNAQFLDL VNYVYRDETL SGSELTAKLE PAFLSGLRCA QPLIRAKFFE VFDNSMKRRV YERLLYVTCS QNWEAMGNH FWIKQCIELL LAVCEKSTPI GTSCQGAMLP SITNVINLAD SHDRAAFAMV THVKQEPRER ENSESKEEDV E IDIELAPG DQTSTPKTKE LSEKDIGNQL HMLTNRHDKF LDTLREVKTG ALLSAFVQLC HISTTLAEKT WVQLFPRLWK IL SDRQQHA LAGEISPFLC SGSHQVQRDC QPSALNCFVE AMSQCVPPIP IRPCVLKYLG KTHNLWFRST LMLEHQAFEK GLS LQIKPK QTTEFYEQES ITPPQQEILD SLAELYSLLQ EEDMWAGLWQ KRCKYSETAT AIAYEQHGFF EQAQESYEKA MDKA KKEHE RSNASPAIFP EYQLWEDHWI RCSKELNQWE ALTEYGQSKG HINPYLVLEC AWRVSNWTAM KEALVQVEVS CPKEM AWKV NMYRGYLAIC HPEEQQLSFI ERLVEMASSL AIREWRRLPH VVSHVHTPLL QAAQQIIELQ EAAQINAGLQ PTNLGR NNS LHDMKTVVKT WRNRLPIVSD DLSHWSSIFM WRQHHYQGKP TWSGMHSSSI VTAYENSSQH DPSSNNAMLG VHASASA II QYGKIARKQG LVNVALDILS RIHTIPTVPI VDCFQKIRQQ VKCYLQLAGV MGKNECMQGL EVIESTNLKY FTKEMTAE F YALKGMFLAQ INKSEEANKA FSAAVQMHDV LVKAWAMWGD YLENIFVKER QLHLGVSAIT CYLHACRHQN ESKSRKYLA KVLWLLSFDD DKNTLADAVD KYCIGVPPIQ WLAWIPQLLT CLVGSEGKLL LNLISQVGRV YPQAVYFPIR TLYLTLKIEQ RERYKSDPG PIRATAPMWR CSRIMHMQRE LHPTLLSSLE GIVDQMVWFR ENWHEEVLRQ LQQGLAKCYS VAFEKSGAVS D AKITPHTL NFVKKLVSTF GVGLENVSNV STMFSSAASE SLARRAQATA QDPVFQKLKG QFTTDFDFSV PGSMKLHNLI SK LKKWIKI LEAKTKQLPK FFLIEEKCRF LSNFSAQTAE VEIPGEFLMP KPTHYYIKIA RFMPRVEIVQ KHNTAARRLY IRG HNGKIY PYLVMNDACL TESRREERVL QLLRLLNPCL EKRKETTKRH LFFTVPRVVA VSPQMRLVED NPSSLSLVEI YKQR CAKKG IEHDNPISRY YDRLATVQAR GTQASHQVLR DILKEVQSNM VPRSMLKEWA LHTFPNATDY WTFRKMFTIQ LALIG FAEF VLHLNRLNPE MLQIAQDTGK LNVAYFRFDI NDATGDLDAN RPVPFRLTPN ISEFLTTIGV SGPLTASMIA VARCFA QPN FKVDGILKTV LRDEIIAWHK KTQEDTSSPL SAAGQPENMD SQQLVSLVQK AVTAIMTRLH NLAQFEGGES KVNTLVA AA NSLDNLCRMD PAWHPWL UniProtKB: Transformation/transcription domain-associated protein |
-分子 #2: E1A-binding protein p400
| 分子 | 名称: E1A-binding protein p400 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO EC番号: 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 343.867312 KDa |
| 配列 | 文字列: MHHGTGPQNV QHQLQRSRAC PGSEGEEQPA HPNPPPSPAA PFAPSASPSA PQSPSYQIQQ LMNRSPATGQ NVNITLQSVG PVVGGNQQI TLAPLPLPSP TSPGFQFSAQ PRRFEHGSPS YIQVTSPLSQ QVQTQSPTQP SPGPGQALQN VRAGAPGPGL G LCSSSPTG ...文字列: MHHGTGPQNV QHQLQRSRAC PGSEGEEQPA HPNPPPSPAA PFAPSASPSA PQSPSYQIQQ LMNRSPATGQ NVNITLQSVG PVVGGNQQI TLAPLPLPSP TSPGFQFSAQ PRRFEHGSPS YIQVTSPLSQ QVQTQSPTQP SPGPGQALQN VRAGAPGPGL G LCSSSPTG GFVDASVLVR QISLSPSSGG HFVFQDGSGL TQIAQGAQVQ LQHPGTPITV RERRPSQPHT QSGGTIHHLG PQ SPAAAGG AGLQPLASPS HITTANLPPQ ISSIIQGQLV QQQQVLQGPP LPRPLGFERT PGVLLPGAGG AAGFGMTSPP PPT SPSRTA VPPGLSSLPL TSVGNTGMKK VPKKLEEIPP ASPEMAQMRK QCLDYHYQEM QALKEVFKEY LIELFFLQHF QGNM MDFLA FKKKHYAPLQ AYLRQNDLDI EEEEEEEEEE EEKSEVINDE VKVVTGKDGQ TGTPVAIATQ LPPKVSAAFS SQQQP FQQA LAGSLVAGAG STVETDLFKR QQAMPSTGMA EQSKRPRLEV GHQGVVFQHP GADAGVPLQQ LMPTAQGGMP PTPQAA QLA GQRQSQQQYD PSTGPPVQNA ASLHTPLPQL PGRLPPAGVP TAALSSALQF AQQPQVVEAQ TQLQIPVKTQ QPNVPIP AP PSSQLPIPPS QPAQLALHVP TPGKVQVQAS QLSSLPQMVA STRLPVDPAP PCPRPLPTSS TSSLAPVSGS GPGPSPAR S SPVNRPSSAT NKALSPVTSR TPGVVASAPT KPQSPAQNAT SSQDSSQDTL TEQITLENQV HQRIAELRKA GLWSQRRLP KLQEAPRPKS HWDYLLEEMQ WMATDFAQER RWKVAAAKKL VRTVVRHHEE KQLREERGKK EEQSRLRRIA ASTAREIECF WSNIEQVVE IKLRVELEEK RKKALNLQKV SRRGKELRPK GFDALQESSL DSGMSGRKRK ASISLTDDEV DDEEETIEEE E ANEGVVDH QTELSNLAKE AELPLLDLMK LYEGAFLPSS QWPRPKPDGE DTSGEEDADD CPGDRESRKD LVLIDSLFIM DQ FKAAERM NIGKPNAKDI ADVTAVAEAI LPKGSARVTT SVKFNAPSLL YGALRDYQKI GLDWLAKLYR KNLNGILADE AGL GKTVQI IAFFAHLACN EGNWGPHLVV VRSCNILKWE LELKRWCPGL KILSYIGSHR ELKAKRQEWA EPNSFHVCIT SYTQ FFRGL TAFTRVRWKC LVIDEMQRVK GMTERHWEAV FTLQSQQRLL LIDSPLHNTF LELWTMVHFL VPGISRPYLS SPLRA PSEE SQDYYHKVVI RLHRVTQPFI LRRTKRDVEK QLTKKYEHVL KCRLSNRQKA LYEDVILQPG TQEALKSGHF VNVLSI LVR LQRICNHPGL VEPRHPGSSY VAGPLEYPSA SLILKALERD FWKEADLSMF DLIGLENKIT RHEAELLSKK KIPRKLM EE ISTSAAPAAR PAAAKLKASR LFQPVQYGQK PEGRTVAFPS THPPRTAAPT TASAAPQGPL RGRPPIATFS ANPEAKAA A APFQTSQASA SAPRHQPASA SSTAASPAHP AKLRAQTTAQ ASTPGQPPPQ PQAPSHAAGQ SALPQRLVLP SQAQARLPS GEVVKIAQLA SITGPQSRVA QPETPVTLQF QGSKFTLSHS QLRQLTAGQP LQLQGSVLQI VSAPGQPYLR APGPVVMQTV SQAGAVHGA LGSKPPAGGP SPAPLTPQVG VPGRVAVNAL AVGEPGTASK PASPIGGPTQ EEKTRLLKER LDQIYLVNER R CSQAPVYG RDLLRICALP SHGRVQWRGS LDGRRGKEAG PAHSYTSSSE SPSELMLTLC RCGESLQDVI DRVAFVIPPV VA APPSLRV PRPPPLYSHR MRILRQGLRE HAAPYFQQLR QTTAPRLLQF PELRLVQFDS GKLEALAILL QKLKSEGRRV LIL SQMILM LDILEMFLNF HYLTYVRIDE NASSEQRQEL MRSFNRDRRI FCAILSTHSR TTGINLVEAD TVVFYDNDLN PVMD AKAQE WCDRIGRCKD IHIYRLVSGN SIEEKLLKNG TKDLIREVAA QGNDYSMAFL TQRTIQELFE VYSPMDDAGF PVKAE EFVV LSQEPSVTET IAPKIARPFI EALKSIEYLE EDAQKSAQEG VLGPHTDALS SDSENMPCDE EPSQLEELAD FMEQLT PIE KYALNYLELF HTSIEQEKER NSEDAVMTAV RAWEFWNLKT LQEREARLRL EQEEAELLTY TREDAYSMEY VYEDVDG QT EVMPLWTPPT PPQDDSDIYL DSVMCLMYEA TPIPEAKLPP VYVRKERKRH KTDPSAAGRK KKQRHGEAVV PPRSLFDR A TPGLLKIRRE GKEQKKNILL KQQVPFAKPL PTFAKPTAEP GQDNPEWLIS EDWALLQAVK QLLELPLNLT IVSPAHTPN WDLVSDVVNS CSRIYRSSKQ CRNRYENVII PREEGKSKNN RPLRTSQIYA QDENATHTQL YTSHFDLMKM TAGKRSPPIK PLLGMNPFQ KNPKHASVLA ESGINYDKPL PPIQVASLRA ERIAKEKKAL ADQQKAQQPA VAQPPPPQPQ PPPPPQQPPP P LPQPQAAG SQPPAGPPAV QPQPQPQPQT QPQPVQAPAK AQPAITTGGS AAVLAGTIKT SVTGTSMPTG AVSGNVIVNT IA GVPAATF QSINKRLASP VAPGALTTPG GSAPAQVVHT QPPPRAVGSP ATATPDLVSM ATTQGVRAVT SVTASAVVTT NLT PVQTPA RSLVPQVSQA TGVQLPGKTI TPAHFQLLRQ QQQQQQQQQQ QQQQQQQQQQ QQQQQQQQTT TTSQVQVPQI QGQA QSPAQ IKAVGKLTPE HLIKMQKQKL QMPPQPPPPQ AQSAPPQPTA QVQVQTSQPP QQQSPQLTTV TAPRPGALLT GTTVA NLQV ARLTRVPTSQ LQAQGQMQTQ APQPAQVALA KPPVVSVPAA VVSSPGVTTL PMNVAGISVA IGQPQKAAGQ TVVAQP VHM QQLLKLKQQA VQQQKAIQPQ AAQGPAAVQQ KITAQQITTP GAQQKVAYAA QPALKTQFLT TPISQAQKLA GAQQVQT QI QVAKLPQVVQ QQTPVASIQQ VASASQQASP QTVALTQATA AGQQVQMIPA VTATAQVVQQ KLIQQQVVTT ASAPLQTP G APNPAQVPAS SDSPSQQPKL QMRVPAVRLK TPTKPPCQ UniProtKB: E1A-binding protein p400 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: 4D-STEM / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: INSILICO MODEL |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.4 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 23442 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
 ムービー
ムービー コントローラー
コントローラー





















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)