+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM Structure of the TEFM bound Human Mitochondrial Transcription Elongation Complex in a Closed Fingers Domain Conformation | |||||||||
 マップデータ マップデータ | Structure of the Transcription Elongation Complex of Human Mitochondrial Polymerase with the Closed Conformation of the Fingers Domain | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Mitochondrial RNA Polymerase / Nucleotide Selection / Nucleotide Discrimination / TRANSCRIPTION / Protein-RNA-DNA Complex / POLRMT / TEFM / TRANSCRIPTION-DNA-RNA complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報transcription elongation by mitochondrial RNA polymerase / Mitochondrial transcription initiation / mitochondrial DNA-directed RNA polymerase complex / mitochondrial promoter sequence-specific DNA binding / transcription initiation at mitochondrial promoter / mitochondrial transcription / DNA primase activity / oxidative phosphorylation / DNA polymerase processivity factor activity / mitochondrial nucleoid ...transcription elongation by mitochondrial RNA polymerase / Mitochondrial transcription initiation / mitochondrial DNA-directed RNA polymerase complex / mitochondrial promoter sequence-specific DNA binding / transcription initiation at mitochondrial promoter / mitochondrial transcription / DNA primase activity / oxidative phosphorylation / DNA polymerase processivity factor activity / mitochondrial nucleoid / Transcriptional activation of mitochondrial biogenesis / DNA-directed 5'-3' RNA polymerase activity / DNA-directed RNA polymerase / 3'-5'-RNA exonuclease activity / sequence-specific DNA binding / mitochondrial matrix / ribonucleoprotein complex / protein-containing complex / mitochondrion / RNA binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) / synthetic construct (人工物) Homo sapiens (ヒト) / synthetic construct (人工物) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.54 Å | |||||||||
 データ登録者 データ登録者 | Herbine KH / Nayak AR / Temiakov D | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2024 ジャーナル: Nat Commun / 年: 2024タイトル: Structural basis for substrate binding and selection by human mitochondrial RNA polymerase. 著者: Karl Herbine / Ashok R Nayak / Dmitry Temiakov /  要旨: The mechanism by which RNAP selects cognate substrates and discriminates between deoxy and ribonucleotides is of fundamental importance to the fidelity of transcription. Here, we present cryo-EM ...The mechanism by which RNAP selects cognate substrates and discriminates between deoxy and ribonucleotides is of fundamental importance to the fidelity of transcription. Here, we present cryo-EM structures of human mitochondrial transcription elongation complexes that reveal substrate ATP bound in Entry and Insertion Sites. In the Entry Site, the substrate binds along the O helix of the fingers domain of mtRNAP but does not interact with the templating DNA base. Interactions between RNAP and the triphosphate moiety of the NTP in the Entry Site ensure discrimination against nucleosides and their diphosphate and monophosphate derivatives but not against non-cognate rNTPs and dNTPs. Closing of the fingers domain over the catalytic site results in delivery of both the templating DNA base and the substrate into the Insertion Site and recruitment of the catalytic magnesium ions. The cryo-EM data also reveal a conformation adopted by mtRNAP to reject a non-cognate substrate from its active site. Our findings establish a structural basis for substrate binding and suggest a unified mechanism of NTP selection for single-subunit RNAPs. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_44448.map.gz emd_44448.map.gz | 85.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-44448-v30.xml emd-44448-v30.xml emd-44448.xml emd-44448.xml | 22.6 KB 22.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_44448_fsc.xml emd_44448_fsc.xml | 9.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_44448.png emd_44448.png | 91.3 KB | ||
| Filedesc metadata |  emd-44448.cif.gz emd-44448.cif.gz | 7.9 KB | ||
| その他 |  emd_44448_half_map_1.map.gz emd_44448_half_map_1.map.gz emd_44448_half_map_2.map.gz emd_44448_half_map_2.map.gz | 91.8 MB 91.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-44448 http://ftp.pdbj.org/pub/emdb/structures/EMD-44448 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-44448 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-44448 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_44448_validation.pdf.gz emd_44448_validation.pdf.gz | 672.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_44448_full_validation.pdf.gz emd_44448_full_validation.pdf.gz | 672 KB | 表示 | |
| XML形式データ |  emd_44448_validation.xml.gz emd_44448_validation.xml.gz | 17.7 KB | 表示 | |
| CIF形式データ |  emd_44448_validation.cif.gz emd_44448_validation.cif.gz | 22.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-44448 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-44448 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-44448 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-44448 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_44448.map.gz / 形式: CCP4 / 大きさ: 98.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_44448.map.gz / 形式: CCP4 / 大きさ: 98.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Structure of the Transcription Elongation Complex of Human Mitochondrial Polymerase with the Closed Conformation of the Fingers Domain | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.8348 Å | ||||||||||||||||||||||||||||||||||||
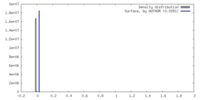
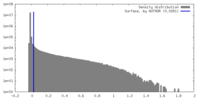
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Structure of the Transcription Elongation Complex of Human Mitoch...
| 全体 | 名称: Structure of the Transcription Elongation Complex of Human Mitochondrial Polymerase with the Closed Conformation of the Fingers Domain |
|---|---|
| 要素 |
|
-超分子 #1: Structure of the Transcription Elongation Complex of Human Mitoch...
| 超分子 | 名称: Structure of the Transcription Elongation Complex of Human Mitochondrial Polymerase with the Closed Conformation of the Fingers Domain タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#5 詳細: Human Mitochondrial RNA polymerase (d119) and Human TEFM (d135) assembled on an RNA-DNA Scaffold in presence of methylene a,b-ATP |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 227 KDa |
-分子 #1: Transcription elongation factor, mitochondrial
| 分子 | 名称: Transcription elongation factor, mitochondrial / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 27.262389 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MDENAKEPEN RFLRKLLKPD IERERLKAVN SIISIVFGTR RIAWAHLDRK LTVLDWQQSD RWSLMRGIYS SSVYLEEISS IISKMPKAD FYVLEKTGLS IQNSSLFPIL LHFHIMEAML YALLNKTFAQ DGQHQVLSMN RNAVGKHFEL MIGDSRTSGK E LVKQFLFD ...文字列: MDENAKEPEN RFLRKLLKPD IERERLKAVN SIISIVFGTR RIAWAHLDRK LTVLDWQQSD RWSLMRGIYS SSVYLEEISS IISKMPKAD FYVLEKTGLS IQNSSLFPIL LHFHIMEAML YALLNKTFAQ DGQHQVLSMN RNAVGKHFEL MIGDSRTSGK E LVKQFLFD SILKADPRVF FPSDKIVHYR QMFLSTELQR VEELYDSLLQ AIAFYELAVF DSQPLEHHHH HHHH UniProtKB: Transcription elongation factor, mitochondrial |
-分子 #2: DNA-directed RNA polymerase, mitochondrial
| 分子 | 名称: DNA-directed RNA polymerase, mitochondrial / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 127.150102 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGHHHHHHGR WAKILEKDKR TQQMRMQRLK AKLQMPFQSG EFKALTRRLQ VEPRLLSKQM AGCLEDCTRQ APESPWEEQL ARLLQEAPG KLSLDVEQAP SGQHSQAQLS GQQQRLLAFF KCCLLTDQLP LAHHLLVVHH GQRQKRKLLT LDMYNAVMLG W ARQGAFKE ...文字列: MGHHHHHHGR WAKILEKDKR TQQMRMQRLK AKLQMPFQSG EFKALTRRLQ VEPRLLSKQM AGCLEDCTRQ APESPWEEQL ARLLQEAPG KLSLDVEQAP SGQHSQAQLS GQQQRLLAFF KCCLLTDQLP LAHHLLVVHH GQRQKRKLLT LDMYNAVMLG W ARQGAFKE LVYVLFMVKD AGLTPDLLSY AAALQCMGRQ DQDAGTIERC LEQMSQEGLK LQALFTAVLL SEEDRATVLK AV HKVKPTF SLPPQLPPPV NTSKLLRDVY AKDGRVSYPK LHLPLKTLQC LFEKQLHMEL ASRVCVVSVE KPTLPSKEVK HAR KTLKTL RDQWEKALCR ALRETKNRLE REVYEGRFSL YPFLCLLDER EVVRMLLQVL QALPAQGESF TTLARELSAR TFSR HVVQR QRVSGQVQAL QNHYRKYLCL LASDAEVPEP CLPRQYWEAL GAPEALREQP WPLPVQMELG KLLAEMLVQA TQMPC SLDK PHRSSRLVPV LYHVYSFRNV QQIGILKPHP AYVQLLEKAA EPTLTFEAVD VPMLCPPLPW TSPHSGAFLL SPTKLM RTV EGATQHQELL ETCPPTALHG ALDALTQLGN CAWRVNGRVL DLVLQLFQAK GCPQLGVPAP PSEAPQPPEA HLPHSAA PA RKAELRRELA HCQKVAREMH SLRAEALYRL SLAQHLRDRV FWLPHNMDFR GRTYPCPPHF NHLGSDVARA LLEFAQGR P LGPHGLDWLK IHLVNLTGLK KREPLRKRLA FAEEVMDDIL DSADQPLTGR KWWMGAEEPW QTLACCMEVA NAVRASDPA AYVSHLPVHQ DGSCNGLQHY AALGRDSVGA ASVNLEPSDV PQDVYSGVAA QVEVFRRQDA QRGMRVAQVL EGFITRKVVK QTVMTVVYG VTRYGGRLQI EKRLRELSDF PQEFVWEASH YLVRQVFKSL QEMFSGTRAI QHWLTESARL ISHMGSVVEW V TPLGVPVI QPYRLDSKVK QIGGGIQSIT YTHNGDISRK PNTRKQKNGF PPNFIHSLDS SHMMLTALHC YRKGLTFVSV HD CYWTHAA DVSVMNQVCR EQFVRLHSEP ILQDLSRFLV KRFCSEPQKI LEASQLKETL QAVPKPGAFD LEQVKRSTYF FS UniProtKB: DNA-directed RNA polymerase, mitochondrial |
-分子 #3: Non-Template Strand DNA (NT27mt_+1T)
| 分子 | 名称: Non-Template Strand DNA (NT27mt_+1T) / タイプ: dna / ID: 3 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 10.474738 KDa |
| 配列 | 文字列: (DG)(DG)(DA)(DC)(DA)(DT)(DG)(DG)(DT)(DG) (DT)(DA)(DA)(DT)(DT)(DA)(DT)(DT)(DT)(DC) (DG)(DT)(DC)(DG)(DC)(DC)(DA)(DG)(DA) (DC)(DG)(DA)(DC)(DC) |
-分子 #5: Template Strand DNA (TS31mt_+1A)
| 分子 | 名称: Template Strand DNA (TS31mt_+1A) / タイプ: dna / ID: 5 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 10.428664 KDa |
| 配列 | 文字列: (DG)(DG)(DT)(DC)(DG)(DT)(DC)(DT)(DG)(DG) (DC)(DG)(DA)(DG)(DC)(DG)(DC)(DG)(DC)(DC) (DG)(DT)(DT)(DA)(DC)(DA)(DC)(DC)(DA) (DT)(DG)(DT)(DC)(DC) |
-分子 #4: RNA (RNA14mt)
| 分子 | 名称: RNA (RNA14mt) / タイプ: rna / ID: 4 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 4.493723 KDa |
| 配列 | 文字列: AGUCUGCGGC GCGC |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.75 mg/mL |
|---|---|
| 緩衝液 | pH: 7.9 詳細: 20 mM Tris Buffer, pH 7.9, 150 mM NaCl, 10 mM DTT, and 5 mM MgCl2 |
| グリッド | モデル: UltrAuFoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: GOLD / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. / 前処理 - 雰囲気: AIR / 詳細: 15 mA Discharge |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
| 詳細 | 5 uM complex of (D119) wild-type mtRNAP, (D50) wild-type TEFM, an RNA-DNA scaffold (R14/T31_+1A/NT27_+1T), and a,b methylene ATP. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - スリット幅: 20 eV |
| 詳細 | Preliminary grid screening performed manually using TFS Glacios. |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 2 / 実像数: 21764 / 平均露光時間: 1.89 sec. / 平均電子線量: 50.6 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.8 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Source name: PDB / Chain - Initial model type: experimental model |
|---|---|
| 詳細 | Initial Fitting was done in Chimera and flexible/refined fitting done with Coot Phenix Real-Space Refinement. |
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT / 温度因子: 45 当てはまり具合の基準: Cross-correlation Coefficient |
| 得られたモデル |  PDB-9bdc: |
 ムービー
ムービー コントローラー
コントローラー












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)