+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-4264 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM Structure of INO80core | ||||||||||||
 マップデータ マップデータ | |||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | DNA BINDING PROTEIN | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報DASH complex / protein transport along microtubule to mitotic spindle pole body / mitotic sister chromatid biorientation / attachment of spindle microtubules to kinetochore / attachment of mitotic spindle microtubules to kinetochore / Ino80 complex / ATP-dependent activity, acting on DNA / helicase activity / mitotic spindle / kinetochore ...DASH complex / protein transport along microtubule to mitotic spindle pole body / mitotic sister chromatid biorientation / attachment of spindle microtubules to kinetochore / attachment of mitotic spindle microtubules to kinetochore / Ino80 complex / ATP-dependent activity, acting on DNA / helicase activity / mitotic spindle / kinetochore / chromatin organization / DNA helicase / chromatin remodeling / DNA repair / ATP hydrolysis activity / ATP binding / nucleus 類似検索 - 分子機能 | ||||||||||||
| 生物種 |  Chaetomium thermophilum var. thermophilum DSM 1495 (菌類) Chaetomium thermophilum var. thermophilum DSM 1495 (菌類) | ||||||||||||
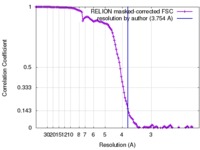
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.754 Å | ||||||||||||
 データ登録者 データ登録者 | Eustermann S / Schall K | ||||||||||||
| 資金援助 |  ドイツ, 3件 ドイツ, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2018 ジャーナル: Nature / 年: 2018タイトル: Structural basis for ATP-dependent chromatin remodelling by the INO80 complex. 著者: Sebastian Eustermann / Kevin Schall / Dirk Kostrewa / Kristina Lakomek / Mike Strauss / Manuela Moldt / Karl-Peter Hopfner /  要旨: In the eukaryotic nucleus, DNA is packaged in the form of nucleosomes, each of which comprises about 147 base pairs of DNA wrapped around a histone protein octamer. The position and histone ...In the eukaryotic nucleus, DNA is packaged in the form of nucleosomes, each of which comprises about 147 base pairs of DNA wrapped around a histone protein octamer. The position and histone composition of nucleosomes is governed by ATP-dependent chromatin remodellers such as the 15-subunit INO80 complex . INO80 regulates gene expression, DNA repair and replication by sliding nucleosomes, the exchange of histone H2A.Z with H2A, and the positioning of + 1 and -1 nucleosomes at promoter DNA. The structures and mechanisms of these remodelling reactions are currently unknown. Here we report the cryo-electron microscopy structure of the evolutionarily conserved core of the INO80 complex from the fungus Chaetomium thermophilum bound to a nucleosome, at a global resolution of 4.3 Å and with major parts at 3.7 Å. The INO80 core cradles one entire gyre of the nucleosome through multivalent DNA and histone contacts. An Rvb1/Rvb2 AAA ATPase heterohexamer is an assembly scaffold for the complex and acts as a 'stator' for the motor and nucleosome-gripping subunits. The Swi2/Snf2 ATPase motor binds to nucleosomal DNA at superhelical location -6, unwraps approximately 15 base pairs, disrupts the H2A-DNA contacts and is poised to pump entry DNA into the nucleosome. Arp5 and Ies6 bind superhelical locations -2 and -3 to act as a counter grip for the motor, on the other side of the H2A-H2B dimer. The Arp5 insertion domain forms a grappler element that binds the nucleosome dyad, connects the Arp5 actin-fold and entry DNA over a distance of about 90 Å and packs against histone H2A-H2B near the 'acidic patch'. Our structure together with biochemical data suggests a unified mechanism for nucleosome sliding and histone editing by INO80. The motor is part of a macromolecular ratchet, persistently pumping entry DNA across the H2A-H2B dimer against the Arp5 grip until a large nucleosome translocation step occurs. The transient exposure of H2A-H2B by motor activity as well as differential recognition of H2A.Z and H2A may regulate histone exchange. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_4264.map.gz emd_4264.map.gz | 88.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-4264-v30.xml emd-4264-v30.xml emd-4264.xml emd-4264.xml | 23.5 KB 23.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_4264_fsc.xml emd_4264_fsc.xml | 12.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_4264.png emd_4264.png | 48.6 KB | ||
| Filedesc metadata |  emd-4264.cif.gz emd-4264.cif.gz | 8.6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-4264 http://ftp.pdbj.org/pub/emdb/structures/EMD-4264 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4264 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4264 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_4264_validation.pdf.gz emd_4264_validation.pdf.gz | 253.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_4264_full_validation.pdf.gz emd_4264_full_validation.pdf.gz | 252.6 KB | 表示 | |
| XML形式データ |  emd_4264_validation.xml.gz emd_4264_validation.xml.gz | 12.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4264 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4264 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4264 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4264 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_4264.map.gz / 形式: CCP4 / 大きさ: 149.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_4264.map.gz / 形式: CCP4 / 大きさ: 149.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.06 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : INO80core masked reconstruction
| 全体 | 名称: INO80core masked reconstruction |
|---|---|
| 要素 |
|
-超分子 #1: INO80core masked reconstruction
| 超分子 | 名称: INO80core masked reconstruction / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#6 詳細: 3xRvb1, 3x Rvb2, Ino80insert, Arp5, Ies2, Ies6 other parts of complex are masked out |
|---|---|
| 由来(天然) | 生物種:  Chaetomium thermophilum var. thermophilum DSM 1495 (菌類) Chaetomium thermophilum var. thermophilum DSM 1495 (菌類) |
| 分子量 | 理論値: 600 KDa |
-分子 #1: RuvB-like helicase
| 分子 | 名称: RuvB-like helicase / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO / EC番号: DNA helicase |
|---|---|
| 由来(天然) | 生物種:  Chaetomium thermophilum var. thermophilum DSM 1495 (菌類) Chaetomium thermophilum var. thermophilum DSM 1495 (菌類)株: DSM 1495 / CBS 144.50 / IMI 039719 |
| 分子量 | 理論値: 50.451848 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MVQISEVRGN TRDHRTAAHT HIKGLGLNSS GIAEKQAAGF VGQCAAREAC GVVVDLIKAH KMAGRGVLLA GGPGTGKTAL ALAISQELG TKIPFCPITG SEIYSTEVKK TEVLMENFRR AIGLRVRETK DVYEGEVTEM TPEEAENPLG GYGKTISTLL I GLKSARGQ ...文字列: MVQISEVRGN TRDHRTAAHT HIKGLGLNSS GIAEKQAAGF VGQCAAREAC GVVVDLIKAH KMAGRGVLLA GGPGTGKTAL ALAISQELG TKIPFCPITG SEIYSTEVKK TEVLMENFRR AIGLRVRETK DVYEGEVTEM TPEEAENPLG GYGKTISTLL I GLKSARGQ KKLRLDPSIY EAIQKERVQV GDVIYIETNT GACKRVGRSD AYATEFDLEA EEYVPIPKGE VHKKKEIVQD VT LHDLDVA NARPQGGQDI ISMMGQLMKP KMTEITDKLR MEINKVVQKY INQGVAELIP GVLFIDEAHM LDIECFTYLN KAL ESPIAP IVVLASNRGI ATIRGADDLK AAHGIPPDFL QRLLIIPTHP YEPDEIRRIV RIRAQTEGVQ LTDAAVDRVA EHGV RISLR YCLQLLAPAS ILARVNGRTQ VDVQDIAEAE ELFLDARRSA NILTSTGESG GLHGFIS UniProtKB: RuvB-like helicase |
-分子 #2: RuvB-like helicase
| 分子 | 名称: RuvB-like helicase / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO / EC番号: DNA helicase |
|---|---|
| 由来(天然) | 生物種:  Chaetomium thermophilum var. thermophilum DSM 1495 (菌類) Chaetomium thermophilum var. thermophilum DSM 1495 (菌類)株: DSM 1495 / CBS 144.50 / IMI 039719 |
| 分子量 | 理論値: 53.212746 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MAAPLVTSVT ETKELRGLNL IAAHSHIRGL GVDADTLEPR PSSQGLVGQE KARKAAAVVL EMIKQGKIAG RAVLIAGPPS TGKTAIAMG MAQSLGQDVP FTTLAASEIF SLEMSKTEAL TQAFRKSIGV RIKEESEIME GEVVEIQIDR SVTGGAKQGK L TIKTTDME ...文字列: MAAPLVTSVT ETKELRGLNL IAAHSHIRGL GVDADTLEPR PSSQGLVGQE KARKAAAVVL EMIKQGKIAG RAVLIAGPPS TGKTAIAMG MAQSLGQDVP FTTLAASEIF SLEMSKTEAL TQAFRKSIGV RIKEESEIME GEVVEIQIDR SVTGGAKQGK L TIKTTDME AIYDMGSKMI DAMTKERVMA GDIISIDKSS GKITKLGRSY ARSRDYDAMG VDTKFLQCPE GELQKRKEVV HT VSLHEID VINSRTQGFL ALFSGDTGEI RSEIRDQINT KVAEWKEEGK AEIVPGVLFI DEVHMLDIEC FSYINRALES DLA PIVIMA SNRGVSRIRG TDYKSPHGLP LDFLDRVVII NTHPYTPDEL RQILSIRAQE EEVDLTPDAL ALLTKIGQEA GLRY ASNLI TTSQLIAAKR RAKQVGVEDV QRSFKLFYDP ARSVRFVQES EKRLIGNDGV VDFSYQGAAE AAAPTLPAAA PVDPV GGEK MDMS UniProtKB: RuvB-like helicase |
-分子 #3: Ino80
| 分子 | 名称: Ino80 / タイプ: protein_or_peptide / ID: 3 / 詳細: C-terminal double FlagTag / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Chaetomium thermophilum var. thermophilum DSM 1495 (菌類) Chaetomium thermophilum var. thermophilum DSM 1495 (菌類) |
| 分子量 | 理論値: 127.529781 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MTDSYATKAS NLKKTAILAS KEAKRWQLRT NKGTKDLQAR AKRVMRDMMG FWKRNEREER DLRKAAERLE LENARKEEAD REAARQRRK LNFLISQTEL YSHFISKKIK THEVERSTDH PDVATDEKDK IPEPTLNINV PEPTGPIAPK VTDFNSLDFD N EDESALQA ...文字列: MTDSYATKAS NLKKTAILAS KEAKRWQLRT NKGTKDLQAR AKRVMRDMMG FWKRNEREER DLRKAAERLE LENARKEEAD REAARQRRK LNFLISQTEL YSHFISKKIK THEVERSTDH PDVATDEKDK IPEPTLNINV PEPTGPIAPK VTDFNSLDFD N EDESALQA AAMANAQNAI AEAQKKAREF NKDETKLDED GEMNFQHPEL TEFEVAQPKL LNCQLKEYQL KGLNWLVNLY EQ GINGILA DEMGLGKTVQ SISVMAYLAE RYDIWGPFLV VAPASTLHNW QQEVSKFVPD FKVLPYWGTA ADRKVLRKFW DRK HTTYKK DSPFHVMITS YQLVVSDVAY FQKMKWQYMI LDEAQAIKSS QSSRWKCLLG FHCRNRLLLT GTPIQNNMQE LWAL LHFIM PSLFDSHDEF SEWFSKDIES HAQSNTKLNE DQLKRLHMIL KPFMLRRVKK HVQKELGDKI EIDVFCELSY RQRAM YQSL RNQISIMDLI EKATVGDNED SATLMNLVMQ FRKVCNHPDL FERADTSSPF FCGHFAETGS FLREGTNVAL GYSTRS LVE YRLPRLIWCD GGRLDKPGPG NLVAGFRSKY LNHMMNIWTP ENIRSSLEGI ENFTWLRFVD TSLQEAYRAS HTDVFAR AV DLASKQNRLG HMQIVYDEPE DKKWTPVHAL FQICERENPK AVAEITTEGV LRDLMNIARV KYRELGLCRL EKAARPRA S APPIEVVCDS RSAVIERENI MFHPAMRKAL FGPTPSEIKE ASFGPRPVTL YPPRALLPAP DHDKQRFTNI TVPSMARFV TDSGKLAKLD ELLRELKEGG HRVLLYFQMT RMIDLMEEYL TYRNYKYCRL DGSTKLEDRR DTVADFQTRP EIFIFLLSTR AGGLGINLT TADTVIFYDS DWNPTIDSQA MDRAHRLGQT KQVTVYRLIT RGTIEERIRK RALQKEEVQR VVITGTGSVD F SGRRPPEN RNRDIAMWLA DDEQAEMIER REKELIESGE YDKIMQQRRK GGKRKRGAAN GDTVPSLEDM YHEGEGHFDD NK GSGAATP VDADSLGRGG KRKKAGGSKK AKTTKQRLAI ADGEIDDGEI DIDYKDDDDK GTDYKDDDDK |
-分子 #4: les2
| 分子 | 名称: les2 / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Chaetomium thermophilum var. thermophilum DSM 1495 (菌類) Chaetomium thermophilum var. thermophilum DSM 1495 (菌類)株: DSM 1495 / CBS 144.50 / IMI 039719 |
| 分子量 | 理論値: 53.258902 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MSTRPRRHAA QRASQAITDL ADRDRESDHS HGPISSRMSS FNSSSRSRLP GKGIASVSRS EAGGASDPEH IHLTVKLPSS KLRQATSSS GIKKAGSVGS SSSSSGGGKA AVKRARGGKR SRVLESSEEE EEENEVEVLG DEDEEEEEEE DEIEVREGEG Y DEDEEDVE ...文字列: MSTRPRRHAA QRASQAITDL ADRDRESDHS HGPISSRMSS FNSSSRSRLP GKGIASVSRS EAGGASDPEH IHLTVKLPSS KLRQATSSS GIKKAGSVGS SSSSSGGGKA AVKRARGGKR SRVLESSEEE EEENEVEVLG DEDEEEEEEE DEIEVREGEG Y DEDEEDVE DEDEEMQDLG EEDADGEDDE MDVDAEGEED ADGDVNMDAG VVGARATTVR AVPPAIKVTK PPKESPSNGK AA TASKAND NAVPVKRPAP DSDDESLSSL ESEPEEEVNV AGGEDAEGED DDAEGEVDAE GEEEEEEEEI EVADEDAEGE DVE QDEDED EEEEDDDDEM ISRAQTPDMS RLTARQRARL GEASGEYLKL SDEVQSKKHF TAEELSMRRA EMARRRRNLS EKRN EEIKM ETVNKLLKKQ APRTTRRAAQ AAAAAEEAEE AAKQPKRPDP MMIRWVNNKM GSVVAVPEEL LGTHAGVVFG AGPGK GLPA GKMVEEV UniProtKB: INO80 complex subunit B-like conserved region domain-containing protein |
-分子 #5: les6
| 分子 | 名称: les6 / タイプ: protein_or_peptide / ID: 5 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Chaetomium thermophilum var. thermophilum DSM 1495 (菌類) Chaetomium thermophilum var. thermophilum DSM 1495 (菌類)株: DSM 1495 / CBS 144.50 / IMI 039719 |
| 分子量 | 理論値: 23.127523 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MSNPDAQSAQ AAHQALVEQL DLHSIHKTFR NPNWRPNQRR NKTIKAILGE SQRKEASSTS AVATPRADDN GGGSGADTPA NNDNNDGLS TSGTSTPANG NGSGAGTPAS NGQPNLAQAS RSLQKLVLEK SLASAQAPDK KAANGFASSA PTATYTNIES A PSLAPMKH ...文字列: MSNPDAQSAQ AAHQALVEQL DLHSIHKTFR NPNWRPNQRR NKTIKAILGE SQRKEASSTS AVATPRADDN GGGSGADTPA NNDNNDGLS TSGTSTPANG NGSGAGTPAS NGQPNLAQAS RSLQKLVLEK SLASAQAPDK KAANGFASSA PTATYTNIES A PSLAPMKH YCDVTGLPAP YLDPKTRLRY HNKEIFAMIR NLPQGMGEQF LEARGAHTVL K UniProtKB: Vps72/YL1 C-terminal domain-containing protein |
-分子 #6: Arp5
| 分子 | 名称: Arp5 / タイプ: protein_or_peptide / ID: 6 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Chaetomium thermophilum var. thermophilum DSM 1495 (菌類) Chaetomium thermophilum var. thermophilum DSM 1495 (菌類)株: DSM 1495 / CBS 144.50 / IMI 039719 |
| 分子量 | 理論値: 87.773086 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MAPSAVAEPP PIPQRDEPWK RLPPPTVYPV KEARFEKYIP PQLDGRERAL AQPPGQVAIV IDNGSHSVRA GWNFEDKPRL AIPPIMSKY RDRKMGKTFS FAGSDCYADT TARSHIRNAF EAGTGIVSNW DVMEHVLDYV FVKLGMNECD GAIDMPIVMT E AVANLPYS ...文字列: MAPSAVAEPP PIPQRDEPWK RLPPPTVYPV KEARFEKYIP PQLDGRERAL AQPPGQVAIV IDNGSHSVRA GWNFEDKPRL AIPPIMSKY RDRKMGKTFS FAGSDCYADT TARSHIRNAF EAGTGIVSNW DVMEHVLDYV FVKLGMNECD GAIDMPIVMT E AVANLPYS RKSMSEIIFE CYGAPSLVYG IDSLFSFRHN QGQTGLVVSS SYSATHVIPV YNRKALLSQA IRLNWGGWHM AE YMLKLLK LKYYTGFPGK LNSSQTEHMV RDFCYVSLDY DRELAGYLDW TGLEDRERIV QYPYTEEVVV QKTEEELARI AER KKESGR RLQEQAAKMR LERLMKKEQE LEYYKDIQRR MQGESKKEIK RLLDEAELKD EAALERVIRD LERSIKRARQ KDLG EPEEE EVPDFSLLDV PDDQLDEAGL RQKRQQRLLK SNWEARQRAK AEKEAEKARL AEEARLDEER RKNDLEGWLE EKRQL RLAK LNQLKERERL KADLGNRKSL ASQIRMKNIA NLASDNPTGS GSRKRRRGGA GADQDDDFGA DDADWGVYRS VAIGAN KGD DSDDEEGEED LEAAIRSLEN DLLRYDKTFS YDMTLDAQRD WSKSLLHAFR YGPRPFDPSS QAETHRVHLN VERIRVP EV LFQPAAIAGV DQAGLVEIAG DILCQRLPSL PGIQDAPDAF LRDVFLTGGN TLFQNFDERL RQGLMALLPV GAPLRVRR A QDAILDAWRG AAGWACTEEA KAAWITREEY LEKGGEYIKE HDLGNAFA UniProtKB: Uncharacterized protein |
-分子 #7: ADENOSINE-5'-DIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-DIPHOSPHATE / タイプ: ligand / ID: 7 / コピー数: 6 / 式: ADP |
|---|---|
| 分子量 | 理論値: 427.201 Da |
| Chemical component information |  ChemComp-ADP: |
-分子 #8: ADENOSINE-5'-TRIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-TRIPHOSPHATE / タイプ: ligand / ID: 8 / コピー数: 1 / 式: ATP |
|---|---|
| 分子量 | 理論値: 507.181 Da |
| Chemical component information |  ChemComp-ATP: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL |
|---|---|
| 緩衝液 | pH: 8 詳細: 20 mM HEPES pH 8, 60 mM KCl, 0.5% glycerol, 0.25 mM CaCl2, 20 uM ZnCl2, 0.25 mM DTT, 0.05% Octyl-beta-glucoside |
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 10 sec. / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 281 K / 装置: LEICA EM GP |
| 詳細 | Monodisperse sample: INO80core complex reconstituted with nucleosomal substrate was purified by gelfiltration. Addition of nucleotides or crosslinking was not required. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - 画像ごとのフレーム数: 1-40 / 撮影したグリッド数: 1 / 実像数: 3992 / 平均電子線量: 59.6 e/Å2 詳細: Images were collected in movie mode with 4 frames per second and 10s total aquisition |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 最小 デフォーカス(補正後): 1.3 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 3.5 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー




















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)