+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of the Measles virus Fusion protein in the pre-fusion conformation with bound [FIP-HRC]2-PEG11 | ||||||||||||||||||
 マップデータ マップデータ | map sharp | ||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||
 キーワード キーワード | VIRAL PROTEIN / glycoprotein / immune system / measles / high-resolution / ectodomain / post-fusion / small molecule / inhibitor / complex | ||||||||||||||||||
| 機能・相同性 | Precursor fusion glycoprotein F0, Paramyxoviridae / Fusion glycoprotein F0 / symbiont entry into host cell / fusion of virus membrane with host plasma membrane / viral envelope / host cell plasma membrane / virion membrane / membrane / Fusion glycoprotein F0 機能・相同性情報 機能・相同性情報 | ||||||||||||||||||
| 生物種 |  Measles virus strain Ichinose-B95a (ウイルス) / synthetic construct (人工物) Measles virus strain Ichinose-B95a (ウイルス) / synthetic construct (人工物) | ||||||||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.34 Å | ||||||||||||||||||
 データ登録者 データ登録者 | Zyla D / Saphire EO | ||||||||||||||||||
| 資金援助 |  スイス, スイス,  米国, 5件 米国, 5件
| ||||||||||||||||||
 引用 引用 |  ジャーナル: Science / 年: 2024 ジャーナル: Science / 年: 2024タイトル: A neutralizing antibody prevents postfusion transition of measles virus fusion protein. 著者: Dawid S Zyla / Roberta Della Marca / Gele Niemeyer / Gillian Zipursky / Kyle Stearns / Cameron Leedale / Elizabeth B Sobolik / Heather M Callaway / Chitra Hariharan / Weiwei Peng / Diptiben ...著者: Dawid S Zyla / Roberta Della Marca / Gele Niemeyer / Gillian Zipursky / Kyle Stearns / Cameron Leedale / Elizabeth B Sobolik / Heather M Callaway / Chitra Hariharan / Weiwei Peng / Diptiben Parekh / Tara C Marcink / Ruben Diaz Avalos / Branka Horvat / Cyrille Mathieu / Joost Snijder / Alexander L Greninger / Kathryn M Hastie / Stefan Niewiesk / Anne Moscona / Matteo Porotto / Erica Ollmann Saphire /      要旨: Measles virus (MeV) presents a public health threat that is escalating as vaccine coverage in the general population declines and as populations of immunocompromised individuals, who cannot be ...Measles virus (MeV) presents a public health threat that is escalating as vaccine coverage in the general population declines and as populations of immunocompromised individuals, who cannot be vaccinated, increase. There are no approved therapeutics for MeV. Neutralizing antibodies targeting viral fusion are one potential therapeutic approach but have not yet been structurally characterized or advanced to clinical use. We present cryo-electron microscopy (cryo-EM) structures of prefusion F alone [2.1-angstrom (Å) resolution], F complexed with a fusion-inhibitory peptide (2.3-Å resolution), F complexed with the neutralizing and protective monoclonal antibody (mAb) 77 (2.6-Å resolution), and an additional structure of postfusion F (2.7-Å resolution). In vitro assays and examination of additional EM classes show that mAb 77 binds prefusion F, arrests F in an intermediate state, and prevents transition to the postfusion conformation. These structures shed light on antibody-mediated neutralization that involves arrest of fusion proteins in an intermediate state. | ||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_42595.map.gz emd_42595.map.gz | 97.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-42595-v30.xml emd-42595-v30.xml emd-42595.xml emd-42595.xml | 22.2 KB 22.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_42595.png emd_42595.png | 73 KB | ||
| マスクデータ |  emd_42595_msk_1.map emd_42595_msk_1.map | 103 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-42595.cif.gz emd-42595.cif.gz | 7.1 KB | ||
| その他 |  emd_42595_half_map_1.map.gz emd_42595_half_map_1.map.gz emd_42595_half_map_2.map.gz emd_42595_half_map_2.map.gz | 95.5 MB 95.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-42595 http://ftp.pdbj.org/pub/emdb/structures/EMD-42595 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-42595 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-42595 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_42595_validation.pdf.gz emd_42595_validation.pdf.gz | 1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_42595_full_validation.pdf.gz emd_42595_full_validation.pdf.gz | 1 MB | 表示 | |
| XML形式データ |  emd_42595_validation.xml.gz emd_42595_validation.xml.gz | 13.5 KB | 表示 | |
| CIF形式データ |  emd_42595_validation.cif.gz emd_42595_validation.cif.gz | 15.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42595 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42595 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42595 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42595 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_42595.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_42595.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | map sharp | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.88 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_42595_msk_1.map emd_42595_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
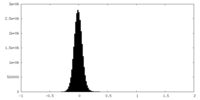
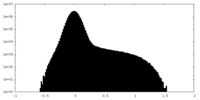
| 密度ヒストグラム |
-ハーフマップ: half 1
| ファイル | emd_42595_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half 2
| ファイル | emd_42595_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Structure of the Measles virus Fusion protein in the pre-fusion c...
| 全体 | 名称: Structure of the Measles virus Fusion protein in the pre-fusion conformation with bound [FIP-HRC]2-PEG11 |
|---|---|
| 要素 |
|
-超分子 #1: Structure of the Measles virus Fusion protein in the pre-fusion c...
| 超分子 | 名称: Structure of the Measles virus Fusion protein in the pre-fusion conformation with bound [FIP-HRC]2-PEG11 タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 |
|---|---|
| 由来(天然) | 生物種:  Measles virus strain Ichinose-B95a (ウイルス) Measles virus strain Ichinose-B95a (ウイルス)株: Ichinose-B95a |
-分子 #1: Fusion glycoprotein F0
| 分子 | 名称: Fusion glycoprotein F0 / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Measles virus strain Ichinose-B95a (ウイルス) Measles virus strain Ichinose-B95a (ウイルス) |
| 分子量 | 理論値: 12.498768 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGLKVNVSAI FMAVLLTLQT PTGQIHWGNL SKIGVVGIGS ASYKVMTRSS HQSLVIKLMP NITLLNNCTR VEIAEYRRLL RTVLEPIRD ALNAMTQNIR PVQSVASSRR HKR UniProtKB: Fusion glycoprotein F0 |
-分子 #2: Fusion glycoprotein F0
| 分子 | 名称: Fusion glycoprotein F0 / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Measles virus strain Ichinose-B95a (ウイルス) Measles virus strain Ichinose-B95a (ウイルス) |
| 分子量 | 理論値: 44.89882 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: FAGVVLAGAA LGVATAAQIT AGIALHQSML NSQAIDNLRA SLETTNQAIE AIRQAGQGMI LAVQGVQDYI NNELIPSMNQ LSCDLIGQK LGLKLLRYYT EILSLFGPSL RDPISAEISI QALSYALGGD INKVLEKLGY SGGDLLGILE SRGIKARITH V DTESYFIV ...文字列: FAGVVLAGAA LGVATAAQIT AGIALHQSML NSQAIDNLRA SLETTNQAIE AIRQAGQGMI LAVQGVQDYI NNELIPSMNQ LSCDLIGQK LGLKLLRYYT EILSLFGPSL RDPISAEISI QALSYALGGD INKVLEKLGY SGGDLLGILE SRGIKARITH V DTESYFIV LSIAYPTLSE IKGVIVHRLE GVSYNIGSQE WYTTVPKYVA TQGYLISNFD ESSCTFMPEG TVCSQNALYP MS PLLQECL RGSTKSCART LVSGSFGNRF ILSQGNLIAN CASILCKCYT TGTIINQDPD KILTYIAADH CPVVEVNGVT IQV GSRRYP DAVYLHRIDL GPPISLGRLD VGTNLGNAIA KLEDAKELLE SSDQILRSMK GLSSTSIGVD DDDKAGWSHP QFEK GGGSG GGSGGGSWSH PQFEK UniProtKB: Fusion glycoprotein F0 |
-分子 #3: [FIP-HRC]2-PEG11
| 分子 | 名称: [FIP-HRC]2-PEG11 / タイプ: protein_or_peptide / ID: 3 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 503.546 Da |
| 配列 | 文字列: (P6S)FFG |
-分子 #6: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 6 / コピー数: 3 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-分子 #7: water
| 分子 | 名称: water / タイプ: ligand / ID: 7 / コピー数: 33 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.38 mg/mL |
|---|---|
| 緩衝液 | pH: 8 / 詳細: HEPES 50 mM, pH 8.0, NaCl 150 mM |
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: GRAPHENE OXIDE / 支持フィルム - トポロジー: CONTINUOUS / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 15 sec. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 298 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 3 / 実像数: 10716 / 平均露光時間: 2.0 sec. / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー














 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)