+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-4225 | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the human CPSF160-WDR33-CPSF30 complex bound to the PAS AAUAAA motif at 3.1 Angstrom resolution. | |||||||||||||||
 マップデータ マップデータ | Output from Relion2 PostProcess | |||||||||||||||
 試料 試料 |
| |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報co-transcriptional RNA 3'-end processing, cleavage and polyadenylation pathway / Inhibition of Host mRNA Processing and RNA Silencing / Processing of Intronless Pre-mRNAs / mRNA cleavage and polyadenylation specificity factor complex / : / collagen trimer / mRNA 3'-UTR AU-rich region binding / Transport of Mature mRNA Derived from an Intronless Transcript / mRNA 3'-end processing / postreplication repair ...co-transcriptional RNA 3'-end processing, cleavage and polyadenylation pathway / Inhibition of Host mRNA Processing and RNA Silencing / Processing of Intronless Pre-mRNAs / mRNA cleavage and polyadenylation specificity factor complex / : / collagen trimer / mRNA 3'-UTR AU-rich region binding / Transport of Mature mRNA Derived from an Intronless Transcript / mRNA 3'-end processing / postreplication repair / tRNA processing in the nucleus / RNA Polymerase II Transcription Termination / Processing of Capped Intron-Containing Pre-mRNA / fibrillar center / mRNA processing / sequence-specific double-stranded DNA binding / spermatogenesis / intracellular membrane-bounded organelle / enzyme binding / RNA binding / zinc ion binding / nucleoplasm / nucleus 類似検索 - 分子機能 | |||||||||||||||
| 生物種 |  Homo sapiens (ヒト) / Homo sapiens (ヒト) /  unidentified adenovirus (ウイルス) unidentified adenovirus (ウイルス) | |||||||||||||||
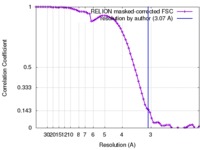
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.07 Å | |||||||||||||||
 データ登録者 データ登録者 | Clerici M / Faini M / Jinek M | |||||||||||||||
| 資金援助 |  ベルギー, ベルギー,  ドイツ, 4件 ドイツ, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2018 ジャーナル: Nat Struct Mol Biol / 年: 2018タイトル: Structural basis of AAUAAA polyadenylation signal recognition by the human CPSF complex. 著者: Marcello Clerici / Marco Faini / Lena M Muckenfuss / Ruedi Aebersold / Martin Jinek /  要旨: Mammalian mRNA biogenesis requires specific recognition of a hexanucleotide AAUAAA motif in the polyadenylation signals (PAS) of precursor mRNA (pre-mRNA) transcripts by the cleavage and ...Mammalian mRNA biogenesis requires specific recognition of a hexanucleotide AAUAAA motif in the polyadenylation signals (PAS) of precursor mRNA (pre-mRNA) transcripts by the cleavage and polyadenylation specificity factor (CPSF) complex. Here we present a 3.1-Å-resolution cryo-EM structure of a core CPSF module bound to the PAS hexamer motif. The structure reveals the molecular interactions responsible for base-specific recognition, providing a rationale for mechanistic differences between mammalian and yeast 3' polyadenylation. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_4225.map.gz emd_4225.map.gz | 28.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-4225-v30.xml emd-4225-v30.xml emd-4225.xml emd-4225.xml | 20.8 KB 20.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_4225_fsc.xml emd_4225_fsc.xml | 7.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_4225.png emd_4225.png | 137.3 KB | ||
| マスクデータ |  emd_4225_msk_1.map emd_4225_msk_1.map | 30.5 MB |  マスクマップ マスクマップ | |
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-4225 http://ftp.pdbj.org/pub/emdb/structures/EMD-4225 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4225 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4225 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_4225_validation.pdf.gz emd_4225_validation.pdf.gz | 552.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_4225_full_validation.pdf.gz emd_4225_full_validation.pdf.gz | 552.1 KB | 表示 | |
| XML形式データ |  emd_4225_validation.xml.gz emd_4225_validation.xml.gz | 9.6 KB | 表示 | |
| CIF形式データ |  emd_4225_validation.cif.gz emd_4225_validation.cif.gz | 12.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4225 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4225 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4225 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4225 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_4225.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_4225.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Output from Relion2 PostProcess | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.058 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_4225_msk_1.map emd_4225_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Human CPSF160-WDR33-CPSF30 heterotrimer bound to the AAUAAA motif...
| 全体 | 名称: Human CPSF160-WDR33-CPSF30 heterotrimer bound to the AAUAAA motif of the Polyadenylation Signal |
|---|---|
| 要素 |
|
-超分子 #1: Human CPSF160-WDR33-CPSF30 heterotrimer bound to the AAUAAA motif...
| 超分子 | 名称: Human CPSF160-WDR33-CPSF30 heterotrimer bound to the AAUAAA motif of the Polyadenylation Signal タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 |
|---|---|
| 分子量 | 理論値: 220 KDa |
-超分子 #2: Human CPSF160-WDR33-CPSF30 heterotrimer
| 超分子 | 名称: Human CPSF160-WDR33-CPSF30 heterotrimer / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1-#3 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-超分子 #3: AAUAAA motif of the Polyadenylation Signal
| 超分子 | 名称: AAUAAA motif of the Polyadenylation Signal / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #4 |
|---|---|
| 由来(天然) | 生物種:  unidentified adenovirus (ウイルス) unidentified adenovirus (ウイルス) |
-分子 #1: Cleavage and polyadenylation specificity factor subunit 1
| 分子 | 名称: Cleavage and polyadenylation specificity factor subunit 1 タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 161.346484 KDa |
| 組換発現 | 生物種:  Spodoptera aff. frugiperda 2 RZ-2014 (蝶・蛾) Spodoptera aff. frugiperda 2 RZ-2014 (蝶・蛾) |
| 配列 | 文字列: SNAMYAVYKQ AHPPTGLEFS MYCNFFNNSE RNLVVAGTSQ LYVYRLNRDA EALTKNDRST EGKAHREKLE LAASFSFFGN VMSMASVQL AGAKRDALLL SFKDAKLSVV EYDPGTHDLK TLSLHYFEEP ELRDGFVQNV HTPRVRVDPD GRCAAMLVYG T RLVVLPFR ...文字列: SNAMYAVYKQ AHPPTGLEFS MYCNFFNNSE RNLVVAGTSQ LYVYRLNRDA EALTKNDRST EGKAHREKLE LAASFSFFGN VMSMASVQL AGAKRDALLL SFKDAKLSVV EYDPGTHDLK TLSLHYFEEP ELRDGFVQNV HTPRVRVDPD GRCAAMLVYG T RLVVLPFR RESLAEEHEG LVGEGQRSSF LPSYIIDVRA LDEKLLNIID LQFLHGYYEP TLLILFEPNQ TWPGRVAVRQ DT CSIVAIS LNITQKVHPV IWSLTSLPFD CTQALAVPKP IGGVVVFAVN SLLYLNQSVP PYGVALNSLT TGTTAFPLRT QEG VRITLD CAQATFISYD KMVISLKGGE IYVLTLITDG MRSVRAFHFD KAAASVLTTS MVTMEPGYLF LGSRLGNSLL LKYT EKLQE PPASAVREAA DKEEPPSKKK RVDATAGWSA AGKSVPQDEV DEIEVYGSEA QSGTQLATYS FEVCDSILNI GPCAN AAVG EPAFLSEEFQ NSPEPDLEIV VCSGHGKNGA LSVLQKSIRP QVVTTFELPG CYDMWTVIAP VRKEEEDNPK GEGTEQ EPS TTPEADDDGR RHGFLILSRE DSTMILQTGQ EIMELDTSGF ATQGPTVFAG NIGDNRYIVQ VSPLGIRLLE GVNQLHF IP VDLGAPIVQC AVADPYVVIM SAEGHVTMFL LKSDSYGGRH HRLALHKPPL HHQSKVITLC LYRDLSGMFT TESRLGGA R DELGGRSGPE AEGLGSETSP TVDDEEEMLY GDSGSLFSPS KEEARRSSQP PADRDPAPFR AEPTHWCLLV RENGTMEIY QLPDWRLVFL VKNFPVGQRV LVDSSFGQPT TQGEARREEA TRQGELPLVK EVLLVALGSR QSRPYLLVHV DQELLIYEAF PHDSQLGQG NLKVRFKKVP HNINFREKKP KPSKKKAEGG GAEEGAGARG RVARFRYFED IYGYSGVFIC GPSPHWLLVT G RGALRLHP MAIDGPVDSF APFHNVNCPR GFLYFNRQGE LRISVLPAYL SYDAPWPVRK IPLRCTAHYV AYHVESKVYA VA TSTNTPC ARIPRMTGEE KEFETIERDE RYIHPQQEAF SIQLISPVSW EAIPNARIEL QEWEHVTCMK TVSLRSEETV SGL KGYVAA GTCLMQGEEV TCRGRILIMD VIEVVPEPGQ PLTKNKFKVL YEKEQKGPVT ALCHCNGHLV SAIGQKIFLW SLRA SELTG MAFIDTQLYI HQMISVKNFI LAADVMKSIS LLRYQEESKT LSLVSRDAKP LEVYSVDFMV DNAQLGFLVS DRDRN LMVY MYLPEAKESF GGMRLLRRAD FHVGAHVNTF WRTPCRGATE GLSKKSVVWE NKHITWFATL DGGIGLLLPM QEKTYR RLL MLQNALTTML PHHAGLNPRA FRMLHVDRRT LQNAVRNVLD GELLNRYLYL STMERSELAK KIGTTPDIIL DDLLETD RV TAHF |
-分子 #2: pre-mRNA 3' end processing protein WDR33
| 分子 | 名称: pre-mRNA 3' end processing protein WDR33 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 47.44891 KDa |
| 組換発現 | 生物種:  Spodoptera aff. frugiperda 2 RZ-2014 (蝶・蛾) Spodoptera aff. frugiperda 2 RZ-2014 (蝶・蛾) |
| 配列 | 文字列: SNAMATEIGS PPRFFHMPRF QHQAPRQLFY KRPDFAQQQA MQQLTFDGKR MRKAVNRKTI DYNPSVIKYL ENRIWQRDQR DMRAIQPDA GYYNDLVPPI GMLNNPMNAV TTKFVRTSTN KVKCPVFVVR WTPEGRRLVT GASSGEFTLW NGLTFNFETI L QAHDSPVR ...文字列: SNAMATEIGS PPRFFHMPRF QHQAPRQLFY KRPDFAQQQA MQQLTFDGKR MRKAVNRKTI DYNPSVIKYL ENRIWQRDQR DMRAIQPDA GYYNDLVPPI GMLNNPMNAV TTKFVRTSTN KVKCPVFVVR WTPEGRRLVT GASSGEFTLW NGLTFNFETI L QAHDSPVR AMTWSHNDMW MLTADHGGYV KYWQSNMNNV KMFQAHKEAI REASFSPTDN KFATCSDDGT VRIWDFLRCH EE RILRGHG ADVKCVDWHP TKGLVVSGSK DSQQPIKFWD PKTGQSLATL HAHKNTVMEV KLNLNGNWLL TASRDHLCKL FDI RNLKEE LQVFRGHKKE ATAVAWHPVH EGLFASGGSD GSLLFWHVGV EKEVGGMEMA HEGMIWSLAW HPLGHILCSG SNDH TSKFW TRNRPGDK |
-分子 #3: Cleavage and polyadenylation specificity factor subunit 4
| 分子 | 名称: Cleavage and polyadenylation specificity factor subunit 4 タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 20.422855 KDa |
| 組換発現 | 生物種:  Spodoptera aff. frugiperda 2 RZ-2014 (蝶・蛾) Spodoptera aff. frugiperda 2 RZ-2014 (蝶・蛾) |
| 配列 | 文字列: MQEIIASVDH IKFDLEIAVE QQLGAQPLPF PGMDKSGAAV CEFFLKAACG KGGMCPFRHI SGEKTVVCKH WLRGLCKKGD QCEFLHEYD MTKMPECYFY SKFGECSNKE CPFLHIDPES KIKDCPWYDR GFCKHGPLCR HRHTRRVICV NYLVGFCPEG P SCKFMHPR FELPMGTTEQ |
-分子 #4: RNA (5'-R(P*AP*AP*UP*AP*AP*AP*GP*G)-3')
| 分子 | 名称: RNA (5'-R(P*AP*AP*UP*AP*AP*AP*GP*G)-3') / タイプ: rna / ID: 4 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:  unidentified adenovirus (ウイルス) unidentified adenovirus (ウイルス) |
| 分子量 | 理論値: 3.232036 KDa |
| 配列 | 文字列: ACAAUAAAGG |
-分子 #5: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 5 / コピー数: 3 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.3 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. | ||||||||||||
| 凍結 | 凍結剤: ETHANE-PROPANE / チャンバー内湿度: 100 % / チャンバー内温度: 293 K / 装置: FEI VITROBOT MARK IV / 詳細: 15 seconds wait time prior to blotting. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum LS |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 撮影したグリッド数: 1 / 実像数: 1070 / 平均露光時間: 10.0 sec. / 平均電子線量: 80.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 47259 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.2 µm / 最小 デフォーカス(公称値): 1.1 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー




















 Z
Z Y
Y X
X