+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | mGluR3 in the presence of the agonist LY379268 | ||||||||||||
 マップデータ マップデータ | Full length map | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | GPCR / synaptic protein / MEMBRANE PROTEIN | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Class C/3 (Metabotropic glutamate/pheromone receptors) / group II metabotropic glutamate receptor activity / G protein-coupled glutamate receptor signaling pathway / astrocyte projection / G alpha (i) signalling events / postsynaptic modulation of chemical synaptic transmission / calcium channel regulator activity / regulation of synaptic transmission, glutamatergic / sensory perception of pain / modulation of chemical synaptic transmission ...Class C/3 (Metabotropic glutamate/pheromone receptors) / group II metabotropic glutamate receptor activity / G protein-coupled glutamate receptor signaling pathway / astrocyte projection / G alpha (i) signalling events / postsynaptic modulation of chemical synaptic transmission / calcium channel regulator activity / regulation of synaptic transmission, glutamatergic / sensory perception of pain / modulation of chemical synaptic transmission / presynaptic membrane / gene expression / scaffold protein binding / postsynapse / postsynaptic membrane / dendritic spine / postsynaptic density / neuron projection / axon / glutamatergic synapse / plasma membrane 類似検索 - 分子機能 | ||||||||||||
| 生物種 |  | ||||||||||||
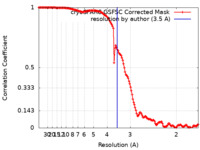
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.5 Å | ||||||||||||
 データ登録者 データ登録者 | Strauss A / Levitz J | ||||||||||||
| 資金援助 |  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: bioRxiv / 年: 2023 ジャーナル: bioRxiv / 年: 2023タイトル: Structural basis of allosteric modulation of metabotropic glutamate receptor activation and desensitization. 要旨: The metabotropic glutamate receptors (mGluRs) are neuromodulatory family C G protein coupled receptors which assemble as dimers and allosterically couple extracellular ligand binding domains (LBDs) ...The metabotropic glutamate receptors (mGluRs) are neuromodulatory family C G protein coupled receptors which assemble as dimers and allosterically couple extracellular ligand binding domains (LBDs) to transmembrane domains (TMDs) to drive intracellular signaling. Pharmacologically, mGluRs can be targeted either at the LBDs by glutamate and synthetic orthosteric compounds or at the TMDs by allosteric modulators. Despite the potential of allosteric TMD-targeting compounds as therapeutics, an understanding of the functional and structural basis of their effects on mGluRs is limited. Here we use a battery of approaches to dissect the distinct functional and structural effects of orthosteric versus allosteric ligands. We find using electrophysiological and live cell imaging assays that both agonists and positive allosteric modulators (PAMs) can drive activation and desensitization of mGluRs. The effects of PAMs are pleiotropic, including both the ability to boost the maximal response to orthosteric agonists and to serve independently as desensitization-biased agonists across mGluR subtypes. Conformational sensors reveal PAM-driven inter-subunit re-arrangements at both the LBD and TMD. Motivated by this, we determine cryo-electron microscopy structures of mGluR3 in the presence of either an agonist or antagonist alone or in combination with a PAM. These structures reveal PAM-driven re-shaping of intra- and inter-subunit conformations and provide evidence for a rolling TMD dimer interface activation pathway that controls G protein and beta-arrestin coupling. HIGHLIGHTS: -Agonists and PAMs drive mGluR activation, desensitization, and endocytosis-PAMs are desensitization-biased and synergistic with agonists-Four combinatorial ligand conditions reveal an ...HIGHLIGHTS: -Agonists and PAMs drive mGluR activation, desensitization, and endocytosis-PAMs are desensitization-biased and synergistic with agonists-Four combinatorial ligand conditions reveal an ensemble of full-length mGluR structures with novel interfaces-Activation and desensitization involve rolling TMD interfaces which are re-shaped by PAM. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_41568.map.gz emd_41568.map.gz | 137.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-41568-v30.xml emd-41568-v30.xml emd-41568.xml emd-41568.xml | 34.4 KB 34.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_41568_fsc.xml emd_41568_fsc.xml | 13.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_41568.png emd_41568.png | 50 KB | ||
| Filedesc metadata |  emd-41568.cif.gz emd-41568.cif.gz | 6.8 KB | ||
| その他 |  emd_41568_additional_1.map.gz emd_41568_additional_1.map.gz emd_41568_additional_2.map.gz emd_41568_additional_2.map.gz emd_41568_additional_3.map.gz emd_41568_additional_3.map.gz emd_41568_additional_4.map.gz emd_41568_additional_4.map.gz emd_41568_additional_5.map.gz emd_41568_additional_5.map.gz emd_41568_additional_6.map.gz emd_41568_additional_6.map.gz emd_41568_additional_7.map.gz emd_41568_additional_7.map.gz emd_41568_additional_8.map.gz emd_41568_additional_8.map.gz emd_41568_half_map_1.map.gz emd_41568_half_map_1.map.gz emd_41568_half_map_2.map.gz emd_41568_half_map_2.map.gz | 254.7 MB 204.9 MB 254.7 MB 254.7 MB 131 MB 254.8 MB 254.7 MB 132.7 MB 254.4 MB 254.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-41568 http://ftp.pdbj.org/pub/emdb/structures/EMD-41568 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41568 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41568 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_41568_validation.pdf.gz emd_41568_validation.pdf.gz | 852.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_41568_full_validation.pdf.gz emd_41568_full_validation.pdf.gz | 852.4 KB | 表示 | |
| XML形式データ |  emd_41568_validation.xml.gz emd_41568_validation.xml.gz | 23 KB | 表示 | |
| CIF形式データ |  emd_41568_validation.cif.gz emd_41568_validation.cif.gz | 29.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41568 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41568 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41568 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41568 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_41568.map.gz / 形式: CCP4 / 大きさ: 274.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_41568.map.gz / 形式: CCP4 / 大きさ: 274.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Full length map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.852 Å | ||||||||||||||||||||||||||||||||||||
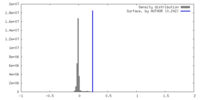
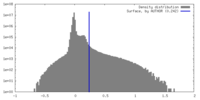
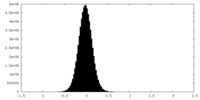
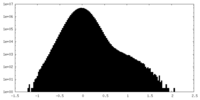
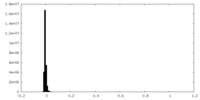
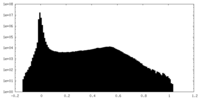
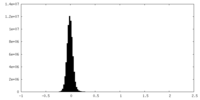
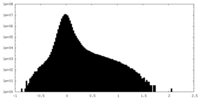
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
+追加マップ: Half map A for locally refined map of the extracellular domain
+追加マップ: Half map A for locally refined map of the extracellular domain
+追加マップ: Half map B for local map of the...
+追加マップ: Half map A for locally refined map of the extracellular domain
+追加マップ: Locally refined map of the extracellular domain
+追加マップ: Half map A for local map of the...
+追加マップ: Half map A for local map of the...
+追加マップ: Local map of the transmembrane domain following particle subtraction
+ハーフマップ: Half map B for full length map
+ハーフマップ: Half map A for full length map
- 試料の構成要素
試料の構成要素
-全体 : Metabotropic Glutamate Receptor 3 dimer
| 全体 | 名称: Metabotropic Glutamate Receptor 3 dimer |
|---|---|
| 要素 |
|
-超分子 #1: Metabotropic Glutamate Receptor 3 dimer
| 超分子 | 名称: Metabotropic Glutamate Receptor 3 dimer / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 260 KDa |
-分子 #1: Metabotropic glutamate receptor 3
| 分子 | 名称: Metabotropic glutamate receptor 3 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 103.161555 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MKMLTRLQIL MLALFSKGFL LSLGDHNFMR REIKIEGDLV LGGLFPINEK GTGTEECGRI NEDRGIQRLE AMLFAIDEIN KDNYLLPGV KLGVHILDTC SRDTYALEQS LEFVRASLTK VDEAEYMCPD GSYAIQENIP LLIAGVIGGS YSSVSIQVAN L LRLFQIPQ ...文字列: MKMLTRLQIL MLALFSKGFL LSLGDHNFMR REIKIEGDLV LGGLFPINEK GTGTEECGRI NEDRGIQRLE AMLFAIDEIN KDNYLLPGV KLGVHILDTC SRDTYALEQS LEFVRASLTK VDEAEYMCPD GSYAIQENIP LLIAGVIGGS YSSVSIQVAN L LRLFQIPQ ISYASTSAKL SDKSRYDYFA RTVPPDFYQA KAMAEILRFF NWTYVSTVAS EGDYGETGIE AFEQEARLRN IC IATAEKV GRSNIRKSYD SVIRELLQKP NARVVVLFMR SDDSRELIAA ANRVNASFTW VASDGWGAQE SIVKGSEHVA YGA ITLELA SHPVRQFDRY FQSLNPYNNH RNPWFRDFWE QKFQCSLQNK RNHRQVCDKH LAIDSSNYEQ ESKIMFVVNA VYAM AHALH KMQRTLCPNT TKLCDAMKIL DGKKLYKEYL LKINFTAPFN PNKGADSIVK FDTFGDGMGR YNVFNLQQTG GKYSY LKVG HWAETLSLDV DSIHWSRNSV PTSQCSDPCA PNEMKNMQPG DVCCWICIPC EPYEYLVDEF TCMDCGPGQW PTADLS GCY NLPEDYIKWE DAWAIGPVTI ACLGFLCTCI VITVFIKHNN TPLVKASGRE LCYILLFGVS LSYCMTFFFI AKPSPVI CA LRRLGLGTSF AICYSALLTK TNCIARIFDG VKNGAQRPKF ISPSSQVFIC LGLILVQIVM VSVWLILETP GTRRYTLP E KRETVILKCN VKDSSMLISL TYDVVLVILC TVYAFKTRKC PENFNEAKFI GFTMYTTCII WLAFLPIFYV TSSDYRVQT TTMCISVSLS GFVVLGCLFA PKVHIVLFQP QKNVVTHRLH LNRFSVSGTA TTYSQSSAST YVPTVCNGRE VLDSTTSSLA GLVPRGSAA AKSAWSHPQF EKGGGSGGGS GGGSWSHPQF EK UniProtKB: Metabotropic glutamate receptor 3 |
-分子 #2: (1S,4R,5R,6S)-4-amino-2-oxabicyclo[3.1.0]hexane-4,6-dicarboxylic acid
| 分子 | 名称: (1S,4R,5R,6S)-4-amino-2-oxabicyclo[3.1.0]hexane-4,6-dicarboxylic acid タイプ: ligand / ID: 2 / コピー数: 2 / 式: JIX |
|---|---|
| 分子量 | 理論値: 187.15 Da |
-分子 #3: CALCIUM ION
| 分子 | 名称: CALCIUM ION / タイプ: ligand / ID: 3 / コピー数: 4 / 式: CA |
|---|---|
| 分子量 | 理論値: 40.078 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製 #1
試料調製 #1
| Preparation ID | 1 |
|---|---|
| 濃度 | 4.5 mg/mL |
| 緩衝液 | pH: 7.5 |
| グリッド | モデル: UltrAuFoil R1.2/1.3 / 材質: GOLD / 支持フィルム - Film type ID: 1 / 支持フィルム - 材質: GOLD / 支持フィルム - トポロジー: HOLEY |
| 凍結 | 凍結剤: ETHANE / 装置: FEI VITROBOT MARK IV |
- 試料調製 #2
試料調製 #2
| Preparation ID | 2 |
|---|---|
| 緩衝液 | pH: 7.5 |
| グリッド | モデル: UltrAuFoil R1.2/1.3 / 材質: GOLD / 支持フィルム - Film type ID: 1 / 支持フィルム - 材質: GOLD / 支持フィルム - トポロジー: HOLEY |
| 凍結 | 凍結剤: ETHANE / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 検出モード: COUNTING / 平均電子線量: 58.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.0 µm 最小 デフォーカス(公称値): 0.7000000000000001 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL |
|---|---|
| 得られたモデル |  PDB-8tr2: |
 ムービー
ムービー コントローラー
コントローラー
















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)