+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-4063 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Single particle reconstruction of slow bee paralysis virus virion at pH 5.5 | |||||||||
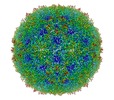
 マップデータ マップデータ | single particle reconstruction of SBPV virion at low pH | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | icosahedral virus / honebee virus / iflavirus / virus | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報host cell membrane / viral capsid / RNA helicase activity / viral RNA genome replication / cysteine-type endopeptidase activity / RNA-dependent RNA polymerase activity / DNA-templated transcription / structural molecule activity / proteolysis / RNA binding ...host cell membrane / viral capsid / RNA helicase activity / viral RNA genome replication / cysteine-type endopeptidase activity / RNA-dependent RNA polymerase activity / DNA-templated transcription / structural molecule activity / proteolysis / RNA binding / ATP binding / membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Slow bee paralysis virus (ウイルス) Slow bee paralysis virus (ウイルス) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.42 Å | |||||||||
 データ登録者 データ登録者 | Kalynych S / Fuzik T / Plevka P | |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2017 ジャーナル: Proc Natl Acad Sci U S A / 年: 2017タイトル: Cryo-EM study of slow bee paralysis virus at low pH reveals iflavirus genome release mechanism. 著者: Sergei Kalynych / Tibor Füzik / Antonín Přidal / Joachim de Miranda / Pavel Plevka /   要旨: Viruses from the family Iflaviridae are insect pathogens. Many of them, including slow bee paralysis virus (SBPV), cause lethal diseases in honeybees and bumblebees, resulting in agricultural losses. ...Viruses from the family Iflaviridae are insect pathogens. Many of them, including slow bee paralysis virus (SBPV), cause lethal diseases in honeybees and bumblebees, resulting in agricultural losses. Iflaviruses have nonenveloped icosahedral virions containing single-stranded RNA genomes. However, their genome release mechanism is unknown. Here, we show that low pH promotes SBPV genome release, indicating that the virus may use endosomes to enter host cells. We used cryo-EM to study a heterogeneous population of SBPV virions at pH 5.5. We determined the structures of SBPV particles before and after genome release to resolutions of 3.3 and 3.4 Å, respectively. The capsids of SBPV virions in low pH are not expanded. Thus, SBPV does not appear to form "altered" particles with pores in their capsids before genome release, as is the case in many related picornaviruses. The egress of the genome from SBPV virions is associated with a loss of interpentamer contacts mediated by N-terminal arms of VP2 capsid proteins, which result in the expansion of the capsid. Pores that are 7 Å in diameter form around icosahedral threefold symmetry axes. We speculate that they serve as channels for the genome release. Our findings provide an atomic-level characterization of the genome release mechanism of iflaviruses. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_4063.map.gz emd_4063.map.gz | 28.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-4063-v30.xml emd-4063-v30.xml emd-4063.xml emd-4063.xml | 15 KB 15 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_4063_fsc.xml emd_4063_fsc.xml | 21 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_4063.png emd_4063.png | 293.9 KB | ||
| Filedesc metadata |  emd-4063.cif.gz emd-4063.cif.gz | 6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-4063 http://ftp.pdbj.org/pub/emdb/structures/EMD-4063 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4063 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4063 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_4063_validation.pdf.gz emd_4063_validation.pdf.gz | 623.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_4063_full_validation.pdf.gz emd_4063_full_validation.pdf.gz | 623.4 KB | 表示 | |
| XML形式データ |  emd_4063_validation.xml.gz emd_4063_validation.xml.gz | 15.8 KB | 表示 | |
| CIF形式データ |  emd_4063_validation.cif.gz emd_4063_validation.cif.gz | 21.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4063 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4063 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4063 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4063 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_4063.map.gz / 形式: CCP4 / 大きさ: 209.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_4063.map.gz / 形式: CCP4 / 大きさ: 209.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | single particle reconstruction of SBPV virion at low pH | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.07 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Slow bee paralysis virus
| 全体 | 名称:  Slow bee paralysis virus (ウイルス) Slow bee paralysis virus (ウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Slow bee paralysis virus
| 超分子 | 名称: Slow bee paralysis virus / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all / NCBI-ID: 458132 / 生物種: Slow bee paralysis virus / ウイルスタイプ: VIRION / ウイルス・単離状態: SPECIES / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:  |
| ウイルス殻 | Shell ID: 1 / 名称: icosahedral |
-分子 #1: VP1
| 分子 | 名称: VP1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Slow bee paralysis virus (ウイルス) Slow bee paralysis virus (ウイルス) |
| 分子量 | 理論値: 30.307211 KDa |
| 配列 | 文字列: MERTFTPNVM QPTPLLPTTN DGRVTFGEAF NDLKDLARRY QLYWEGTILE GNLRAIRRNS ALVQLPLYPH GLRIQPDVNN PIWNIMRDG HIPVISSGFR YFRGGLRLRI VVEGLNSCVW VQHHPDRPSI FSRPIIGRYI AAKDAYRNHA YAAYVQNMSV N RTIEVEVP ...文字列: MERTFTPNVM QPTPLLPTTN DGRVTFGEAF NDLKDLARRY QLYWEGTILE GNLRAIRRNS ALVQLPLYPH GLRIQPDVNN PIWNIMRDG HIPVISSGFR YFRGGLRLRI VVEGLNSCVW VQHHPDRPSI FSRPIIGRYI AAKDAYRNHA YAAYVQNMSV N RTIEVEVP FYQPGLYGML NASDNNTANS FDRLRFTGLG DLLIGIEGEQ PIPKEGIEIS VYYSIADDFS FNIFCGFPPM VY CDETYSA ATPDLAQYFE DEVTIAQPE UniProtKB: Genome polyprotein |
-分子 #2: VP2
| 分子 | 名称: VP2 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Slow bee paralysis virus (ウイルス) Slow bee paralysis virus (ウイルス) |
| 分子量 | 理論値: 29.824205 KDa |
| 配列 | 文字列: MDRPEGSEER TVQTSNVVLG ETNIESQDIA SKEYSPTWDR LASSEVSDEY PMLTDRWLFW KSVKWEVNDS AFGKMLVQEK FPQSWVQMD VNVNNIPRYT NIPNFIPFNI HQYMRADFEV KIYVNPNDFV SGWLIMAFLY QGSEMFDYKL RRNPAALMQM P HVLVNVGA ...文字列: MDRPEGSEER TVQTSNVVLG ETNIESQDIA SKEYSPTWDR LASSEVSDEY PMLTDRWLFW KSVKWEVNDS AFGKMLVQEK FPQSWVQMD VNVNNIPRYT NIPNFIPFNI HQYMRADFEV KIYVNPNDFV SGWLIMAFLY QGSEMFDYKL RRNPAALMQM P HVLVNVGA ANEATLKIPY RYVRPFMRCK DILRGDNLIT GVTEPLNMGV LFVEVLIPFR TSAASSAPKS LDVSLFVKMT NA KFTGMVD GSIALLSKPI ALPE UniProtKB: Genome polyprotein |
-分子 #3: VP3
| 分子 | 名称: VP3 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Slow bee paralysis virus (ウイルス) Slow bee paralysis virus (ウイルス) |
| 分子量 | 理論値: 47.62391 KDa |
| 配列 | 文字列: DNPPDPTPAK FFVPIPSHSW AHGTNTSEPT NTLRLDGGVV GVGRSDDIGT SDTAISGIIG VYGLLKPFDW NANDTGRNVG GHLLWSMPV HPQVDKDQVI QVMTQSKLTQ YYLPPISVVS SLYAYTRGSI KYKFLFGNNP RHNARLLVAY IPGISSDNRL T LERARNSA ...文字列: DNPPDPTPAK FFVPIPSHSW AHGTNTSEPT NTLRLDGGVV GVGRSDDIGT SDTAISGIIG VYGLLKPFDW NANDTGRNVG GHLLWSMPV HPQVDKDQVI QVMTQSKLTQ YYLPPISVVS SLYAYTRGSI KYKFLFGNNP RHNARLLVAY IPGISSDNRL T LERARNSA HVVFSLNEVS EFVFTVPYIT DTMWWPRKYG GPQAAGEFVA PSYICMFILN PLVAMESVPS IVTIVPMIAA GD DFEVAVP AQPAVGLSRN IDVIYPKDSI ISFKSGYFPV YVGSWHSFFD STKAILRYGA VSDHIAQLGN IPANVNRKAF WIV VGDTIK FKTKLDKING TEWFIPEGEY TLGYGVVWRD GAYAYMVPYP LTPLGEKIAQ YTASLLASNT AISQIRPYIP DYIV DSAAS KDNILWSPIE DRLRAQTEWV MAEPE UniProtKB: Genome polyprotein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.8 mg/mL |
|---|---|
| 緩衝液 | pH: 5.5 / 詳細: 30 mM Sodium Acetate 50 mM Sodium Chloride |
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 300 |
| 凍結 | 凍結剤: ETHANE / 装置: FEI VITROBOT MARK IV / 詳細: blot force 0, blot time 2 s.. |
| 詳細 | sample was purifed froim natural source and dialyzed overnight into sodium acetate buffer |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 検出モード: INTEGRATING / デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / デジタル化 - 画像ごとのフレーム数: 1-7 / 撮影したグリッド数: 1 / 実像数: 5325 / 平均露光時間: 0.5 sec. / 平均電子線量: 21.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: OTHER / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 75000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: RECIPROCAL / プロトコル: OTHER / 温度因子: 49.7 |
|---|---|
| 得られたモデル |  PDB-5lk7: |
 ムービー
ムービー コントローラー
コントローラー