+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-4038 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
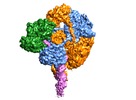
| タイトル | Cryo-EM structure of RecBCD+DNA complex revealing activated nuclease domain | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Helicase / Nuclease / SH3 / Homologous Recombination / hydrolase | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報exodeoxyribonuclease V / exodeoxyribonuclease V activity / exodeoxyribonuclease V complex / clearance of foreign intracellular DNA / DNA 5'-3' helicase / DNA translocase activity / DNA 3'-5' helicase / recombinational repair / single-stranded DNA helicase activity / 3'-5' DNA helicase activity ...exodeoxyribonuclease V / exodeoxyribonuclease V activity / exodeoxyribonuclease V complex / clearance of foreign intracellular DNA / DNA 5'-3' helicase / DNA translocase activity / DNA 3'-5' helicase / recombinational repair / single-stranded DNA helicase activity / 3'-5' DNA helicase activity / ATP-dependent activity, acting on DNA / DNA helicase activity / isomerase activity / helicase activity / DNA endonuclease activity / double-strand break repair via homologous recombination / response to radiation / 5'-3' DNA helicase activity / DNA recombination / DNA damage response / magnesium ion binding / ATP hydrolysis activity / DNA binding / ATP binding / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |    Endothia gyrosa (菌類) Endothia gyrosa (菌類) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.83 Å | |||||||||
 データ登録者 データ登録者 | Wilkinson M / Chaban Y | |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2016 ジャーナル: Elife / 年: 2016タイトル: Mechanism for nuclease regulation in RecBCD. 著者: Martin Wilkinson / Yuriy Chaban / Dale B Wigley /  要旨: In bacterial cells, processing of double-stranded DNA breaks for repair by homologous recombination is catalysed by AddAB, AdnAB or RecBCD-type helicase-nucleases. These enzyme complexes are highly ...In bacterial cells, processing of double-stranded DNA breaks for repair by homologous recombination is catalysed by AddAB, AdnAB or RecBCD-type helicase-nucleases. These enzyme complexes are highly processive, duplex unwinding and degrading machines that require tight regulation. Here, we report the structure of E.coli RecBCD, determined by cryoEM at 3.8 Å resolution, with a DNA substrate that reveals how the nuclease activity of the complex is activated once unwinding progresses. Extension of the 5'-tail of the unwound duplex induces a large conformational change in the RecD subunit, that is transferred through the RecC subunit to activate the nuclease domain of the RecB subunit. The process involves a SH3 domain that binds to a region of the RecB subunit in a binding mode that is distinct from others observed previously in SH3 domains and, to our knowledge, this is the first example of peptide-binding of an SH3 domain in a bacterial system. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_4038.map.gz emd_4038.map.gz | 20.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-4038-v30.xml emd-4038-v30.xml emd-4038.xml emd-4038.xml | 19.2 KB 19.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_4038.png emd_4038.png | 143 KB | ||
| Filedesc metadata |  emd-4038.cif.gz emd-4038.cif.gz | 8.3 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-4038 http://ftp.pdbj.org/pub/emdb/structures/EMD-4038 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4038 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4038 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_4038_validation.pdf.gz emd_4038_validation.pdf.gz | 276.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_4038_full_validation.pdf.gz emd_4038_full_validation.pdf.gz | 275.2 KB | 表示 | |
| XML形式データ |  emd_4038_validation.xml.gz emd_4038_validation.xml.gz | 5.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4038 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4038 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4038 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4038 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_4038.map.gz / 形式: CCP4 / 大きさ: 22.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_4038.map.gz / 形式: CCP4 / 大きさ: 22.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.34 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Ternary complex of RecBCD with forked DNA substrate and ADPNP
| 全体 | 名称: Ternary complex of RecBCD with forked DNA substrate and ADPNP |
|---|---|
| 要素 |
|
-超分子 #1: Ternary complex of RecBCD with forked DNA substrate and ADPNP
| 超分子 | 名称: Ternary complex of RecBCD with forked DNA substrate and ADPNP タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 350 KDa |
-分子 #1: RecBCD enzyme subunit RecB,RecBCD enzyme subunit RecB,RecBCD enzy...
| 分子 | 名称: RecBCD enzyme subunit RecB,RecBCD enzyme subunit RecB,RecBCD enzyme subunit RecB タイプ: protein_or_peptide / ID: 1 詳細: Residues 913-932 modelled as poly-Alanine in model to reflect uncertainty of the exact position of this loop due to weak density. コピー数: 1 / 光学異性体: LEVO / EC番号: exodeoxyribonuclease V |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 133.717391 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GMSDVAETLD PLRLPLQGER LIEASAGTGK TFTIAALYLR LLLGLGGSAA FPRPLTVEEL LVVTFTEAAT AELRGRIRSN IHELRIACL RETTDNPLYE RLLEEIDDKA QAAQWLLLAE RQMDEAAVFT IHGFCQRMLN LNAFESGMLF EQQLIEDESL L RYQACADF ...文字列: GMSDVAETLD PLRLPLQGER LIEASAGTGK TFTIAALYLR LLLGLGGSAA FPRPLTVEEL LVVTFTEAAT AELRGRIRSN IHELRIACL RETTDNPLYE RLLEEIDDKA QAAQWLLLAE RQMDEAAVFT IHGFCQRMLN LNAFESGMLF EQQLIEDESL L RYQACADF WRRHCYPLPR EIAQVVFETW KGPQALLRDI NRYLQGEAPV IKAPPPDDET LASRHAQIVA RIDTVKQQWR DA VGELDAL IESSGIDRRK FNRSNQAKWI DKISAWAEEE TNSYQLPESL EKFSQRFLED RTKAGGETPR HPLFEAIDQL LAE PLSIRD LVITRALAEI RETVAREKRR RGELGFDDML SRLDSALRSE SGEVLAAAIR TRFPVAMIDE FQDTDPQQYR IFRR IWHHQ PETALLLIGD PKQAIYAFRG ADIFTYMKAR SEVHAHYTLD TNWRSAPGMV NSVNKLFSQT DDAFMFREIP FIPVK SAGK NQALRFVFKG ETQPAMKMWL MEGESCGVGD YQSTMAQVCA AQIRDWLQAG QRGEALLMNG DDARPVRASD ISVLVR SRQ EAAQVRDALT LLEIPSVYLS NRDSVFETLE AQEMLWLLQA VMTPERENTL RSALATSMMG LNALDIETLN NDEHAWD VV VEEFDGYRQI WRKRGVMPML RALMSARNIA ENLLATAGGE RRLTDILHIS ELLQEAGTQL ESEHALVRWL SQHILEPD S NASSQQMRLE SDKHLVQIVT IHKSKGLEYP LVWLPFITNF RVQEQAFYHD RHSFEAVLDL NAAPESVDLA EAERLAEDL RLLYVALTRS VWHCSLGVAP LVRRRGDKKG DTDVHQSALG RLLQKGEPQD AAGLRTCIEA LCDDDIAWQT AQTGDNQPWQ VNDVSTAEL NAKTLQRLPG DNWRVTSYSG LQQR(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) PTLTPHQFP RGASPGTFLH SLFEDLDFTQ PVDPNWVREK LELGGFESQW EPVLTEWITA VLQAPLNETG VSLSQLSARN K QVEMEFYL PISEPLIASQ LDTLIRQFDP LSAGCPPLEF MQVRGMLKGF IDLVFRHEGR YYLLAYKSNW LGEDSSAYTQ QA MAAAMQA HRYDLQYQLY TLALHRYLRH RIADYDYEHH FGGVIYLFLR GVDKEHPQQG IYTTRPNAGL IALMDEMFAG MTL EEA UniProtKB: RecBCD enzyme subunit RecB, RecBCD enzyme subunit RecB |
-分子 #2: RecBCD enzyme subunit RecC
| 分子 | 名称: RecBCD enzyme subunit RecC / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO / EC番号: exodeoxyribonuclease V |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 128.974102 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MLRVYHSNRL DVLEALMEFI VERERLDDPF EPEMILVQST GMAQWLQMTL SQKFGIAANI DFPLPASFIW DMFVRVLPEI PKESAFNKQ SMSWKLMTLL PQLLEREDFT LLRHYLTDDS DKRKLFQLSS KAADLFDQYL VYRPDWLAQW ETGHLVEGLG E AQAWQAPL ...文字列: MLRVYHSNRL DVLEALMEFI VERERLDDPF EPEMILVQST GMAQWLQMTL SQKFGIAANI DFPLPASFIW DMFVRVLPEI PKESAFNKQ SMSWKLMTLL PQLLEREDFT LLRHYLTDDS DKRKLFQLSS KAADLFDQYL VYRPDWLAQW ETGHLVEGLG E AQAWQAPL WKALVEYTHQ LGQPRWHRAN LYQRFIETLE SATTCPPGLP SRVFICGISA LPPVYLQALQ ALGKHIEIHL LF TNPCRYY WGDIKDPAYL AKLLTRQRRH SFEDRELPLF RDSENAGQLF NSDGEQDVGN PLLASWGKLG RDYIYLLSDL ESS QELDAF VDVTPDNLLH NIQSDILELE NRAVAGVNIE EFSRSDNKRP LDPLDSSITF HVCHSPQREV EVLHDRLLAM LEED PTLTP RDIIVMVADI DSYSPFIQAV FGSAPADRYL PYAISDRRAR QSHPVLEAFI SLLSLPDSRF VSEDVLALLD VPVLA ARFD ITEEGLRYLR QWVNESGIRW GIDDDNVREL ELPATGQHTW RFGLTRMLLG YAMESAQGEW QSVLPYDESS GLIAEL VGH LASLLMQLNI WRRGLAQERP LEEWLPVCRD MLNAFFLPDA ETEAAMTLIE QQWQAIIAEG LGAQYGDAVP LSLLRDE LA QRLDQERISQ RFLAGPVNIC TLMPMRSIPF KVVCLLGMND GVYPRQLAPL GFDLMSQKPK RGDRSRRDDD RYLFLEAL I SAQQKLYISY IGRSIQDNSE RFPSVLVQEL IDYIGQSHYL PGDEALNCDE SEARVKAHLT CLHTRMPFDP QNYQPGERQ SYAREWLPAA SQAGKAHSEF VQPLPFTLPE TVPLETLQRF WAHPVRAFFQ MRLQVNFRTE DSEIPDTEPF ILEGLSRYQI NQQLLNALV EQDDAERLFR RFRAAGDLPY GAFGEIFWET QCQEMQQLAD RVIACRQPGQ SMEIDLACNG VQITGWLPQV Q PDGLLRWR PSLLSVAQGM QLWLEHLVYC ASGGNGESRL FLRKDGEWRF PPLAAEQALH YLSQLIEGYR EGMSAPLLVL PE SGGAWLK TCYDAQNDAM LDDDSTLQKA RTKFLQAYEG NMMVRGEGDD IWYQRLWRQL TPETMEAIVE QSQRFLLPLF RFN QS UniProtKB: RecBCD enzyme subunit RecC |
-分子 #3: RecBCD enzyme subunit RecD
| 分子 | 名称: RecBCD enzyme subunit RecD / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO / EC番号: exodeoxyribonuclease V |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 67.047422 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGKLQKQLLE AVEHKQLRPL DVQFALTVAG DEHPAVTLAA ALLSHDAGEG HVCLPLSRLE NNEASHPLLA TCVSEIGELQ NWEECLLAS QAVSRGDEPT PMILCGDRLY LNRMWCNERT VARFFNEVNH AIEVDEALLA QTLDKLFPVS DEINWQKVAA A VALTRRIS ...文字列: MGKLQKQLLE AVEHKQLRPL DVQFALTVAG DEHPAVTLAA ALLSHDAGEG HVCLPLSRLE NNEASHPLLA TCVSEIGELQ NWEECLLAS QAVSRGDEPT PMILCGDRLY LNRMWCNERT VARFFNEVNH AIEVDEALLA QTLDKLFPVS DEINWQKVAA A VALTRRIS VISGGPGTGK TTTVAKLLAA LIQMADGERC RIRLAAPTGK AAARLTESLG KALRQLPLTD EQKKRIPEDA ST LHRLLGA QPGSQRLRHH AGNPLHLDVL VVDEASMIDL PMMSRLIDAL PDHARVIFLG DRDQLASVEA GAVLGDICAY ANA GFTAER ARQLSRLTGT HVPAGTGTEA ASLRDSLCLL QKSYRFGSDS GIGQLAAAIN RGDKTAVKTV FQQDFTDIEK RLLQ SGEDY IAMLEEALAG YGRYLDLLQA RAEPDLIIQA FNEYQLLCAL REGPFGVAGL NERIEQFMQQ KRKIHRHPHS RWYEG RPVM IARNDSALGL FNGDIGIALD RGQGTRVWFA MPDGNIKSVQ PSRLPEHETT WAMTVHKSQG SEFDHAALIL PSQRTP VVT RELVYTAVTR ARRRLSLYAD ERILSAAIAT RTERRSGLAA LFSSRE UniProtKB: RecBCD enzyme subunit RecD |
-分子 #4: Fork-Hairpin DNA (70-MER)
| 分子 | 名称: Fork-Hairpin DNA (70-MER) / タイプ: dna / ID: 4 詳細: Hairpin=38-42 Duplex=13-37&43-67 5'Tail=1-12 3'Tail=68-70 コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  Endothia gyrosa (菌類) Endothia gyrosa (菌類) |
| 分子量 | 理論値: 21.440707 KDa |
| 配列 | 文字列: (DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT) (DT)(DT)(DT)(DC)(DT)(DA)(DA)(DT)(DG)(DC) (DG)(DA)(DG)(DC)(DA)(DC)(DT)(DG)(DC) (DT)(DA)(DC)(DA)(DG)(DC)(DA)(DT)(DT)(DT) (DC) (DC)(DC)(DA)(DT)(DG) ...文字列: (DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT) (DT)(DT)(DT)(DC)(DT)(DA)(DA)(DT)(DG)(DC) (DG)(DA)(DG)(DC)(DA)(DC)(DT)(DG)(DC) (DT)(DA)(DC)(DA)(DG)(DC)(DA)(DT)(DT)(DT) (DC) (DC)(DC)(DA)(DT)(DG)(DC)(DT)(DG) (DT)(DA)(DG)(DC)(DA)(DG)(DT)(DG)(DC)(DT) (DC)(DG) (DC)(DA)(DT)(DT)(DA)(DG)(DA) (DT)(DT)(DT) |
-分子 #5: PHOSPHOAMINOPHOSPHONIC ACID-ADENYLATE ESTER
| 分子 | 名称: PHOSPHOAMINOPHOSPHONIC ACID-ADENYLATE ESTER / タイプ: ligand / ID: 5 / コピー数: 1 / 式: ANP |
|---|---|
| 分子量 | 理論値: 506.196 Da |
| Chemical component information |  ChemComp-ANP: |
-分子 #6: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 6 / コピー数: 1 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 5 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| |||||||||||||||
| グリッド | モデル: C-flat / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY 詳細: Glow discharge was used to thin the carbon film, but allowed to dissipate prior to use. Detergent or amphipols treatment was used to render carbon hydrophilic. See article for details. | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / 装置: FEI VITROBOT MARK III |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - 画像ごとのフレーム数: 1-30 / 撮影したグリッド数: 1 / 実像数: 1674 / 平均露光時間: 0.4 sec. / 平均電子線量: 1.2 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 最大 デフォーカス(補正後): 3.8000000000000003 µm 最小 デフォーカス(補正後): 0.5 µm / 倍率(補正後): 37313 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: PDB ENTRY PDBモデル - PDB ID: |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.83 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: RELION (ver. 1.4) / 使用した粒子像数: 74656 |
| 初期 角度割当 | タイプ: PROJECTION MATCHING / ソフトウェア - 名称: RELION (ver. 1.3) |
| 最終 角度割当 | タイプ: PROJECTION MATCHING / ソフトウェア - 名称: RELION (ver. 1.4) |
 ムービー
ムービー コントローラー
コントローラー