+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-3761 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
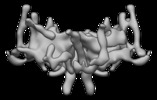
| タイトル | Cryo-EM structure of the TOM core complex from Neurospora crassa | |||||||||
 マップデータ マップデータ | TOM core complex consisting of Tom40, Tom22, Tom5, Tom6 and Tom7 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | TOM-Complex / Protein Import / Mitochondria / Cryo-EM / PROTEIN TRANSPORT | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報mitochondrial outer membrane translocase complex / protein import into mitochondrial matrix / porin activity / pore complex / protein transmembrane transporter activity 類似検索 - 分子機能 | |||||||||
| 生物種 |  Neurospora crassa (菌類) / Neurospora crassa (菌類) /  Neurospora crassa (strain ATCC 24698 / 74-OR23-1A / CBS 708.71 / DSM 1257 / FGSC 987) (菌類) Neurospora crassa (strain ATCC 24698 / 74-OR23-1A / CBS 708.71 / DSM 1257 / FGSC 987) (菌類) | |||||||||
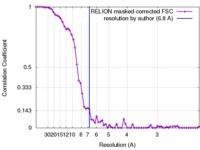
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 6.8 Å | |||||||||
 データ登録者 データ登録者 | Bausewein T / Mills DJ | |||||||||
| 資金援助 |  ドイツ, 1件 ドイツ, 1件
| |||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2017 ジャーナル: Cell / 年: 2017タイトル: Cryo-EM Structure of the TOM Core Complex from Neurospora crassa. 著者: Thomas Bausewein / Deryck J Mills / Julian D Langer / Beate Nitschke / Stephan Nussberger / Werner Kühlbrandt /  要旨: The TOM complex is the main entry gate for protein precursors from the cytosol into mitochondria. We have determined the structure of the TOM core complex by cryoelectron microscopy (cryo-EM). The ...The TOM complex is the main entry gate for protein precursors from the cytosol into mitochondria. We have determined the structure of the TOM core complex by cryoelectron microscopy (cryo-EM). The complex is a 148 kDa symmetrical dimer of ten membrane protein subunits that create a shallow funnel on the cytoplasmic membrane surface. In the core of the dimer, the β-barrels of the Tom40 pore form two identical preprotein conduits. Each Tom40 pore is surrounded by the transmembrane segments of the α-helical subunits Tom5, Tom6, and Tom7. Tom22, the central preprotein receptor, connects the two Tom40 pores at the dimer interface. Our structure offers detailed insights into the molecular architecture of the mitochondrial preprotein import machinery. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_3761.map.gz emd_3761.map.gz | 21.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-3761-v30.xml emd-3761-v30.xml emd-3761.xml emd-3761.xml | 9.5 KB 9.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_3761_fsc.xml emd_3761_fsc.xml | 6.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_3761.png emd_3761.png | 73.3 KB | ||
| Filedesc metadata |  emd-3761.cif.gz emd-3761.cif.gz | 4.8 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-3761 http://ftp.pdbj.org/pub/emdb/structures/EMD-3761 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3761 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3761 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_3761_validation.pdf.gz emd_3761_validation.pdf.gz | 259.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_3761_full_validation.pdf.gz emd_3761_full_validation.pdf.gz | 259 KB | 表示 | |
| XML形式データ |  emd_3761_validation.xml.gz emd_3761_validation.xml.gz | 9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3761 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3761 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3761 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3761 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_3761.map.gz / 形式: CCP4 / 大きさ: 23 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_3761.map.gz / 形式: CCP4 / 大きさ: 23 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | TOM core complex consisting of Tom40, Tom22, Tom5, Tom6 and Tom7 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.12 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : TOM core complex consisting of Tom40, Tom22, Tom5, Tom6 and Tom7
| 全体 | 名称: TOM core complex consisting of Tom40, Tom22, Tom5, Tom6 and Tom7 |
|---|---|
| 要素 |
|
-超分子 #1: TOM core complex consisting of Tom40, Tom22, Tom5, Tom6 and Tom7
| 超分子 | 名称: TOM core complex consisting of Tom40, Tom22, Tom5, Tom6 and Tom7 タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Neurospora crassa (菌類) / 株: GR-107 Neurospora crassa (菌類) / 株: GR-107 |
-分子 #1: Mitochondrial import receptor subunit tom40
| 分子 | 名称: Mitochondrial import receptor subunit tom40 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Neurospora crassa (strain ATCC 24698 / 74-OR23-1A / CBS 708.71 / DSM 1257 / FGSC 987) (菌類) Neurospora crassa (strain ATCC 24698 / 74-OR23-1A / CBS 708.71 / DSM 1257 / FGSC 987) (菌類)株: ATCC 24698 / 74-OR23-1A / CBS 708.71 / DSM 1257 / FGSC 987 |
| 分子量 | 理論値: 38.184797 KDa |
| 配列 | 文字列: MASFSTESPL AMLRDNAIYS SLSDAFNAFQ ERRKQFGLSN PGTIETIARE VQRDTLLTNY MFSGLRADVT KAFSLAPLFQ VSHQFAMGE RLNPYAFAAL YGTNQIFAQG NLDNEGALST RFNYRWGDRT ITKTQFSIGG GQDMAQFEHE HLGDDFSASL K AINPSFLD ...文字列: MASFSTESPL AMLRDNAIYS SLSDAFNAFQ ERRKQFGLSN PGTIETIARE VQRDTLLTNY MFSGLRADVT KAFSLAPLFQ VSHQFAMGE RLNPYAFAAL YGTNQIFAQG NLDNEGALST RFNYRWGDRT ITKTQFSIGG GQDMAQFEHE HLGDDFSASL K AINPSFLD GGLTGIFVGD YLQAVTPRLG LGLQAVWQRQ GLTQGPDTAI SYFARYKAGD WVASAQLQAQ GALNTSFWKK LT DRVQAGV DMTLSVAPSQ SMMGGLTKEG ITTFGAKYDF RMSTFRAQID SKGKLSCLLE KRLGAAPVTL TFAADVDHVT QQA KLGMSV SIEASDVDLQ EQQEGAQSLN IPF UniProtKB: Mitochondrial import receptor subunit tom40 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL |
|---|---|
| 緩衝液 | pH: 7.2 |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL 3200FSC |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | プロトコル: FLEXIBLE FIT |
|---|---|
| 得られたモデル |  PDB-5o8o: |
 ムービー
ムービー コントローラー
コントローラー