+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
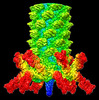
| タイトル | Cyanophage Pam3 baseplate proteins | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | baseplate / VIRUS / VIRAL PROTEIN | |||||||||
| 生物種 |  uncultured cyanophage (ファージ) uncultured cyanophage (ファージ) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.19 Å | |||||||||
 データ登録者 データ登録者 | Yang F / Jiang YL / Zhou CZ | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2023 ジャーナル: Proc Natl Acad Sci U S A / 年: 2023タイトル: Fine structure and assembly pattern of a minimal myophage Pam3. 著者: Feng Yang / Yong-Liang Jiang / Jun-Tao Zhang / Jie Zhu / Kang Du / Rong-Cheng Yu / Zi-Lu Wei / Wen-Wen Kong / Ning Cui / Wei-Fang Li / Yuxing Chen / Qiong Li / Cong-Zhao Zhou /  要旨: The myophage possesses a contractile tail that penetrates its host cell envelope. Except for investigations on the bacteriophage T4 with a rather complicated structure, the assembly pattern and tail ...The myophage possesses a contractile tail that penetrates its host cell envelope. Except for investigations on the bacteriophage T4 with a rather complicated structure, the assembly pattern and tail contraction mechanism of myophage remain largely unknown. Here, we present the fine structure of a freshwater cyanophage Pam3, which has an icosahedral capsid of ~680 Å in diameter, connected via a three-section neck to an 840-Å-long contractile tail, ending with a three-module baseplate composed of only six protein components. This simplified baseplate consists of a central hub-spike surrounded by six wedge heterotriplexes, to which twelve tail fibers are covalently attached via disulfide bonds in alternating upward and downward configurations. In vitro reduction assays revealed a putative redox-dependent mechanism of baseplate assembly and tail sheath contraction. These findings establish a minimal myophage that might become a user-friendly chassis phage in synthetic biology. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_33802.map.gz emd_33802.map.gz | 393.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-33802-v30.xml emd-33802-v30.xml emd-33802.xml emd-33802.xml | 19.6 KB 19.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_33802.png emd_33802.png | 184.3 KB | ||
| Filedesc metadata |  emd-33802.cif.gz emd-33802.cif.gz | 6.2 KB | ||
| その他 |  emd_33802_half_map_1.map.gz emd_33802_half_map_1.map.gz emd_33802_half_map_2.map.gz emd_33802_half_map_2.map.gz | 337.9 MB 337.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-33802 http://ftp.pdbj.org/pub/emdb/structures/EMD-33802 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33802 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33802 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_33802_validation.pdf.gz emd_33802_validation.pdf.gz | 1.1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_33802_full_validation.pdf.gz emd_33802_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  emd_33802_validation.xml.gz emd_33802_validation.xml.gz | 17.9 KB | 表示 | |
| CIF形式データ |  emd_33802_validation.cif.gz emd_33802_validation.cif.gz | 21.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33802 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33802 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33802 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33802 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_33802.map.gz / 形式: CCP4 / 大きさ: 421.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_33802.map.gz / 形式: CCP4 / 大きさ: 421.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.07 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_33802_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_33802_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : uncultured cyanophage
| 全体 | 名称:  uncultured cyanophage (ファージ) uncultured cyanophage (ファージ) |
|---|---|
| 要素 |
|
-超分子 #1: uncultured cyanophage
| 超分子 | 名称: uncultured cyanophage / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all / NCBI-ID: 215796 / 生物種: uncultured cyanophage / ウイルスタイプ: VIRION / ウイルス・単離状態: OTHER / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:  Pseudanabaena mucicola (バクテリア) Pseudanabaena mucicola (バクテリア) |
-分子 #1: Pam3 baseplate wedge gp22
| 分子 | 名称: Pam3 baseplate wedge gp22 / タイプ: protein_or_peptide / ID: 1 / コピー数: 12 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  uncultured cyanophage (ファージ) uncultured cyanophage (ファージ) |
| 分子量 | 理論値: 32.686787 KDa |
| 配列 | 文字列: MTYGVQPTGY VKKPLAVHLA EIEASMVDLF GPGVIQTEQS PLGQLNGLYA DLSYDLDERG EDLYQSFDPE QAEGSRLDIL ARYRLLSRR AGESDESFRR AITNVDRARI DLSDLSTALS AINGVSWSRV YVNEDATTDA DGIPPNTVSV AVIGGDDDEV A QLVRRYVV ...文字列: MTYGVQPTGY VKKPLAVHLA EIEASMVDLF GPGVIQTEQS PLGQLNGLYA DLSYDLDERG EDLYQSFDPE QAEGSRLDIL ARYRLLSRR AGESDESFRR AITNVDRARI DLSDLSTALS AINGVSWSRV YVNEDATTDA DGIPPNTVSV AVIGGDDDEV A QLVRRYVV PGVGMYGNTT IETTIGGFCR RIRVIRPVLI PTSVEIDVQS RPLKNGCPPP SVNAMAAGLY TELTGPDRPG NG EDGTVYL FRKIMERLYP NVEVVDVRLS QAPAAPTTPP LVMSFFQMMS FNADDILVEI VP |
-分子 #2: Pam3 baseplate wedge gp23
| 分子 | 名称: Pam3 baseplate wedge gp23 / タイプ: protein_or_peptide / ID: 2 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  uncultured cyanophage (ファージ) uncultured cyanophage (ファージ) |
| 分子量 | 理論値: 26.194811 KDa |
| 配列 | 文字列: MSNLCADPEA LVEARIDEVL TQYRESPYLL NLIRAYLSKL AETSMSYCDM VEKFDLDTAV GDQLTIIGRI LGFPRCHCVC DTIPVVGYD CGGSYAGSYQ LAGYCEPGSS WIHCSPYGNS ELCVDEDEIY RSLLKARRYQ MLGLYDIESL HEALQIVWGE D AMVAETKV ...文字列: MSNLCADPEA LVEARIDEVL TQYRESPYLL NLIRAYLSKL AETSMSYCDM VEKFDLDTAV GDQLTIIGRI LGFPRCHCVC DTIPVVGYD CGGSYAGSYQ LAGYCEPGSS WIHCSPYGNS ELCVDEDEIY RSLLKARRYQ MLGLYDIESL HEALQIVWGE D AMVAETKV GQVVVTPGRS LTATETRYLP IVFRALPIAP GIKGMIHIDQ GPIAGYGDGW AGYCGGDWLC PVDPHAYTCS |
-分子 #3: Pam3 sheath initiator gp21
| 分子 | 名称: Pam3 sheath initiator gp21 / タイプ: protein_or_peptide / ID: 3 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  uncultured cyanophage (ファージ) uncultured cyanophage (ファージ) |
| 分子量 | 理論値: 13.889669 KDa |
| 配列 | 文字列: MAAPSRIGLA LKKNEQGIAD IYLDASGNLS MVQNTEAVGQ HARQRLMTYL GEWFLNKNVG VPWLRDLLGK GYDPVLAEAV IKAEILSTD GVTEITSFSI RFDQGRRALE AFDIEVTTEY DEETVL |
-分子 #4: Pam3 tube initiator gp17
| 分子 | 名称: Pam3 tube initiator gp17 / タイプ: protein_or_peptide / ID: 4 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  uncultured cyanophage (ファージ) uncultured cyanophage (ファージ) |
| 分子量 | 理論値: 19.854342 KDa |
| 配列 | 文字列: MIIAFSSAIG PVPLTVVISE KHTSKVELTT NPIESGADVT DHAYVKGKEI ELEVADRNAA ATWAALVAFQ ESRVPFVLMT GLSMYRNMI ITEIDATRNA QHSKILKGTV RLREVKIVET GTAEDSSGKD GTDKNKSSNP SKDKAADAKT ADKANSGVNA G DKGGTTVA ...文字列: MIIAFSSAIG PVPLTVVISE KHTSKVELTT NPIESGADVT DHAYVKGKEI ELEVADRNAA ATWAALVAFQ ESRVPFVLMT GLSMYRNMI ITEIDATRNA QHSKILKGTV RLREVKIVET GTAEDSSGKD GTDKNKSSNP SKDKAADAKT ADKANSGVNA G DKGGTTVA APRAQSLLKG VFGGSSASGG AAP |
-分子 #5: Pam3 plug gp18
| 分子 | 名称: Pam3 plug gp18 / タイプ: protein_or_peptide / ID: 5 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  uncultured cyanophage (ファージ) uncultured cyanophage (ファージ) |
| 分子量 | 理論値: 11.724286 KDa |
| 配列 | 文字列: MIELEVLDES KQKFSVILND RRVTIELWYN TTNDRWSFSL ALDGDNVVTG RRLVTGVDLL APFGLGIGAL FLLSENGEPP TRANLPLGL VKLYHATQEE IDAAISA |
-分子 #6: Pam3 hub gp19
| 分子 | 名称: Pam3 hub gp19 / タイプ: protein_or_peptide / ID: 6 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  uncultured cyanophage (ファージ) uncultured cyanophage (ファージ) |
| 分子量 | 理論値: 25.201736 KDa |
| 配列 | 文字列: MLEVGYTPPG GGNNIGIIAK GKIRDYQHDR EGGDIISTVS CGDGDKAYRR ATISKTIPKG TPLTEVVDEI HKEMKKEGIA KGEWSFPDK VKNTQLKRPY SMCGSCTREL DTLGRSHGFY WNIQNETMEI IPGNGHLPGS LLITPDTGLI GTPTITDNGI K VKALINPE ...文字列: MLEVGYTPPG GGNNIGIIAK GKIRDYQHDR EGGDIISTVS CGDGDKAYRR ATISKTIPKG TPLTEVVDEI HKEMKKEGIA KGEWSFPDK VKNTQLKRPY SMCGSCTREL DTLGRSHGFY WNIQNETMEI IPGNGHLPGS LLITPDTGLI GTPTITDNGI K VKALINPE ARPNRMMKVK SQVLKMNRKG EDYRISEVSF NGDNQEGDWI MTLTGEAIGG DKKVDEGKKA K |
-分子 #7: Pam3 spike gp20
| 分子 | 名称: Pam3 spike gp20 / タイプ: protein_or_peptide / ID: 7 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  uncultured cyanophage (ファージ) uncultured cyanophage (ファージ) |
| 分子量 | 理論値: 23.537537 KDa |
| 配列 | 文字列: MTGYLGKTTN QDRDVVSAVA QSERESVWGE MPGRVVSVSA DGRTVTVQPL YKPKFNGVPT DMPVLQEVPL RQALMGGVGV TVPVKPGQN VTLRPQMRSM DNYHVEGDGS ASDSRSFSLS DMEAHIAGGE SLKDVIPNLD SENVHVRANA DGSKGMKLSP E GKVEFQGP ...文字列: MTGYLGKTTN QDRDVVSAVA QSERESVWGE MPGRVVSVSA DGRTVTVQPL YKPKFNGVPT DMPVLQEVPL RQALMGGVGV TVPVKPGQN VTLRPQMRSM DNYHVEGDGS ASDSRSFSLS DMEAHIAGGE SLKDVIPNLD SENVHVRANA DGSKGMKLSP E GKVEFQGP EGNLLDLIAD FMELVANDKL QINYGSSAGT NHAMANKAAM LALAAKVRTT GTI |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 300 K |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: NONE |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.19 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: RELION (ver. 3.1) / 使用した粒子像数: 45155 |
| 初期 角度割当 | タイプ: RANDOM ASSIGNMENT |
| 最終 角度割当 | タイプ: RANDOM ASSIGNMENT |
 ムービー
ムービー コントローラー
コントローラー














 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)