+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | 3.1 Angstrom cryoEM icosahedral reconstruction of mud crab reovirus | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Reovirus / ds-RNA virus / VIRUS | |||||||||
| 機能・相同性 | VP3 / VP11 / VP12 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Scylla serrata reovirus SZ-2007 (ウイルス) Scylla serrata reovirus SZ-2007 (ウイルス) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.1 Å | |||||||||
 データ登録者 データ登録者 | Zhang Q / Gao Y | |||||||||
| 資金援助 |  中国, 2件 中国, 2件
| |||||||||
 引用 引用 |  ジャーナル: PLoS Pathog / 年: 2023 ジャーナル: PLoS Pathog / 年: 2023タイトル: The structure of a 12-segmented dsRNA reovirus: New insights into capsid stabilization and organization. 著者: Qinfen Zhang / Yuanzhu Gao / Matthew L Baker / Shanshan Liu / Xudong Jia / Haidong Xu / Jianguo He / Jason T Kaelber / Shaoping Weng / Wen Jiang /   要旨: Infecting a wide range of hosts, members of Reovirales (formerly Reoviridae) consist of a genome with different numbers of segmented double stranded RNAs (dsRNA) encapsulated by a proteinaceous shell ...Infecting a wide range of hosts, members of Reovirales (formerly Reoviridae) consist of a genome with different numbers of segmented double stranded RNAs (dsRNA) encapsulated by a proteinaceous shell and carry out genome replication and transcription inside the virion. Several cryo-electron microscopy (cryo-EM) structures of reoviruses with 9, 10 or 11 segmented dsRNA genomes have revealed insights into genome arrangement and transcription. However, the structure and genome arrangement of 12-segmented Reovirales members remain poorly understood. Using cryo-EM, we determined the structure of mud crab reovirus (MCRV), a 12-segmented dsRNA virus that is a putative member of Reovirales in the non-turreted Sedoreoviridae family, to near-atomic resolutions with icosahedral symmetry (3.1 Å) and without imposing icosahedral symmetry (3.4 Å). These structures revealed the organization of the major capsid proteins in two layers: an outer T = 13 layer consisting of VP12 trimers and unique VP11 clamps, and an inner T = 1 layer consisting of VP3 dimers. Additionally, ten RNA dependent RNA polymerases (RdRp) were well resolved just below the VP3 layer but were offset from the 5-fold axes and arranged with D5 symmetry, which has not previously been seen in other members of Reovirales. The N-termini of VP3 were shown to adopt four unique conformations; two of which anchor the RdRps, while the other two conformations are likely involved in genome organization and capsid stability. Taken together, these structures provide a new level of understanding for capsid stabilization and genome organization of segmented dsRNA viruses. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_33403.map.gz emd_33403.map.gz | 706.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-33403-v30.xml emd-33403-v30.xml emd-33403.xml emd-33403.xml | 19.7 KB 19.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_33403_fsc.xml emd_33403_fsc.xml | 28 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_33403.png emd_33403.png | 296.2 KB | ||
| Filedesc metadata |  emd-33403.cif.gz emd-33403.cif.gz | 6.2 KB | ||
| その他 |  emd_33403_half_map_1.map.gz emd_33403_half_map_1.map.gz emd_33403_half_map_2.map.gz emd_33403_half_map_2.map.gz | 706.8 MB 706.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-33403 http://ftp.pdbj.org/pub/emdb/structures/EMD-33403 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33403 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33403 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_33403_validation.pdf.gz emd_33403_validation.pdf.gz | 1.1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_33403_full_validation.pdf.gz emd_33403_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  emd_33403_validation.xml.gz emd_33403_validation.xml.gz | 36.7 KB | 表示 | |
| CIF形式データ |  emd_33403_validation.cif.gz emd_33403_validation.cif.gz | 49.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33403 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33403 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33403 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33403 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_33403.map.gz / 形式: CCP4 / 大きさ: 1.9 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_33403.map.gz / 形式: CCP4 / 大きさ: 1.9 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.09 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_33403_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
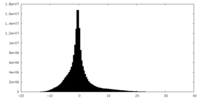
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_33403_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
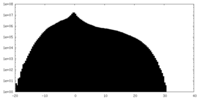
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Scylla serrata reovirus SZ-2007
| 全体 | 名称:  Scylla serrata reovirus SZ-2007 (ウイルス) Scylla serrata reovirus SZ-2007 (ウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Scylla serrata reovirus SZ-2007
| 超分子 | 名称: Scylla serrata reovirus SZ-2007 / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all / NCBI-ID: 458682 / 生物種: Scylla serrata reovirus SZ-2007 / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:  Scylla serrata (ノコギリガザミ) Scylla serrata (ノコギリガザミ) |
-分子 #1: VP3
| 分子 | 名称: VP3 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Scylla serrata reovirus SZ-2007 (ウイルス) Scylla serrata reovirus SZ-2007 (ウイルス) |
| 分子量 | 理論値: 97.019383 KDa |
| 配列 | 文字列: MASTTRLVND RKQLEQQVKD DARILADARG LNITTVANDS ATGGQAIRNV GPNDEATIKA LDNVIKQIEA LSVIVNRSEK ADDAQILGP NTYKQLLEHL FSPEENVYIL LPIQAYTGGV IDRRDASFSN FAYSIASKLM MELSAATHNK IFTDYTRIAA S ALGPEIST ...文字列: MASTTRLVND RKQLEQQVKD DARILADARG LNITTVANDS ATGGQAIRNV GPNDEATIKA LDNVIKQIEA LSVIVNRSEK ADDAQILGP NTYKQLLEHL FSPEENVYIL LPIQAYTGGV IDRRDASFSN FAYSIASKLM MELSAATHNK IFTDYTRIAA S ALGPEIST EGMPLFSLIE SLELTEAETS RLPVIQDSMV IQKSTATVGN AQQGISTINI KRVPFVGSAF QQVIDQLLWE YS TTSLTTK EQRRQRITEM VNDRRIMIQK LTLAEKPQVM RHVTTEINND LFFKMSPVAQ LYIYHLDRAF LDGVGFTPLA EKQ QQLQLQ LKTNILTANL IRSAINGMNT ESNLEVAIKM MQAAQLHRAS IEIAFPMNVS LSPEIIVQCF IVWMSIPEQL LSDR SNFII AAVIWAGFSA DDSYADIMRR SARASDRQNY DIIKAALSSR KFKLPRASTT LFDENEPVVR RYQIGRVYAP FPVDR YGSP VYSNCTKVEL ASDYNAEGFT IRKDDFRALQ AVLRIDEDRA ADMFTTLRIM ISSIPAVWYD AEVVHYPHTA VELEQL AAY GLTGAYPRTN HSVDTIVKTV NNISATYSTI AQMLSTIDLD PTRYGTSESI DKFKIAWENV ESVLNMEGND FVKTIMY AY EDNFPKKDFY MMLKQIASDG QGAHPIAAAI DQLRTIVYRE PERFGYIDSV ILTHNPDVDT AYNRFFHLHP IVTNQPSN T IKNAQLWNEM RLEQQVEHIK AGPVRIIGPF HVTYNYLSEE EDMPATSHII MKDNMILNDH LTFNFVKRER RNNKKRVSS FRYKAVEMYV AVRISRFQLE VLRDLHDLVR SRTYLDVSKS PLATTPIRVV EYVR UniProtKB: VP3 |
-分子 #2: VP11
| 分子 | 名称: VP11 / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Scylla serrata reovirus SZ-2007 (ウイルス) Scylla serrata reovirus SZ-2007 (ウイルス) |
| 分子量 | 理論値: 23.750701 KDa |
| 配列 | 文字列: MNWSKAINFQ PFMLETRPPL TTIPIMDQLV EIGERSNQKW SMTDRLFFAI RKINPIFVTS SQIPSKFDYT ILQMPTQLIA SLKETLLFL AFSYYLREYQ DKVGQMKFYP VAMKNMIPIV NYLKDRVHNN FDTTLEQAYR QNVVHTLSAS DAFDLLSGMI A TTRLDLIQ ...文字列: MNWSKAINFQ PFMLETRPPL TTIPIMDQLV EIGERSNQKW SMTDRLFFAI RKINPIFVTS SQIPSKFDYT ILQMPTQLIA SLKETLLFL AFSYYLREYQ DKVGQMKFYP VAMKNMIPIV NYLKDRVHNN FDTTLEQAYR QNVVHTLSAS DAFDLLSGMI A TTRLDLIQ RTRICPELLN VLNKMSFILI YAPNRPSILS WKNQS UniProtKB: VP11 |
-分子 #3: VP12
| 分子 | 名称: VP12 / タイプ: protein_or_peptide / ID: 3 / コピー数: 13 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Scylla serrata reovirus SZ-2007 (ウイルス) Scylla serrata reovirus SZ-2007 (ウイルス) |
| 分子量 | 理論値: 29.730963 KDa |
| 配列 | 文字列: MNLEINNFAP AISSIGSQLC SLSAQKLLTC RKQYGNGAKS FEEFYAEIGG IIGMMGINSQ TPSGIREAIY RLYQSAFLFG DIFPESFGI QNTQNIKPPP GFTAPAKKLE VVLPQGGAFD LIYNNGEIRV TTTRNVQAGD LVCTVTFPIQ GSVIATRNCH V NEIGGQLT ...文字列: MNLEINNFAP AISSIGSQLC SLSAQKLLTC RKQYGNGAKS FEEFYAEIGG IIGMMGINSQ TPSGIREAIY RLYQSAFLFG DIFPESFGI QNTQNIKPPP GFTAPAKKLE VVLPQGGAFD LIYNNGEIRV TTTRNVQAGD LVCTVTFPIQ GSVIATRNCH V NEIGGQLT TTRPEIIASV PMPARTVIVA SFDAIEIGYG EGDDLFAIGI AILSNRFNGQ ITPMSRHNYM TQMFANLPAN MS ERDSSAV LHFAQAAPVV LGMMERLTGA PKWVLDY UniProtKB: VP12 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 101.325 kPa |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 温度 | 最低: 80.0 K / 最高: 100.0 K |
| 撮影 | フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 検出モード: INTEGRATING / デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / デジタル化 - 画像ごとのフレーム数: 1-16 / 撮影したグリッド数: 1 / 実像数: 3595 / 平均露光時間: 1.1 sec. / 平均電子線量: 25.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 最大 デフォーカス(補正後): 3.0 µm / 最小 デフォーカス(補正後): 0.6 µm / 倍率(補正後): 128440 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 0.6 µm / 倍率(公称値): 75000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)