+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-31519 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | structure of a channel | |||||||||
 マップデータ マップデータ | Cryo-EM map of a channel | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報voltage-gated sodium channel activity involved in AV node cell action potential / voltage-gated sodium channel activity involved in bundle of His cell action potential / voltage-gated sodium channel activity involved in SA node cell action potential / bundle of His cell action potential / AV node cell action potential / sodium channel complex / SA node cell action potential / AV node cell to bundle of His cell communication / membrane depolarization during SA node cell action potential / response to denervation involved in regulation of muscle adaptation ...voltage-gated sodium channel activity involved in AV node cell action potential / voltage-gated sodium channel activity involved in bundle of His cell action potential / voltage-gated sodium channel activity involved in SA node cell action potential / bundle of His cell action potential / AV node cell action potential / sodium channel complex / SA node cell action potential / AV node cell to bundle of His cell communication / membrane depolarization during SA node cell action potential / response to denervation involved in regulation of muscle adaptation / cardiac ventricle development / regulation of ventricular cardiac muscle cell membrane depolarization / regulation of atrial cardiac muscle cell membrane repolarization / membrane depolarization during atrial cardiac muscle cell action potential / voltage-gated sodium channel activity involved in Purkinje myocyte action potential / regulation of sodium ion transmembrane transport / brainstem development / membrane depolarization during AV node cell action potential / voltage-gated sodium channel activity involved in cardiac muscle cell action potential / membrane depolarization during bundle of His cell action potential / positive regulation of action potential / atrial cardiac muscle cell action potential / membrane depolarization during Purkinje myocyte cell action potential / telencephalon development / regulation of atrial cardiac muscle cell membrane depolarization / membrane depolarization during cardiac muscle cell action potential / positive regulation of sodium ion transport / sodium ion import across plasma membrane / ventricular cardiac muscle cell action potential / cardiac muscle cell action potential involved in contraction / regulation of ventricular cardiac muscle cell membrane repolarization / regulation of cardiac muscle cell contraction / voltage-gated sodium channel complex / membrane depolarization during action potential / voltage-gated sodium channel activity / ankyrin binding / sodium ion transport / positive regulation of heart rate / nitric-oxide synthase binding / fibroblast growth factor binding / regulation of heart rate by cardiac conduction / odontogenesis of dentin-containing tooth / membrane depolarization / intercalated disc / sodium ion transmembrane transport / lateral plasma membrane / neuronal action potential / cardiac muscle contraction / T-tubule / cellular response to calcium ion / regulation of heart rate / bioluminescence / cerebellum development / generation of precursor metabolites and energy / caveola / positive regulation of epithelial cell proliferation / response to organic cyclic compound / sarcolemma / Z disc / scaffold protein binding / transmembrane transporter binding / calmodulin binding / symbiont entry into host cell / protein domain specific binding / axon / viral envelope / ubiquitin protein ligase binding / virion attachment to host cell / protein kinase binding / perinuclear region of cytoplasm / enzyme binding / cell surface / endoplasmic reticulum / membrane / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |   Recombinant vesicular stomatitis Indiana virus rVSV-G/GFP (ウイルス) Recombinant vesicular stomatitis Indiana virus rVSV-G/GFP (ウイルス) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.4 Å | |||||||||
 データ登録者 データ登録者 | Jiang DJ / Catterall WA | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2021 ジャーナル: Cell / 年: 2021タイトル: Open-state structure and pore gating mechanism of the cardiac sodium channel. 著者: Daohua Jiang / Richard Banh / Tamer M Gamal El-Din / Lige Tonggu / Michael J Lenaeus / Régis Pomès / Ning Zheng / William A Catterall /    要旨: The heartbeat is initiated by voltage-gated sodium channel Na1.5, which opens rapidly and triggers the cardiac action potential; however, the structural basis for pore opening remains unknown. Here, ...The heartbeat is initiated by voltage-gated sodium channel Na1.5, which opens rapidly and triggers the cardiac action potential; however, the structural basis for pore opening remains unknown. Here, we blocked fast inactivation with a mutation and captured the elusive open-state structure. The fast inactivation gate moves away from its receptor, allowing asymmetric opening of pore-lining S6 segments, which bend and rotate at their intracellular ends to dilate the activation gate to ∼10 Å diameter. Molecular dynamics analyses predict physiological rates of Na conductance. The open-state pore blocker propafenone binds in a high-affinity pose, and drug-access pathways are revealed through the open activation gate and fenestrations. Comparison with mutagenesis results provides a structural map of arrhythmia mutations that target the activation and fast inactivation gates. These results give atomic-level insights into molecular events that underlie generation of the action potential, open-state drug block, and fast inactivation of cardiac sodium channels, which initiate the heartbeat. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_31519.map.gz emd_31519.map.gz | 117.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-31519-v30.xml emd-31519-v30.xml emd-31519.xml emd-31519.xml | 14 KB 14 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_31519_fsc.xml emd_31519_fsc.xml | 9.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_31519.png emd_31519.png | 71 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-31519 http://ftp.pdbj.org/pub/emdb/structures/EMD-31519 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-31519 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-31519 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_31519_validation.pdf.gz emd_31519_validation.pdf.gz | 456.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_31519_full_validation.pdf.gz emd_31519_full_validation.pdf.gz | 456.2 KB | 表示 | |
| XML形式データ |  emd_31519_validation.xml.gz emd_31519_validation.xml.gz | 11.6 KB | 表示 | |
| CIF形式データ |  emd_31519_validation.cif.gz emd_31519_validation.cif.gz | 15.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31519 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31519 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31519 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31519 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_31519.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_31519.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM map of a channel | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.056 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : sodium channel with four homologous domains
| 全体 | 名称: sodium channel with four homologous domains |
|---|---|
| 要素 |
|
-超分子 #1: sodium channel with four homologous domains
| 超分子 | 名称: sodium channel with four homologous domains / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  Mammalian 1 orthobornavirus (ウイルス) Mammalian 1 orthobornavirus (ウイルス) |
| 分子量 | 理論値: 250 KDa |
-分子 #1: Sodium channel protein type 5 subunit alpha,Sodium channel protei...
| 分子 | 名称: Sodium channel protein type 5 subunit alpha,Sodium channel protein type 5 subunit alpha,Sodium channel protein type 5 subunit alpha,G protein/GFP fusion protein タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Recombinant vesicular stomatitis Indiana virus rVSV-G/GFP (ウイルス) Recombinant vesicular stomatitis Indiana virus rVSV-G/GFP (ウイルス) |
| 分子量 | 理論値: 209.030625 KDa |
| 組換発現 | 生物種:  Mammalian 1 orthobornavirus (ウイルス) Mammalian 1 orthobornavirus (ウイルス) |
| 配列 | 文字列: MANLLLPRGT SSFRRFTRES LAAIEKRMAE KQARGGSATS QESREGLQEE EAPRPQLDLQ ASKKLPDLYG NPPRELIGEP LEDLDPFYS TQKTFIVLNK GKTIFRFSAT NALYVLSPFH PVRRAAVKIL VHSLFSMLIM CTILTNCVFM AQHDPPPWTK Y VEYTFTAI ...文字列: MANLLLPRGT SSFRRFTRES LAAIEKRMAE KQARGGSATS QESREGLQEE EAPRPQLDLQ ASKKLPDLYG NPPRELIGEP LEDLDPFYS TQKTFIVLNK GKTIFRFSAT NALYVLSPFH PVRRAAVKIL VHSLFSMLIM CTILTNCVFM AQHDPPPWTK Y VEYTFTAI YTFESLVKIL ARGFCLHAFT FLRDPWNWLD FSVIVMAYTT EFVDLGNVSA LRTFRVLRAL KTISVISGLK TI VGALIQS VKKLADVMVL TVFCLSVFAL IGLQLFMGNL RHKCVRNFTE LNGTNGSVEA DGLVWNSLDV YLNDPANYLL KNG TTDVLL CGNSSDAGTC PEGYRCLKAG ENPDHGYTSF DSFAWAFLAL FRLMTQDCWE RLYQQTLRSA GKIYMIFFML VIFL GSFYL VNLILAVVAM AYEEQNQATI AETEEKEKRF QEAMEMLKKE HEALTIRGVD TVSRSSARQR ALSAVSVLTS ALEEL EESH RKCPPCWNRF AQHYLIWECC PLWMSIKQKV KFVVMDPFAD LTITMCIVLN TLFMALEHYN MTAEFEEMLQ VGNLVF TGI FTAEMTFKII ALDPYYYFQQ GWNIFDSIIV ILSLMELGLS RMGNLSVLRS FRLLRVFKLA KSWPTLNTLI KIIGNSV GA LGNLTLVLAI IVFIFAVVGM QLFGKNYSEL RHRISDSGLL PRWHMMDFFH AFLIIFRILC GEWIETMWDC MEVSGQSL C LLVFLLVMVI GNLVVLNLFL ALLLSSFSAD NLTAPDEDGE MNNLQLALAR IQRGLRFVKR TTWDFCCGIL RRRPKKPAA LATHSQLPSC ITAPRSPPPP EVEKVPPARK ETRFEEDKRP GQGTPGDSEP VCVPIAVAES DTEDQEEDEE NGKVWWRLRK TCYRIVEHS WFETFIIFMI LLSSGALAFE DIYLEERKTI KVLLEYADKM FTYVFVLEML LKWVAYGFKK YFTNAWCWLD F LIVDVSLV SLVANTLGFA EMGPIKSLRT LRALRPLRAL SRFEGMRVVV NALVGAIPSI MNVLLVCLIF WLIFSIMGVN LF AGKFGRC INQTEGDLPL NYTIVNNKSE CESFNVTGEL YWTKVKVNFD NVGAGYLALL QVATFKGWMD IMYAAVDSRG YEE QPQWED NLYMYIYFVV FIIFGSFFTL NLFIGVIIDN FNQQKKKLGG QDQQQTEEQK KYYNAMKKLG SKKPQKPIPR PLNK YQGFI FDIVTKQAFD VTIMFLICLN MVTMMVETDD QSPEKVNILA KINLLFVAIF TGECIVKMAA LRHYYFTNSW NIFDF VVVI LSIVGTVLSD IIQKYFFSPT LFRVIRLARI GRILRLIRGA KGIRTLLFAL MMSLPALFNI GLLLFLVMFI YSIFGM ANF AYVKWEAGID DMFNFQTFAN SMLCLFQITT SAGWDGLLSP ILNTGPPYCD PNLPNSNGSR GNCGSPAVGI LFFTTYI II SFLIVVNMYI AIILENFSVA TEESTEPLSE DDFDMFYEIW EKFDPEATQF IEYLALSDFA DALSEPLRIA KPNQISLI N MDLPMVSGDR IHCMDILFAF TKRVLGESGE MDALKIQMEE KFMAANPSKI SYEPITTTLE VLFQGPGSMV SKGEELFTG VVPILVELDG DVNGHKFSVS GEGEGDATYG KLTLKFICTT GKLPVPWPTL VTTLTYGVQC FSRYPDHMKQ HDFFKSAMPE GYVQERTIF FKDDGNYKTR AEVKFEGDTL VNRIELKGID FKEDGNILGH KLEYNYNSHN VYIMADKQKN GIKVNFKIRH N IEDGSVQL ADHYQQNTPI GDGPVLLPDN HYLSTQSALS KDPNEKRDHM VLLEFVTAAG ITLGMDELYK GSDYKDDDDK |
-分子 #2: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 2 / コピー数: 3 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-分子 #3: [(2~{R})-1-[2-azanylethoxy(oxidanyl)phosphoryl]oxy-3-hexadecanoyl...
| 分子 | 名称: [(2~{R})-1-[2-azanylethoxy(oxidanyl)phosphoryl]oxy-3-hexadecanoyloxy-propan-2-yl] (~{Z})-octadec-9-enoate タイプ: ligand / ID: 3 / コピー数: 11 / 式: 6OU |
|---|---|
| 分子量 | 理論値: 717.996 Da |
| Chemical component information | 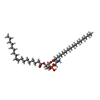 ChemComp-6OU: |
-分子 #4: 1-[2-[(2R)-2-oxidanyl-3-(propylamino)propoxy]phenyl]-3-phenyl-pro...
| 分子 | 名称: 1-[2-[(2R)-2-oxidanyl-3-(propylamino)propoxy]phenyl]-3-phenyl-propan-1-one タイプ: ligand / ID: 4 / コピー数: 1 / 式: 4Y4 |
|---|---|
| 分子量 | 理論値: 341.444 Da |
| Chemical component information | 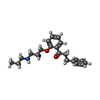 ChemComp-4Y4: |
-分子 #5: (3beta,14beta,17beta,25R)-3-[4-methoxy-3-(methoxymethyl)butoxy]sp...
| 分子 | 名称: (3beta,14beta,17beta,25R)-3-[4-methoxy-3-(methoxymethyl)butoxy]spirost-5-en タイプ: ligand / ID: 5 / コピー数: 2 / 式: 9Z9 |
|---|---|
| 分子量 | 理論値: 544.805 Da |
| Chemical component information | 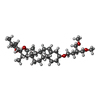 ChemComp-9Z9: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 5 mg/mL |
|---|---|
| 緩衝液 | pH: 6 |
| グリッド | 材質: GOLD / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum ER / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.4 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 287306 |
|---|---|
| 初期 角度割当 | タイプ: ANGULAR RECONSTITUTION |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| FSC曲線 (解像度の算出) | 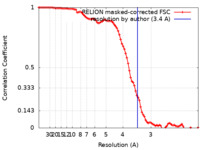 |
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
|---|---|
| 得られたモデル |  PDB-7fbs: |
 ムービー
ムービー コントローラー
コントローラー















