+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-30583 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
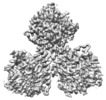
| タイトル | CryoEM structure of cotton cellulose synthase isoform 7 | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | cellulose synthase / cotton / MEMBRANE PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報cellulose synthase activity / plant-type primary cell wall biogenesis / cellulose synthase (UDP-forming) / cellulose synthase (UDP-forming) activity / cellulose biosynthetic process / trans-Golgi network / cell wall organization / membrane => GO:0016020 / metal ion binding / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.5 Å | |||||||||
 データ登録者 データ登録者 | Guan ZY / Xue Y | |||||||||
 引用 引用 |  ジャーナル: Plant Biotechnol J / 年: 2021 ジャーナル: Plant Biotechnol J / 年: 2021タイトル: Structural insights into homotrimeric assembly of cellulose synthase CesA7 from Gossypium hirsutum. 著者: Xiangnan Zhang / Yuan Xue / Zeyuan Guan / Chen Zhou / Yangfan Nie / She Men / Qiang Wang / Cuicui Shen / Delin Zhang / Shuangxia Jin / Lili Tu / Ping Yin / Xianlong Zhang /  要旨: Cellulose is one of the most abundant organic polymers in nature. It contains multiple β-1,4-glucan chains synthesized by cellulose synthases (CesAs) on the plasma membrane of higher plants. CesA ...Cellulose is one of the most abundant organic polymers in nature. It contains multiple β-1,4-glucan chains synthesized by cellulose synthases (CesAs) on the plasma membrane of higher plants. CesA subunits assemble into a pseudo-sixfold symmetric cellulose synthase complex (CSC), known as a 'rosette complex'. The structure of CesA remains enigmatic. Here, we report the cryo-EM structure of the homotrimeric CesA7 from Gossypium hirsutum at 3.5-angstrom resolution. The GhCesA7 homotrimer shows a C3 symmetrical assembly. Each protomer contains seven transmembrane helices (TMs) which form a channel potentially facilitating the release of newly synthesized glucans. The cytoplasmic glycosyltransferase domain (GT domain) of GhCesA7 protrudes from the membrane, and its catalytic pocket is directed towards the TM pore. The homotrimer GhCesA7 is stabilized by the transmembrane helix 7 (TM7) and the plant-conserved region (PCR) domains. It represents the building block of CSCs and facilitates microfibril formation. This structure provides insight into how eukaryotic cellulose synthase assembles and provides a mechanistic basis for the improvement of cotton fibre quality in the future. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_30583.map.gz emd_30583.map.gz | 5.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-30583-v30.xml emd-30583-v30.xml emd-30583.xml emd-30583.xml | 10.5 KB 10.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_30583.png emd_30583.png | 87.4 KB | ||
| Filedesc metadata |  emd-30583.cif.gz emd-30583.cif.gz | 5.7 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-30583 http://ftp.pdbj.org/pub/emdb/structures/EMD-30583 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-30583 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-30583 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_30583_validation.pdf.gz emd_30583_validation.pdf.gz | 371.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_30583_full_validation.pdf.gz emd_30583_full_validation.pdf.gz | 370.7 KB | 表示 | |
| XML形式データ |  emd_30583_validation.xml.gz emd_30583_validation.xml.gz | 6.4 KB | 表示 | |
| CIF形式データ |  emd_30583_validation.cif.gz emd_30583_validation.cif.gz | 7.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-30583 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-30583 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-30583 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-30583 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_30583.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_30583.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.087 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Cellulose synthase
| 全体 | 名称: Cellulose synthase |
|---|---|
| 要素 |
|
-超分子 #1: Cellulose synthase
| 超分子 | 名称: Cellulose synthase / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Cellulose synthase
| 分子 | 名称: Cellulose synthase / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO / EC番号: cellulose synthase (UDP-forming) |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 118.129711 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MEASAGLVAG SHNRNELVVI HGHEEPKPLK NLDGQVCEIC GDEIGVTVDG DLFVACNECG FPVCRPCYEY ERREGTQQCP QCKTRYKRL KGSPRVEGDE DEEDVDDIEH EFNIDDEQNK HRNVVESILH GKMSYGRGPE DDETPQIPVI TGVRSRPVSG E FPIAGALA ...文字列: MEASAGLVAG SHNRNELVVI HGHEEPKPLK NLDGQVCEIC GDEIGVTVDG DLFVACNECG FPVCRPCYEY ERREGTQQCP QCKTRYKRL KGSPRVEGDE DEEDVDDIEH EFNIDDEQNK HRNVVESILH GKMSYGRGPE DDETPQIPVI TGVRSRPVSG E FPIAGALA YGEHMPNASL HKRVHPYPMS ETEGAERWDD KKEGGWKERM DDWKMQQGNL GPEADDAYDD MSMLDEARQP LS RKVPIAS SKINPYRMVI VARLLILAFF LRYRILNPVH DAIGLWLTSV ICEIWFAFSW ILDQFPKWFP IDRETYLDRL SLR YEREGE PNMLAPVDIF VSTVDPMKEP PLVTANTVLS ILAMDYPVDK ISCYISDDGA SMLTFESLSE TAEFARKWVP FCKK FAIEP RAPEMYFTLK VDYLKDKVQP TFVKERRAMK REYEEFKVRI NALVAKAQKV PPEGWIMQDG TPWPGNNTKD HPGMI QVFL GQSGGHDTEG NELPRLVYVS REKRPGFLHH KKAGAMNALV RVSGVLTNAP FMLNLDCDHY INNSKAAREA MCFLMD PQI GRKVCYVQFP QRFDGIDRHD RYANRNTVFF DINMKGLDGI QGPVYVGTGC VFRRQALYGY EPPKGPKRPK MVSCGCC PC FGRRKKDKKY PKNGGNENGP SLEAVEDDKE LLMSQMNFEK KFGQSAIFVT STLMDQGGVP PSSSPAALLK EAIHVISC G YEDKTEWGSE LGWIYGSITE DILTGFKMHC RGWRSIYCMP KLPAFKGSAP INLSDRLNQV LRWALGSVEI FFSRHCPAW YGLKGAKLRW LERFAYVNTT IYPFTSLPLL AYCTLPAICL LTDKFIMPPI STFASLFFIA LFLSIFATGI LELRWSGVSI EEWWRNEQF WVIGGISAHL FAVVQGLLKV LAGIDTNFTV TSKTTDDEEF GELYTFKWTT LLIPPTTVLI INLVGVVAGI S DAINNGYQ SWGPLFGKLF FSFWVIVHLY PFLKGLMGRQ NRTPTIVVIW SVLLASIFSL LWVRIDPFVL KTKGPDTTQC GI NC UniProtKB: Cellulose synthase |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 5 mg/mL |
|---|---|
| 緩衝液 | pH: 8 |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー