+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-30465 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of dengue virus serotype 2 in complex with the scFv fragment of the broadly neutralizing antibody EDE1 C10 | ||||||||||||
 マップデータ マップデータ | Dengue 2 NGC With ScFv C10 | ||||||||||||
 試料 試料 |
| ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報: / flavivirin / symbiont-mediated suppression of host JAK-STAT cascade via inhibition of STAT2 activity / double-stranded RNA binding / viral capsid / nucleoside-triphosphate phosphatase / mRNA (nucleoside-2'-O-)-methyltransferase activity / mRNA 5'-cap (guanine-N7-)-methyltransferase activity / membrane => GO:0016020 / RNA helicase activity ...: / flavivirin / symbiont-mediated suppression of host JAK-STAT cascade via inhibition of STAT2 activity / double-stranded RNA binding / viral capsid / nucleoside-triphosphate phosphatase / mRNA (nucleoside-2'-O-)-methyltransferase activity / mRNA 5'-cap (guanine-N7-)-methyltransferase activity / membrane => GO:0016020 / RNA helicase activity / host cell endoplasmic reticulum membrane / protein dimerization activity / symbiont-mediated suppression of host type I interferon-mediated signaling pathway / RNA helicase / induction by virus of host autophagy / viral RNA genome replication / serine-type endopeptidase activity / RNA-dependent RNA polymerase activity / fusion of virus membrane with host endosome membrane / host cell nucleus / virion attachment to host cell / virion membrane / structural molecule activity / extracellular region / ATP binding / metal ion binding 類似検索 - 分子機能 | ||||||||||||
| 生物種 |  Dengue virus 2 (デング熱ウイルス) / Dengue virus 2 (デング熱ウイルス) /  Homo sapiens (ヒト) / Homo sapiens (ヒト) /   Dengue virus (デング熱ウイルス) Dengue virus (デング熱ウイルス) | ||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.5 Å | ||||||||||||
 データ登録者 データ登録者 | Zhang X / Sharma A / Duquerroy S / Zhou ZH / Rey FA | ||||||||||||
| 資金援助 |  フランス, フランス,  中国, 3件 中国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2021 ジャーナル: Cell / 年: 2021タイトル: The epitope arrangement on flavivirus particles contributes to Mab C10's extraordinary neutralization breadth across Zika and dengue viruses. 著者: Arvind Sharma / Xiaokang Zhang / Wanwisa Dejnirattisai / Xinghong Dai / Danyang Gong / Wiyada Wongwiwat / Stéphane Duquerroy / Alexander Rouvinski / Marie-Christine Vaney / Pablo Guardado- ...著者: Arvind Sharma / Xiaokang Zhang / Wanwisa Dejnirattisai / Xinghong Dai / Danyang Gong / Wiyada Wongwiwat / Stéphane Duquerroy / Alexander Rouvinski / Marie-Christine Vaney / Pablo Guardado-Calvo / Ahmed Haouz / Patrick England / Ren Sun / Z Hong Zhou / Juthathip Mongkolsapaya / Gavin R Screaton / Felix A Rey /      要旨: The human monoclonal antibody C10 exhibits extraordinary cross-reactivity, potently neutralizing Zika virus (ZIKV) and the four serotypes of dengue virus (DENV1-DENV4). Here we describe a comparative ...The human monoclonal antibody C10 exhibits extraordinary cross-reactivity, potently neutralizing Zika virus (ZIKV) and the four serotypes of dengue virus (DENV1-DENV4). Here we describe a comparative structure-function analysis of C10 bound to the envelope (E) protein dimers of the five viruses it neutralizes. We demonstrate that the C10 Fab has high affinity for ZIKV and DENV1 but not for DENV2, DENV3, and DENV4. We further show that the C10 interaction with the latter viruses requires an E protein conformational landscape that limits binding to only one of the three independent epitopes per virion. This limited affinity is nevertheless counterbalanced by the particle's icosahedral organization, which allows two different dimers to be reached by both Fab arms of a C10 immunoglobulin. The epitopes' geometric distribution thus confers C10 its exceptional neutralization breadth. Our results highlight the importance not only of paratope/epitope complementarity but also the topological distribution for epitope-focused vaccine design. #1:  ジャーナル: Nature / 年: 2016 ジャーナル: Nature / 年: 2016タイトル: Structural basis of potent Zika-dengue virus antibody cross-neutralization. 著者: Barba-Spaeth G / Dejnirattisai W / Rouvinski A / Vaney MC / Medits I / Sharma A / Simon-Loriere E / Sakuntabhai A / Cao-Lormeau VM / Haouz A / England P / Stiasny K / Mongkolsapaya J / Heinz ...著者: Barba-Spaeth G / Dejnirattisai W / Rouvinski A / Vaney MC / Medits I / Sharma A / Simon-Loriere E / Sakuntabhai A / Cao-Lormeau VM / Haouz A / England P / Stiasny K / Mongkolsapaya J / Heinz FX / Screaton GR / Rey FA | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_30465.map.gz emd_30465.map.gz | 925.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-30465-v30.xml emd-30465-v30.xml emd-30465.xml emd-30465.xml | 23.7 KB 23.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
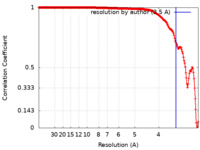
| FSC (解像度算出) |  emd_30465_fsc.xml emd_30465_fsc.xml | 21.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_30465.png emd_30465.png | 110.9 KB | ||
| その他 |  emd_30465_additional_1.map.gz emd_30465_additional_1.map.gz emd_30465_half_map_1.map.gz emd_30465_half_map_1.map.gz emd_30465_half_map_2.map.gz emd_30465_half_map_2.map.gz | 926.5 MB 155.5 MB 155.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-30465 http://ftp.pdbj.org/pub/emdb/structures/EMD-30465 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-30465 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-30465 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_30465_validation.pdf.gz emd_30465_validation.pdf.gz | 981.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_30465_full_validation.pdf.gz emd_30465_full_validation.pdf.gz | 981.5 KB | 表示 | |
| XML形式データ |  emd_30465_validation.xml.gz emd_30465_validation.xml.gz | 29.7 KB | 表示 | |
| CIF形式データ |  emd_30465_validation.cif.gz emd_30465_validation.cif.gz | 39.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-30465 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-30465 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-30465 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-30465 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_30465.map.gz / 形式: CCP4 / 大きさ: 1000 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_30465.map.gz / 形式: CCP4 / 大きさ: 1000 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Dengue 2 NGC With ScFv C10 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.28 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: unsharpen map
| ファイル | emd_30465_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | unsharpen map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half Map A
| ファイル | emd_30465_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half Map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half Map B
| ファイル | emd_30465_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half Map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Dengue virus
| 全体 | 名称:   Dengue virus (デング熱ウイルス) Dengue virus (デング熱ウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Dengue virus
| 超分子 | 名称: Dengue virus / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: #2-#4 / NCBI-ID: 12637 / 生物種: Dengue virus / ウイルスタイプ: VIRION / ウイルス・単離状態: SEROTYPE / ウイルス・エンベロープ: Yes / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Core protein
| 分子 | 名称: Core protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Dengue virus 2 (デング熱ウイルス) Dengue virus 2 (デング熱ウイルス) |
| 分子量 | 理論値: 54.351746 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MRCIGISNRD FVEGVSGGSW VDIVLEHGSC VTTMAKNKPT LDFELIKTEA KQPATLRKYC IEAKLTNTTT DSRCPTQGEP SLNEEQDKR FVCKHSMVDR GWGNGCGLFG KGGIVTCAMF TCKKNMKGKV VQPENLEYTI VITPHSGEEH AVGNDTGKHG K EIKITPQS ...文字列: MRCIGISNRD FVEGVSGGSW VDIVLEHGSC VTTMAKNKPT LDFELIKTEA KQPATLRKYC IEAKLTNTTT DSRCPTQGEP SLNEEQDKR FVCKHSMVDR GWGNGCGLFG KGGIVTCAMF TCKKNMKGKV VQPENLEYTI VITPHSGEEH AVGNDTGKHG K EIKITPQS SITEAELTGY GTVTMECSPR TGLDFNEMVL LQMENKAWLV HRQWFLDLPL PWLPGADTQG SNWIQKETLV TF KNPHAKK QDVVVLGSQE GAMHTALTGA TEIQMSSGNL LFTGHLKCRL RMDKLQLKGM SYSMCTGKFK VVKEIAETQH GTI VIRVQY EGDGSPCKIP FEIMDLEKRH VLGRLITVNP IVTEKDSPVN IEAEPPFGDS YIIIGVEPGQ LKLNWFKKGS SIGQ MIETT MRGAKRMAIL GDTAWDFGSL GGVFTSIGKA LHQVFGAIYG AAFSGVSWTM KILIGVIITW IGMNSRSTSL SVSLV LVGV VTLYLGVMVQ A |
-分子 #2: Core protein
| 分子 | 名称: Core protein / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO / EC番号: flavivirin |
|---|---|
| 由来(天然) | 生物種:  Dengue virus 2 (デング熱ウイルス) Dengue virus 2 (デング熱ウイルス) |
| 分子量 | 理論値: 8.345762 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: SVALVPHVGM GLETRTETWM SSEGAWKHAQ RIETWILRHP GFTIMAAILA YTIGTTHFQR ALIFILLTAV APSMT |
-分子 #3: Single Chain Variable Fragment
| 分子 | 名称: Single Chain Variable Fragment / タイプ: protein_or_peptide / ID: 3 詳細: heavy chain variable region of the broadly neutralizing antibody EDE1 C10 コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 15.679234 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MAEVQLVESG AEVKKPGASV KVSCKASGYT FTSYAMHWVR QAPGQRLEWM GWINAGNGNT KYSQKFQDRV TITRDTSAST AYMELSSLR SEDTAIYYCA RDKVDDYGDY WFPTLWYFDY WGQGTLVTVS SGTGGSGGGG SGGGG |
-分子 #4: Single Chain Variable Fragment
| 分子 | 名称: Single Chain Variable Fragment / タイプ: protein_or_peptide / ID: 4 詳細: light chain variable region of the broadly neutralizing antibody EDE1 C10 コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 15.632767 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: SGGGASQSAL TQPASVSGSP GQSITISCTG TSSDVGGFNY VSWFQQHPGK APKLMLYDVT SRPSGVSSRF SGSKSGNTAS LTISGLQAE DEADYYCSSH TSRGTWVFGG GTKLTVLAAA DDDDKAGWSH PQFEKGGGSG GGSGGGSWSH PQFEK |
-分子 #5: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 5 / コピー数: 4 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー





















 Z
Z Y
Y X
X