[English] 日本語
 Yorodumi
Yorodumi- EMDB-29878: Exploiting Activation and Inactivation Mechanisms in Type I-C CRI... -
+ Open data
Open data
- Basic information
Basic information
| Entry |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Title | Exploiting Activation and Inactivation Mechanisms in Type I-C CRISPR-Cas3 for Genome Editing Applications | |||||||||
 Map data Map data | ||||||||||
 Sample Sample |
| |||||||||
 Keywords Keywords | CRISPR / type I-C / cascade / anti-CRISPR / HYDROLASE-RNA complex | |||||||||
| Function / homology |  Function and homology information Function and homology informationmaintenance of CRISPR repeat elements / endonuclease activity / defense response to virus / Hydrolases; Acting on ester bonds / hydrolase activity / RNA binding Similarity search - Function | |||||||||
| Biological species |  Neisseria lactamica (bacteria) / Neisseria lactamica (bacteria) /  Rhodobacter phage RcNL1 (virus) Rhodobacter phage RcNL1 (virus) | |||||||||
| Method | single particle reconstruction / cryo EM / Resolution: 3.6 Å | |||||||||
 Authors Authors | Hu C / Nam KH / Ke A | |||||||||
| Funding support |  United States, 1 items United States, 1 items
| |||||||||
 Citation Citation |  Journal: Mol Cell / Year: 2024 Journal: Mol Cell / Year: 2024Title: Exploiting activation and inactivation mechanisms in type I-C CRISPR-Cas3 for genome-editing applications. Authors: Chunyi Hu / Mason T Myers / Xufei Zhou / Zhonggang Hou / Macy L Lozen / Ki Hyun Nam / Yan Zhang / Ailong Ke /    Abstract: Type I CRISPR-Cas systems utilize the RNA-guided Cascade complex to identify matching DNA targets and the nuclease-helicase Cas3 to degrade them. Among the seven subtypes, type I-C is compact in size ...Type I CRISPR-Cas systems utilize the RNA-guided Cascade complex to identify matching DNA targets and the nuclease-helicase Cas3 to degrade them. Among the seven subtypes, type I-C is compact in size and highly active in creating large-sized genome deletions in human cells. Here, we use four cryoelectron microscopy snapshots to define its RNA-guided DNA binding and cleavage mechanisms in high resolution. The non-target DNA strand (NTS) is accommodated by I-C Cascade in a continuous binding groove along the juxtaposed Cas11 subunits. Binding of Cas3 further traps a flexible bulge in NTS, enabling NTS nicking. We identified two anti-CRISPR proteins AcrIC8 and AcrIC9 that strongly inhibit Neisseria lactamica I-C function. Structural analysis showed that AcrIC8 inhibits PAM recognition through allosteric inhibition, whereas AcrIC9 achieves so through direct competition. Both Acrs potently inhibit I-C-mediated genome editing and transcriptional modulation in human cells, providing the first off-switches for type I CRISPR eukaryotic genome engineering. | |||||||||
| History |
|
- Structure visualization
Structure visualization
| Supplemental images |
|---|
- Downloads & links
Downloads & links
-EMDB archive
| Map data |  emd_29878.map.gz emd_29878.map.gz | 59.5 MB |  EMDB map data format EMDB map data format | |
|---|---|---|---|---|
| Header (meta data) |  emd-29878-v30.xml emd-29878-v30.xml emd-29878.xml emd-29878.xml | 27.4 KB 27.4 KB | Display Display |  EMDB header EMDB header |
| Images |  emd_29878.png emd_29878.png | 138.1 KB | ||
| Filedesc metadata |  emd-29878.cif.gz emd-29878.cif.gz | 7.3 KB | ||
| Others |  emd_29878_additional_1.map.gz emd_29878_additional_1.map.gz emd_29878_half_map_1.map.gz emd_29878_half_map_1.map.gz emd_29878_half_map_2.map.gz emd_29878_half_map_2.map.gz | 32.5 MB 59.5 MB 59.5 MB | ||
| Archive directory |  http://ftp.pdbj.org/pub/emdb/structures/EMD-29878 http://ftp.pdbj.org/pub/emdb/structures/EMD-29878 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-29878 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-29878 | HTTPS FTP |
-Related structure data
| Related structure data |  8g9tMC  8g9sC  8g9uC  8gafC  8gamC  8ganC M: atomic model generated by this map C: citing same article ( |
|---|---|
| Similar structure data | Similarity search - Function & homology  F&H Search F&H Search |
- Links
Links
| EMDB pages |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| Related items in Molecule of the Month |
- Map
Map
| File |  Download / File: emd_29878.map.gz / Format: CCP4 / Size: 64 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) Download / File: emd_29878.map.gz / Format: CCP4 / Size: 64 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Projections & slices | Image control
Images are generated by Spider. | ||||||||||||||||||||||||||||||||||||
| Voxel size | X=Y=Z: 1.489 Å | ||||||||||||||||||||||||||||||||||||
| Density |
| ||||||||||||||||||||||||||||||||||||
| Symmetry | Space group: 1 | ||||||||||||||||||||||||||||||||||||
| Details | EMDB XML:
|
-Supplemental data
-Additional map: #1
| File | emd_29878_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Projections & Slices |
| ||||||||||||
| Density Histograms |
-Half map: #2
| File | emd_29878_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Projections & Slices |
| ||||||||||||
| Density Histograms |
-Half map: #1
| File | emd_29878_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Projections & Slices |
| ||||||||||||
| Density Histograms |
- Sample components
Sample components
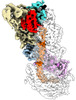
-Entire : Binary complex of AcrIC9 with crRNA bound type I-C Cascade
| Entire | Name: Binary complex of AcrIC9 with crRNA bound type I-C Cascade |
|---|---|
| Components |
|
-Supramolecule #1: Binary complex of AcrIC9 with crRNA bound type I-C Cascade
| Supramolecule | Name: Binary complex of AcrIC9 with crRNA bound type I-C Cascade type: complex / ID: 1 / Parent: 0 / Macromolecule list: all |
|---|---|
| Source (natural) | Organism:  Neisseria lactamica (bacteria) Neisseria lactamica (bacteria) |
| Molecular weight | Theoretical: 400 KDa |
-Macromolecule #1: AcrIC9
| Macromolecule | Name: AcrIC9 / type: protein_or_peptide / ID: 1 / Number of copies: 1 / Enantiomer: LEVO |
|---|---|
| Source (natural) | Organism:  Rhodobacter phage RcNL1 (virus) Rhodobacter phage RcNL1 (virus) |
| Molecular weight | Theoretical: 8.247822 KDa |
| Recombinant expression | Organism:  |
| Sequence | String: MTSFYKITAY NSQALYFWGT DADVDRYVDW LNRDREINVY AAEAIPEAEW AQYEGRDDVL SGEECGWDDF UniProtKB: Uncharacterized protein |
-Macromolecule #2: Cas7
| Macromolecule | Name: Cas7 / type: protein_or_peptide / ID: 2 / Number of copies: 7 / Enantiomer: LEVO |
|---|---|
| Source (natural) | Organism:  Neisseria lactamica (bacteria) Neisseria lactamica (bacteria) |
| Molecular weight | Theoretical: 32.208111 KDa |
| Recombinant expression | Organism:  |
| Sequence | String: TIEKRYDFVF LFDVQDGNPN GDPDAGNLPR IDPQTGEGLV TDVCLKRKVR NFIQMTQNDE HHDIFIREKG ILNNLIDEAH EQENVKGKE KGEKTEAARQ YMCSRYYDIR TFGAVMTTGK NAGQVRGPVQ LTFSRSIDPI MTLEHSITRM AVTNEKDASE T GDNRTMGR ...String: TIEKRYDFVF LFDVQDGNPN GDPDAGNLPR IDPQTGEGLV TDVCLKRKVR NFIQMTQNDE HHDIFIREKG ILNNLIDEAH EQENVKGKE KGEKTEAARQ YMCSRYYDIR TFGAVMTTGK NAGQVRGPVQ LTFSRSIDPI MTLEHSITRM AVTNEKDASE T GDNRTMGR KFTVPYGLYR CHGFISTHFA KQTGFSENDL ELFWQALVNM FDHDHSAARG QMNARGLYVF EHSNNLGDAP AD SLFKRIQ VVKKDGVEVV RSFDDYLVSV DDKNLEETKL LRKLGG UniProtKB: UNIPROTKB: A0A378VEU0 |
-Macromolecule #3: Cas11
| Macromolecule | Name: Cas11 / type: protein_or_peptide / ID: 3 / Number of copies: 4 / Enantiomer: LEVO |
|---|---|
| Source (natural) | Organism:  Neisseria lactamica (bacteria) Neisseria lactamica (bacteria) |
| Molecular weight | Theoretical: 14.245184 KDa |
| Recombinant expression | Organism:  |
| Sequence | String: GLDRNRQDIG YVLGRLFAVL EKIQAEANPG LNATIADRYF GSASSTPIAV FGTLMRLLPH HLNKLEFEGR AVQLQWEIRQ ILEHCQRFP NHLNLEQQGL FAIGYYHETQ FLFTKDALKN LFNEA UniProtKB: UNIPROTKB: A0A378VF47 |
-Macromolecule #4: Cas8
| Macromolecule | Name: Cas8 / type: protein_or_peptide / ID: 4 / Number of copies: 1 / Enantiomer: LEVO |
|---|---|
| Source (natural) | Organism:  Neisseria lactamica (bacteria) Neisseria lactamica (bacteria) |
| Molecular weight | Theoretical: 64.860832 KDa |
| Recombinant expression | Organism:  |
| Sequence | String: MILHALTQYY QRKAESDGGI AQEGFENKEI PFIIVIDKQG NFIQLEDTRE LKVKKKVGRT FLVPKGLGRS GSKSYEVSNL LWDHYGYVL AYAGEKGQEQ ADKQHASFTA KVNELKQALP DDAGVTAVAA FLSSAEEKSK VMQAANWAEC AKVKGCNLSF R LVDEAVDL ...String: MILHALTQYY QRKAESDGGI AQEGFENKEI PFIIVIDKQG NFIQLEDTRE LKVKKKVGRT FLVPKGLGRS GSKSYEVSNL LWDHYGYVL AYAGEKGQEQ ADKQHASFTA KVNELKQALP DDAGVTAVAA FLSSAEEKSK VMQAANWAEC AKVKGCNLSF R LVDEAVDL VCQSKAVREY VSQANQTQSD NAQKGICLVT GKAAPIARLH NAVKGVNAKP APFASVNLSA FESYGKEQGF AF PIGEQAM FEYTTALNTL LAGENRFRIG DVTTVCWGAK RTPLEESLAS MINGGGKDKP DEHIDAVKTL YKSLYNGQYQ KPD GKEKFY LLGLSPNSAR IVVRFWHETT VAALSESIAA WYDDLQMVRG ENSPYPEYMP LPRLLGNLVL DGKMENLPSD LIAQ ITDAA LNNRVLPVSL LQAALRRNKA EQKITYGRAS LLKAYINRAI RAGRLKNMKE LTMGLDRNRQ DIGYVLGRLF AVLEK IQAE ANPGLNATIA DRYFGSASST PIAVFGTLMR LLPHHLNKLE FEGRAVQLQW EIRQILEHCQ RFPNHLNLEQ QGLFAI GYY HETQFLFTKD ALKNLFNEA UniProtKB: UNIPROTKB: A0A378VF47 |
-Macromolecule #5: Cas5
| Macromolecule | Name: Cas5 / type: protein_or_peptide / ID: 5 / Number of copies: 1 / Enantiomer: LEVO |
|---|---|
| Source (natural) | Organism:  Neisseria lactamica (bacteria) Neisseria lactamica (bacteria) |
| Molecular weight | Theoretical: 23.870451 KDa |
| Recombinant expression | Organism:  |
| Sequence | String: RFILEISGDL ACFTRSELKV ERVSYPVITP SAARNILMAI LWKPAIRWKV LKIEILKPIQ WTNIRRNEVG TKMSERSGSL YIEDNRQQR ASMLLKDVAY RIHADFDMTS EAGESDNYVK FAEMFKRRAK KGQYFHQPYL GCREFPCDFR LLEKAEDGLP L EDITQDFG ...String: RFILEISGDL ACFTRSELKV ERVSYPVITP SAARNILMAI LWKPAIRWKV LKIEILKPIQ WTNIRRNEVG TKMSERSGSL YIEDNRQQR ASMLLKDVAY RIHADFDMTS EAGESDNYVK FAEMFKRRAK KGQYFHQPYL GCREFPCDFR LLEKAEDGLP L EDITQDFG FMLYDMDFSK SDPRDSNNAE PMFYQCKAVN GVITVPP UniProtKB: pre-crRNA processing endonuclease |
-Macromolecule #6: crRNA (43-MER)
| Macromolecule | Name: crRNA (43-MER) / type: rna / ID: 6 / Number of copies: 1 |
|---|---|
| Source (natural) | Organism:  Neisseria lactamica (bacteria) Neisseria lactamica (bacteria) |
| Molecular weight | Theoretical: 13.870306 KDa |
| Sequence | String: GAAACAGGGU CAGCUUGCCG UAGGUGGCAU CGCCCUCGUA AAA |
-Experimental details
-Structure determination
| Method | cryo EM |
|---|---|
 Processing Processing | single particle reconstruction |
| Aggregation state | particle |
- Sample preparation
Sample preparation
| Concentration | 1 mg/mL |
|---|---|
| Buffer | pH: 7.5 / Component - Concentration: 150.0 mM / Component - Formula: NaCl / Component - Name: sodium chloride / Details: 25mM Tris pH 7.5, 150mM NaCl |
| Grid | Model: Quantifoil R1.2/1.3 / Material: COPPER / Mesh: 400 / Support film - Material: CARBON / Support film - topology: HOLEY / Pretreatment - Type: GLOW DISCHARGE / Pretreatment - Time: 30 sec. |
| Vitrification | Cryogen name: ETHANE / Chamber humidity: 100 % / Chamber temperature: 278 K / Instrument: FEI VITROBOT MARK IV |
- Electron microscopy
Electron microscopy
| Microscope | FEI TALOS ARCTICA |
|---|---|
| Temperature | Min: 70.0 K / Max: 100.0 K |
| Specialist optics | Energy filter - Name: GIF Bioquantum |
| Image recording | Film or detector model: GATAN K3 (6k x 4k) / Number grids imaged: 1200 / Number real images: 1200 / Average exposure time: 2.5 sec. / Average electron dose: 50.0 e/Å2 |
| Electron beam | Acceleration voltage: 200 kV / Electron source:  FIELD EMISSION GUN FIELD EMISSION GUN |
| Electron optics | C2 aperture diameter: 100.0 µm / Calibrated defocus max: 3.0 µm / Calibrated defocus min: 1.5 µm / Illumination mode: FLOOD BEAM / Imaging mode: DIFFRACTION / Cs: 2.7 mm / Nominal defocus max: 3.5 µm / Nominal defocus min: 2.5 µm / Nominal magnification: 67000 |
| Sample stage | Specimen holder model: FEI TITAN KRIOS AUTOGRID HOLDER / Cooling holder cryogen: NITROGEN |
| Experimental equipment |  Model: Talos Arctica / Image courtesy: FEI Company |
+ Image processing
Image processing
-Atomic model buiding 1
| Refinement | Protocol: RIGID BODY FIT |
|---|---|
| Output model |  PDB-8g9t: |
 Movie
Movie Controller
Controller








 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)