[日本語] English
 万見
万見- EMDB-28248: CryoEM characterization of a unique AAA+ BrxL phage restriction factor -
+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | 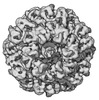 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM characterization of a unique AAA+ BrxL phage restriction factor | |||||||||
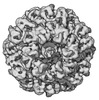
 マップデータ マップデータ | Walker B P45J59 full map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | phage restriction factor / AAA+ protein / ANTIMICROBIAL PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報ATP-dependent peptidase activity / protein catabolic process / serine-type endopeptidase activity / proteolysis / ATP binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Acinetobacter (バクテリア) / Acinetobacter (バクテリア) /  Acinetobacter sp. NEB 394 (バクテリア) / synthetic construct (人工物) Acinetobacter sp. NEB 394 (バクテリア) / synthetic construct (人工物) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.63 Å | |||||||||
 データ登録者 データ登録者 | Shen BW / Stoddard BL | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nucleic Acids Res / 年: 2023 ジャーナル: Nucleic Acids Res / 年: 2023タイトル: Structure, substrate binding and activity of a unique AAA+ protein: the BrxL phage restriction factor. 著者: Betty W Shen / Lindsey A Doyle / Rachel Werther / Abigail A Westburg / Daniel P Bies / Stephanie I Walter / Yvette A Luyten / Richard D Morgan / Barry L Stoddard / Brett K Kaiser /  要旨: Bacteriophage exclusion ('BREX') systems are multi-protein complexes encoded by a variety of bacteria and archaea that restrict phage by an unknown mechanism. One BREX factor, termed BrxL, has been ...Bacteriophage exclusion ('BREX') systems are multi-protein complexes encoded by a variety of bacteria and archaea that restrict phage by an unknown mechanism. One BREX factor, termed BrxL, has been noted to display sequence similarity to various AAA+ protein factors including Lon protease. In this study we describe multiple CryoEM structures of BrxL that demonstrate it to be a chambered, ATP-dependent DNA binding protein. The largest BrxL assemblage corresponds to a dimer of heptamers in the absence of bound DNA, versus a dimer of hexamers when DNA is bound in its central pore. The protein displays DNA-dependent ATPase activity, and ATP binding promotes assembly of the complex on DNA. Point mutations within several regions of the protein-DNA complex alter one or more in vitro behaviors and activities, including ATPase activity and ATP-dependent association with DNA. However, only the disruption of the ATPase active site fully eliminates phage restriction, indicating that other mutations can still complement BrxL function within the context of an otherwise intact BREX system. BrxL displays significant structural homology to MCM subunits (the replicative helicase in archaea and eukaryotes), implying that it and other BREX factors may collaborate to disrupt initiation of phage DNA replication. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_28248.map.gz emd_28248.map.gz | 89 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-28248-v30.xml emd-28248-v30.xml emd-28248.xml emd-28248.xml | 19.6 KB 19.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_28248.png emd_28248.png | 141 KB | ||
| Filedesc metadata |  emd-28248.cif.gz emd-28248.cif.gz | 6.8 KB | ||
| その他 |  emd_28248_half_map_1.map.gz emd_28248_half_map_1.map.gz emd_28248_half_map_2.map.gz emd_28248_half_map_2.map.gz | 165.2 MB 165.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-28248 http://ftp.pdbj.org/pub/emdb/structures/EMD-28248 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28248 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28248 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_28248_validation.pdf.gz emd_28248_validation.pdf.gz | 1010.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_28248_full_validation.pdf.gz emd_28248_full_validation.pdf.gz | 1010.5 KB | 表示 | |
| XML形式データ |  emd_28248_validation.xml.gz emd_28248_validation.xml.gz | 15.1 KB | 表示 | |
| CIF形式データ |  emd_28248_validation.cif.gz emd_28248_validation.cif.gz | 17.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28248 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28248 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28248 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28248 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_28248.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_28248.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Walker B P45J59 full map | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.0694 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: P45J59 half map A
| ファイル | emd_28248_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | P45J59 half map A | ||||||||||||
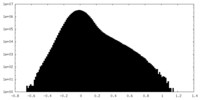
| 投影像・断面図 |
| ||||||||||||
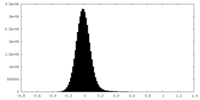
| 密度ヒストグラム |
-ハーフマップ: P45J59 half mapB
| ファイル | emd_28248_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | P45J59 half mapB | ||||||||||||
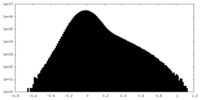
| 投影像・断面図 |
| ||||||||||||
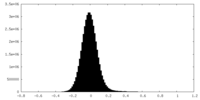
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : complex of walkerB mutant of BrxL with random sequence of DNA fra...
| 全体 | 名称: complex of walkerB mutant of BrxL with random sequence of DNA fragments |
|---|---|
| 要素 |
|
-超分子 #1: complex of walkerB mutant of BrxL with random sequence of DNA fra...
| 超分子 | 名称: complex of walkerB mutant of BrxL with random sequence of DNA fragments タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: 12 strands of KrxL E280Q mutant bound to short random sequence DNA fragments. |
|---|---|
| 由来(天然) | 生物種:  Acinetobacter (バクテリア) Acinetobacter (バクテリア) |
| 分子量 | 理論値: 950 KDa |
-分子 #1: Protease Lon-related BREX system protein BrxL
| 分子 | 名称: Protease Lon-related BREX system protein BrxL / タイプ: protein_or_peptide / ID: 1 / コピー数: 12 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Acinetobacter sp. NEB 394 (バクテリア) Acinetobacter sp. NEB 394 (バクテリア) |
| 分子量 | 理論値: 75.702555 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MESANDKELD QLLNEHFAGR VVRKDLTKLI KEGANVPVYV LEYLLGMYCA SDDPEIIEQG LRNVKTVLAE NYVRPDEAEK VKSLVRERG SYKVIDRVTV KLNERKDKYE ASFSNLGIKD AEISAGIVKE YEKLLVGGIW VIATLSYYFE EGQTSSPFGV S LLKPIQMP ...文字列: MESANDKELD QLLNEHFAGR VVRKDLTKLI KEGANVPVYV LEYLLGMYCA SDDPEIIEQG LRNVKTVLAE NYVRPDEAEK VKSLVRERG SYKVIDRVTV KLNERKDKYE ASFSNLGIKD AEISAGIVKE YEKLLVGGIW VIATLSYYFE EGQTSSPFGV S LLKPIQMP NMNMDELFSG RAALSTDQWR ESLIRSIGME PASLKEDVQW HLLARMVPFV ENNYNVCELG PRGTGKSHIY KE CSPNSIL VSGGQTTVAN LFYNMSSRRI GLVGLWDVVA FDQVAGISFK DKDGVQIMKD YMASGSFARG REQMEASASM VFV GNINQS VESLVKTSHL LAPFPEAMID SAFFDRFHAY IPGWEIPKMR PEFFTNRYGL IVDYLAEFFR EMRKRSFADS IEKY FKLGN NLNQRDVIAV RKTVSGLMKL LYPHGQFNKE DVRQCLEYAL QVRRRVKEQL KKIGGMEFYD VHFSYIDNDT LEEHF VSVK EQGGGGLIPE GPAKPGFLYT IGLSNKGMPG LYRLELQVTK GSGKLATSGL WNSSSAKEQV KIAFDYFKAN ASRISG GSK VMEHDFHLHV VELQNTGPLS HLALPSLVAF ASGLLGRSVQ SQMVVLGDMS LGGSVTPVES IAECLQVAFD AGAKKVA LP MSSAADIPTI PVELFTKFQT SFYADPVDAV FKGLGVD UniProtKB: Protease Lon-related BREX system protein BrxL |
-分子 #2: DNA (64-MER)
| 分子 | 名称: DNA (64-MER) / タイプ: dna / ID: 2 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 19.689639 KDa |
| 配列 | 文字列: (DA)(DC)(DG)(DC)(DG)(DC)(DT)(DA)(DC)(DA) (DC)(DT)(DA)(DA)(DA)(DA)(DG)(DG)(DG)(DC) (DC)(DC)(DT)(DT)(DA)(DA)(DT)(DT)(DC) (DG)(DA)(DT)(DC)(DG)(DA)(DC)(DT)(DA)(DA) (DG) (DA)(DA)(DA)(DG)(DG) ...文字列: (DA)(DC)(DG)(DC)(DG)(DC)(DT)(DA)(DC)(DA) (DC)(DT)(DA)(DA)(DA)(DA)(DG)(DG)(DG)(DC) (DC)(DC)(DT)(DT)(DA)(DA)(DT)(DT)(DC) (DG)(DA)(DT)(DC)(DG)(DA)(DC)(DT)(DA)(DA) (DG) (DA)(DA)(DA)(DG)(DG)(DG)(DC)(DC) (DC)(DT)(DT)(DT)(DA)(DT)(DC)(DG)(DA)(DT) (DC)(DG) (DA)(DC)(DT)(DG) |
-分子 #3: DNA (63-MER)
| 分子 | 名称: DNA (63-MER) / タイプ: dna / ID: 3 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 19.485461 KDa |
| 配列 | 文字列: (DC)(DA)(DG)(DT)(DC)(DG)(DA)(DT)(DC)(DG) (DA)(DT)(DA)(DA)(DA)(DG)(DG)(DG)(DG)(DC) (DC)(DC)(DT)(DT)(DT)(DC)(DT)(DT)(DA) (DG)(DT)(DC)(DG)(DA)(DT)(DC)(DG)(DA)(DA) (DT) (DT)(DA)(DA)(DG)(DG) ...文字列: (DC)(DA)(DG)(DT)(DC)(DG)(DA)(DT)(DC)(DG) (DA)(DT)(DA)(DA)(DA)(DG)(DG)(DG)(DG)(DC) (DC)(DC)(DT)(DT)(DT)(DC)(DT)(DT)(DA) (DG)(DT)(DC)(DG)(DA)(DT)(DC)(DG)(DA)(DA) (DT) (DT)(DA)(DA)(DG)(DG)(DG)(DC)(DC) (DC)(DT)(DT)(DT)(DA)(DG)(DT)(DG)(DT)(DA) (DG)(DC) (DG)(DC)(DG) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 / 詳細: 20 mM TrisHCl, 150 mM NaCl |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 15 sec. / 前処理 - 雰囲気: AIR / 詳細: 40 |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 275 K |
| 詳細 | The sample was mono-disperse |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 温度 | 最高: 70.0 K |
| 撮影 | フィルム・検出器のモデル: DIRECT ELECTRON DE-10 (5k x 4k) 検出モード: SUPER-RESOLUTION / デジタル化 - 画像ごとのフレーム数: 1-49 / 撮影したグリッド数: 2 / 平均露光時間: 2.0 sec. / 平均電子線量: 30.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 最小 デフォーカス(補正後): 12.1 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 5.0 µm / 最小 デフォーカス(公称値): 1.2 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー






 Z
Z Y
Y X
X