+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | The Cryo-EM structure of Drosophila Cryptochrome in complex with Timeless | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Flavoprotein / Nuclear import / Light-sensor / Armadillo-repeat protein / CIRCADIAN CLOCK PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Nuclear import of PER and TIM / Dephosphorylation of TIM / circadian regulation of heart rate / eclosion rhythm / negative phototaxis / Transcription repression by PER and activation by PDP1 / Dephosphorylation of PER / UV-A, blue light phototransduction / photoperiodism / regulation of circadian sleep/wake cycle ...Nuclear import of PER and TIM / Dephosphorylation of TIM / circadian regulation of heart rate / eclosion rhythm / negative phototaxis / Transcription repression by PER and activation by PDP1 / Dephosphorylation of PER / UV-A, blue light phototransduction / photoperiodism / regulation of circadian sleep/wake cycle / magnetoreception / detection of light stimulus involved in magnetoreception / gravitaxis / Phosphorylation of PER and TIM / copulation / Degradation of CRY / Degradation of TIM / circadian temperature homeostasis / rhythmic behavior / blue light signaling pathway / regulation of protein import into nucleus / response to magnetism / methylated-DNA-[protein]-cysteine S-methyltransferase / methylated-DNA-[protein]-cysteine S-methyltransferase activity / replication fork arrest / response to blue light / regulation of circadian sleep/wake cycle, sleep / cellular response to light stimulus / blue light photoreceptor activity / entrainment of circadian clock / regulation of phagocytosis / DNA replication checkpoint signaling / replication fork protection complex / circadian behavior / mating behavior / entrainment of circadian clock by photoperiod / locomotor rhythm / phototransduction / photoreceptor activity / response to light stimulus / positive regulation of phagocytosis / FAD binding / circadian regulation of gene expression / regulation of circadian rhythm / circadian rhythm / flavin adenine dinucleotide binding / methylation / DNA repair / negative regulation of DNA-templated transcription / perinuclear region of cytoplasm / DNA binding / nucleoplasm / nucleus / metal ion binding / cytosol / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.3 Å | |||||||||
 データ登録者 データ登録者 | Feng S / Lin C / DeOliveira CC / Crane BR | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2023 ジャーナル: Nature / 年: 2023タイトル: Cryptochrome-Timeless structure reveals circadian clock timing mechanisms. 著者: Changfan Lin / Shi Feng / Cristina C DeOliveira / Brian R Crane /  要旨: Circadian rhythms influence many behaviours and diseases. They arise from oscillations in gene expression caused by repressor proteins that directly inhibit transcription of their own genes. The fly ...Circadian rhythms influence many behaviours and diseases. They arise from oscillations in gene expression caused by repressor proteins that directly inhibit transcription of their own genes. The fly circadian clock offers a valuable model for studying these processes, wherein Timeless (Tim) plays a critical role in mediating nuclear entry of the transcriptional repressor Period (Per) and the photoreceptor Cryptochrome (Cry) entrains the clock by triggering Tim degradation in light. Here, through cryogenic electron microscopy of the Cry-Tim complex, we show how a light-sensing cryptochrome recognizes its target. Cry engages a continuous core of amino-terminal Tim armadillo repeats, resembling how photolyases recognize damaged DNA, and binds a C-terminal Tim helix, reminiscent of the interactions between light-insensitive cryptochromes and their partners in mammals. The structure highlights how the Cry flavin cofactor undergoes conformational changes that couple to large-scale rearrangements at the molecular interface, and how a phosphorylated segment in Tim may impact clock period by regulating the binding of Importin-α and the nuclear import of Tim-Per. Moreover, the structure reveals that the N terminus of Tim inserts into the restructured Cry pocket to replace the autoinhibitory C-terminal tail released by light, thereby providing a possible explanation for how the long-short Tim polymorphism adapts flies to different climates. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_27335.map.gz emd_27335.map.gz | 95.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-27335-v30.xml emd-27335-v30.xml emd-27335.xml emd-27335.xml | 19.1 KB 19.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_27335_fsc.xml emd_27335_fsc.xml | 11.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_27335.png emd_27335.png | 61.3 KB | ||
| Filedesc metadata |  emd-27335.cif.gz emd-27335.cif.gz | 7 KB | ||
| その他 |  emd_27335_additional_1.map.gz emd_27335_additional_1.map.gz emd_27335_half_map_1.map.gz emd_27335_half_map_1.map.gz emd_27335_half_map_2.map.gz emd_27335_half_map_2.map.gz | 51.8 MB 95.5 MB 95.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-27335 http://ftp.pdbj.org/pub/emdb/structures/EMD-27335 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27335 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27335 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_27335_validation.pdf.gz emd_27335_validation.pdf.gz | 857 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_27335_full_validation.pdf.gz emd_27335_full_validation.pdf.gz | 856.6 KB | 表示 | |
| XML形式データ |  emd_27335_validation.xml.gz emd_27335_validation.xml.gz | 18.7 KB | 表示 | |
| CIF形式データ |  emd_27335_validation.cif.gz emd_27335_validation.cif.gz | 23.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27335 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27335 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27335 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27335 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8dd7MC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_27335.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_27335.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.032 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: #1
| ファイル | emd_27335_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
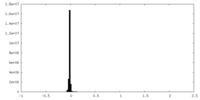
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_27335_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
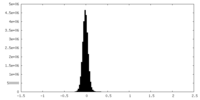
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_27335_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
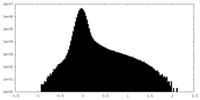
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Complex of Drosophila Cryptochrome and Timeless
| 全体 | 名称: Complex of Drosophila Cryptochrome and Timeless |
|---|---|
| 要素 |
|
-超分子 #1: Complex of Drosophila Cryptochrome and Timeless
| 超分子 | 名称: Complex of Drosophila Cryptochrome and Timeless / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 264 KDa |
-分子 #1: Methylated-DNA--protein-cysteine methyltransferase,Cryptochrome-1...
| 分子 | 名称: Methylated-DNA--protein-cysteine methyltransferase,Cryptochrome-1 fusion タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 83.838617 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MEQKLISEED LGGGEQKLIS EEDLGGGEQK LISEEDLGGG MDKDCEMKRT TLDSPLGKLE LSGCEQGLHR IIFLGKGTSA ADAVEVPAP AAVLGGPEPL IQATAWLNAY FHQPEAIEEF PVPALHHPVF QQESFTRQVL WKLLKVVKFG EVISESHLAA L VGNPAATA ...文字列: MEQKLISEED LGGGEQKLIS EEDLGGGEQK LISEEDLGGG MDKDCEMKRT TLDSPLGKLE LSGCEQGLHR IIFLGKGTSA ADAVEVPAP AAVLGGPEPL IQATAWLNAY FHQPEAIEEF PVPALHHPVF QQESFTRQVL WKLLKVVKFG EVISESHLAA L VGNPAATA AVNTALDGNP VPILIPCHRV VQGDSDVGPY LGGLAVKEWL LAHEGHRLGK PGLGGGSGMA TRGANVIWFR HG LRLHDNP ALLAALADKD QGIALIPVFI FDGESAGTKN VGYNRMRFLL DSLQDIDDQL QAATDGRGRL LVFEGEPAYI FRR LHEQVR LHRICIEQDC EPIWNERDES IRSLCRELNI DFVEKVSHTL WDPQLVIETN GGIPPLTYQM FLHTVQIIGL PPRP TADAR LEDATFVELD PEFCRSLKLF EQLPTPEHFN VYGDNMGFLA KINWRGGETQ ALLLLDERLK VEQHAFERGF YLPNQ ALPN IHDSPKSMSA HLRFGCLSVR RFYWSVHDLF KNVQLRACVR GVQMTGGAHI TGQLIWREYF YTMSVNNPNY DRMEGN DIC LSIPWAKPNE NLLQSWRLGQ TGFPLIDGAM RQLLAEGWLH HTLRNTVATF LTRGGLWQSW EHGLQHFLKY LLDADWS VC AGNWMWVSSS AFERLLDSSL VTCPVALAKR LDPDGTYIKQ YVPELMNVPK EFVHEPWRMS AEQQEQYECL IGVHYPER I IDLSMAVKRN MLAMKSLRNS LITPP UniProtKB: Methylated-DNA--protein-cysteine methyltransferase, Cryptochrome-1 |
-分子 #2: Protein timeless,Methylated-DNA--protein-cysteine methyltransfera...
| 分子 | 名称: Protein timeless,Methylated-DNA--protein-cysteine methyltransferase fusion タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO EC番号: methylated-DNA-[protein]-cysteine S-methyltransferase |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 179.996109 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MDWLLATPQL YSAFSSLGCL EGDTYVVNPN ALAILEEINY KLTYEDQTLR TFRRAIGFGQ NVRSDLIPLL ENAKDDAVLE SVIRILVNL TVPVECLFSV DVMYRTDVGR HTIFELNKLL YTSKEAFTEA RSTKSVVEYM KHILESDPKL SPHKCDQINN C LLLLRNIL ...文字列: MDWLLATPQL YSAFSSLGCL EGDTYVVNPN ALAILEEINY KLTYEDQTLR TFRRAIGFGQ NVRSDLIPLL ENAKDDAVLE SVIRILVNL TVPVECLFSV DVMYRTDVGR HTIFELNKLL YTSKEAFTEA RSTKSVVEYM KHILESDPKL SPHKCDQINN C LLLLRNIL HIPETHAHCV MPMMQSMPHG ISMQNTILWN LFIQSIDKLL LYLMTCPQRA FWGVTMVQLI ALIYKDQHVS TL QKLLSLW FEASLSESSE DNESNTSPPK QGSGDSSPML TSDPTSDSSD NGSNGRGMGG GMREGTAATL QEVSRKGQEY QNA MARVPA DKPDGSEEAS DMTGNDSEQP GSPEQSQPAG ESMDDGDYED QRHRQLNEHG EEDEDEDEVE EEEYLQLGPA SEPL NLTQQ PADKVNNTTN PTSSAPQGCL GNEPFKPPPP LPVRASTSAH AQMQKFNESS YASHVSAVKL GQKSPHAGQL QLTKG KCCP QKRECPSSQS ELSDCGYGTQ VENQESISTS SNDDDGPQGK PQHQKPPCNT KPRNKPRTIM SPMDKKELRR KKLVKR SKS SLINMKGLVQ HTPTDDDISN LLKEFTVDFL LKGYSYLVEE LHMQLLSNAK VPIDTSHFFW LVTYFLKFAA QLELDME HI DTILTYDVLS YLTYEGVSLC EQLELNARQE GSDLKPYLRR MHLVVTAIRE FLQAIDTYNK VTHLNEDDKA HLRQLQLQ I SEMSDLRCLF VLLLRRFNPS IHSKQYLQDL VVTNHILLLI LDSSAKLGGC QTIRLSEHIT QFATLEVMHY YGILLEDFN NNGEFVNDCI FTMMHHIGGD LGQIGVLFQP IILKTYSRIW EADYELCDDW SDLIEYVIHK FMNTPPKSPL TIPTTSLTEM TKEHNQEHT VCSWSQEEMD TLYWYYVQSK KNNDIVGKIV KLFSNNGNKL KTRISIIQQL LQQDIITLLE YDDLMKFEDA E YQRTLLTT PTSATTESGI EIKECAYGKP SDDVQILLDL IIKENKAQHL LWLQRILIEC CFVKLTLRSG LKVPEGDHIM EP VAYHCIC KQKSIPVVQW NNEQSTTMLY QPFVLLLHKL GIQLPADAGS IFARIPDYWT PETMYGLAKK LGPLDKLNLK FDA SELEDA TASSPSRYHH TGPRNSLSSV SSLDVDLGDT EELALIPEVD AAVEKAHAMA STPSPSEIFA VPKTKHCNSI IRYT PDPTP PVPNWLQLVM RSKCNHRTGP SGDPSDCIGS SSTTVDDEGF GKSISAATSQ AASTSMSTVN PTTTLSLNML NTFMG SHNE NSSSSGCGGT VSSLSMVALM STGAAGGGGN TSGLEMDVDA SMKSSFERLE VNGSHFSRAN NLDQEYSAMV ASVYEK EKE LNSDNVSLAS DLTRMYVSDE DDRLERTEIR VPHYHLEGGS GMDKDCEMKR TTLDSPLGKL ELSGCEQGLH RIIFLGK GT SAADAVEVPA PAAVLGGPEP LMQATAWLNA YFHQPEAIEE FPVPALHHPV FQQESFTRQV LWKLLKVVKF GEVISYSH L AALAGNPAAT AAVKTALSGN PVPILIPCHR VVQGDLDVGG YEGGLAVKEW LLAHEGHRLG KPGLGGGGYP YDVPDYART GGGSGSRLEE ELRRRLTE UniProtKB: Protein timeless, Methylated-DNA--protein-cysteine methyltransferase |
-分子 #3: FLAVIN-ADENINE DINUCLEOTIDE
| 分子 | 名称: FLAVIN-ADENINE DINUCLEOTIDE / タイプ: ligand / ID: 3 / コピー数: 1 / 式: FAD |
|---|---|
| 分子量 | 理論値: 785.55 Da |
| Chemical component information |  ChemComp-FAD: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE-PROPANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 53.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー





 X (Sec.)
X (Sec.) Y (Row.)
Y (Row.) Z (Col.)
Z (Col.)