+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | 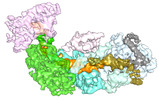 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Apo gRAMP | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | CRISPR / GRAMP / RNA BINDING PROTEIN / RNA BINDING PROTEIN-RNA complex | |||||||||
| 機能・相同性 | CRISPR type III-associated protein / RAMP superfamily / defense response to virus / RNA binding / RAMP superfamily protein 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Candidatus Scalindua brodae (バクテリア) Candidatus Scalindua brodae (バクテリア) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.8 Å | |||||||||
 データ登録者 データ登録者 | Hu C / Nam KH / Schuler G / Ke A | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Science / 年: 2022 ジャーナル: Science / 年: 2022タイトル: Craspase is a CRISPR RNA-guided, RNA-activated protease. 著者: Chunyi Hu / Sam P B van Beljouw / Ki Hyun Nam / Gabriel Schuler / Fran Ding / Yanru Cui / Alicia Rodríguez-Molina / Anna C Haagsma / Menno Valk / Martin Pabst / Stan J J Brouns / Ailong Ke /    要旨: The CRISPR-Cas type III-E RNA-targeting effector complex gRAMP/Cas7-11 is associated with a caspase-like protein (TPR-CHAT/Csx29) to form Craspase (CRISPR-guided caspase). Here, we use cryo-electron ...The CRISPR-Cas type III-E RNA-targeting effector complex gRAMP/Cas7-11 is associated with a caspase-like protein (TPR-CHAT/Csx29) to form Craspase (CRISPR-guided caspase). Here, we use cryo-electron microscopy snapshots of Craspase to explain its target RNA cleavage and protease activation mechanisms. Target-guide pairing extending into the 5' region of the guide RNA displaces a gating loop in gRAMP, which triggers an extensive conformational relay that allosterically aligns the protease catalytic dyad and opens an amino acid side-chain-binding pocket. We further define Csx30 as the endogenous protein substrate that is site-specifically proteolyzed by RNA-activated Craspase. This protease activity is switched off by target RNA cleavage by gRAMP and is not activated by RNA targets containing a matching protospacer flanking sequence. We thus conclude that Craspase is a target RNA-activated protease with self-regulatory capacity. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
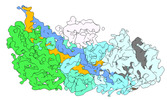
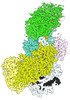
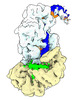
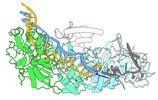
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_27257.map.gz emd_27257.map.gz | 28.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-27257-v30.xml emd-27257-v30.xml emd-27257.xml emd-27257.xml | 18.1 KB 18.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_27257.png emd_27257.png | 114.7 KB | ||
| その他 |  emd_27257_additional_1.map.gz emd_27257_additional_1.map.gz emd_27257_half_map_1.map.gz emd_27257_half_map_1.map.gz emd_27257_half_map_2.map.gz emd_27257_half_map_2.map.gz | 56.5 MB 55.3 MB 55.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-27257 http://ftp.pdbj.org/pub/emdb/structures/EMD-27257 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27257 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27257 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_27257_validation.pdf.gz emd_27257_validation.pdf.gz | 964.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_27257_full_validation.pdf.gz emd_27257_full_validation.pdf.gz | 963.8 KB | 表示 | |
| XML形式データ |  emd_27257_validation.xml.gz emd_27257_validation.xml.gz | 12.4 KB | 表示 | |
| CIF形式データ |  emd_27257_validation.cif.gz emd_27257_validation.cif.gz | 14.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27257 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27257 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27257 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27257 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8d97MC  8d8nC  8d9eC  8d9fC  8d9gC  8d9hC  8d9iC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_27257.map.gz / 形式: CCP4 / 大きさ: 59.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_27257.map.gz / 形式: CCP4 / 大きさ: 59.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.284 Å | ||||||||||||||||||||
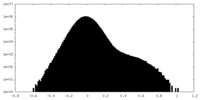
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: #1
| ファイル | emd_27257_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
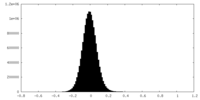
| 投影像・断面図 |
| ||||||||||||
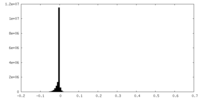
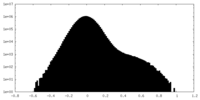
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_27257_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
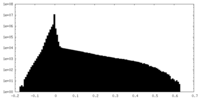
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_27257_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
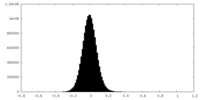
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : APO gRAMP
| 全体 | 名称: APO gRAMP |
|---|---|
| 要素 |
|
-超分子 #1: APO gRAMP
| 超分子 | 名称: APO gRAMP / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:  Candidatus Scalindua brodae (バクテリア) Candidatus Scalindua brodae (バクテリア) |
-分子 #1: RAMP superfamily protein
| 分子 | 名称: RAMP superfamily protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Candidatus Scalindua brodae (バクテリア) Candidatus Scalindua brodae (バクテリア) |
| 分子量 | 理論値: 184.351406 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MNITVELTFF EPYRLVEWFD WDARKKSHSA MRGQAFAQWT WKGKGRTAGK SFITGTLVRS AVIKAVEELL SLNNGKWEGV PCCNGSFQT DESKGKKPSF LRKRHTLQWQ ANNKNICDKE EACPFCILLG RFDNAGKVHE RNKDYDIHFS NFDLDHDLRL V DIASGRIL ...文字列: MNITVELTFF EPYRLVEWFD WDARKKSHSA MRGQAFAQWT WKGKGRTAGK SFITGTLVRS AVIKAVEELL SLNNGKWEGV PCCNGSFQT DESKGKKPSF LRKRHTLQWQ ANNKNICDKE EACPFCILLG RFDNAGKVHE RNKDYDIHFS NFDLDHDLRL V DIASGRIL NRVDFDTGKA KDYFRTWEAD YETYGTYTGR ITLRNEHAKK LLLASLGFVD KLCGALCRIE VIKSEDHNDE LR KQAEVIV EAFKQNDKLE KIRILADAIR TLRLHGEGVI EKDELPDGKE ERDKGHHLWD IKVQGTALRT KLKELWQSNK DIG WRKFTE MLGSNLYLIY KKETGGVSTR FRILGDTEYY SKAHDSEGSD LFIPVTPPEG IETKEWIIVG RLKAATPFYF GVQQ PSDSI PGKEKKSEDS LVINEHTSFN ILLDKENRYR IPRSALRGAL RRDLRTAFGS GCNVSLGGQI LCNCKVCIEM RRITL KDSV SDFSEPPEIR YRIAKNPGTA TVEDGSLFDI EVGPEGLTFP FVLRYRGHKF PEQLSSVIRY WEENDGKNGM AWLGGL DST GKGRFALKDI KIFEWDLNQK INEYIKERGM RGKEKELLEM GESSLPDGLI PYKFFEEREC LFPYKENLKP QWSEVQY TI EVGSPLLTAD TISALTEPGN RDAIAYKKRV YNDGNNAIEP EPRFAVKSET HRGIFRTAVG RRTGDLGKED HEDCTCDM C IIFGNEHESS KIRFEDLELI NGNEFEKLEK HIDHVAIDRF TGGALDKAKF DTYPLAGSPK KPLKLKGRFW IKKGFSGDH KLLITTALSD IRDGLYPLGS KGGVGYGWVA GISIDDNVPD DFKEMINKTY VHPGHQSPKQ DHKNKNIYYP HYFLDSGSKV YREKDIITH EEFTEELLSG KINCKLETLT PLIIPDTSDE NGLKLQGNKP GHKNYKFFNI NGELMIPGSE LRGMLRTHFE A LTKSCFAI FGETLSWRMN ADEKDYKIDS NSIRKMESQR NPKYRIPDEL QKELRNSGNG LFNRLYTSER RFWSDVSNKF EN SIDYKRE ILRCAGRPKN YKGGIIRQRK DSLMAEELKV HRLPLYDNFD IPDSAYKAND HCRKSATCST SRGCRERFTC GIK VRDKNR VFLNAANNNR QYLNNIKKSN HDLYLQYLKG EKKIRFNSKV ITGSERSPID VIAELNERGR QTGFIKLSGL NNSN KSQGN TGTTFNSGWD RFELNILLDD LETRPSKSDY PRPRLLFTKD QYEYNITKRC ERVFEIDKGN KTGYPVDDQI KKNYE DILD SYDGIKDQEV AERFDTFTRG SKLKVGDLVY FHIDGDNKID SLIPVRGKLD KALHPCTGLS DGLCPGCHLF GTTDYK GRV KFGFAKYENG PEWLITRGNN PERSLTLGVL ESPRPAFSIP DDESEIPGRK FYLHHNGWRI IRQKQLEIRE TVQPERN VT TEVMDKGNVF SFDVRFENLR EWELGLLLQS LDPGKNIAHK LGKGKPYGFG SVKIKIDSLH TFKIIKRVPQ SDIREYIN K GYQKLIEWSL PQWHVIPHID KLYKLLWVPF LNDSKLEPDV RYPVLNYTYK KLGDKDNLPY KTRVKGLTTP WSPWNPFQV |
-分子 #2: RNA (42-MER)
| 分子 | 名称: RNA (42-MER) / タイプ: rna / ID: 2 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:  Candidatus Scalindua brodae (バクテリア) Candidatus Scalindua brodae (バクテリア) |
| 分子量 | 理論値: 13.295887 KDa |
| 配列 | 文字列: UUAAUGUCAC GGUACCCAAU UUUCUGCCCC GGACUCCACG GC |
-分子 #3: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 3 / コピー数: 4 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 59.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: NONE |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.8 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 21741 |
| 初期 角度割当 | タイプ: NOT APPLICABLE |
| 最終 角度割当 | タイプ: NOT APPLICABLE |
 ムービー
ムービー コントローラー
コントローラー





 Z
Z Y
Y X
X