+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | IscB and wRNA bound to Target DNA (locked state) | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | CRISPR / IscB / HEARO RNA / omega RNA / RNA BINDING PROTEIN-RNA-DNA complex | |||||||||
| 生物種 | synthetic construct (人工物) / unidentified (未定義) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.1 Å | |||||||||
 データ登録者 データ登録者 | Schuler GA / Hu C / Ke A | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Science / 年: 2022 ジャーナル: Science / 年: 2022タイトル: Structural basis for RNA-guided DNA cleavage by IscB-ωRNA and mechanistic comparison with Cas9. 著者: Gabriel Schuler / Chunyi Hu / Ailong Ke /  要旨: Class 2 CRISPR effectors Cas9 and Cas12 may have evolved from nucleases in IS200/IS605 transposons. IscB is about two-fifths the size of Cas9 but shares a similar domain organization. The associated ...Class 2 CRISPR effectors Cas9 and Cas12 may have evolved from nucleases in IS200/IS605 transposons. IscB is about two-fifths the size of Cas9 but shares a similar domain organization. The associated ωRNA plays the combined role of CRISPR RNA (crRNA) and trans-activating CRISPR RNA tracrRNA) to guide double-stranded DNA (dsDNA) cleavage. Here we report a 2.78-angstrom cryo-electron microscopy structure of IscB-ωRNA bound to a dsDNA target, revealing the architectural and mechanistic similarities between IscB and Cas9 ribonucleoproteins. Target-adjacent motif recognition, R-loop formation, and DNA cleavage mechanisms are explained at high resolution. ωRNA plays the equivalent function of REC domains in Cas9 and contacts the RNA-DNA heteroduplex. The IscB-specific PLMP domain is dispensable for RNA-guided DNA cleavage. The transition from ancestral IscB to Cas9 involved dwarfing the ωRNA and introducing protein domain replacements. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_26994.map.gz emd_26994.map.gz | 79.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-26994-v30.xml emd-26994-v30.xml emd-26994.xml emd-26994.xml | 17.8 KB 17.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_26994.png emd_26994.png | 101.2 KB | ||
| Filedesc metadata |  emd-26994.cif.gz emd-26994.cif.gz | 6.2 KB | ||
| その他 |  emd_26994_half_map_1.map.gz emd_26994_half_map_1.map.gz emd_26994_half_map_2.map.gz emd_26994_half_map_2.map.gz | 77.8 MB 77.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-26994 http://ftp.pdbj.org/pub/emdb/structures/EMD-26994 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26994 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26994 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_26994.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_26994.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.07 Å | ||||||||||||||||||||||||||||||||||||
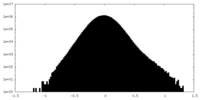
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_26994_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
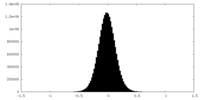
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_26994_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
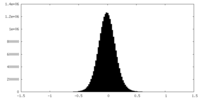
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Cryo-EM structure of IscB in complex with RNA and target DNA
| 全体 | 名称: Cryo-EM structure of IscB in complex with RNA and target DNA |
|---|---|
| 要素 |
|
-超分子 #1: Cryo-EM structure of IscB in complex with RNA and target DNA
| 超分子 | 名称: Cryo-EM structure of IscB in complex with RNA and target DNA タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 190 kDa/nm |
-分子 #1: IscB
| 分子 | 名称: IscB / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 56.81968 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MMAVVYVISK SGKPLMPTTR CGHVRILLKE GKARVVERKP FTIQLTYESA EETQPLVLGI DPGRTNIGMS VVTESGESVF NAQIETRNK DVPKLMKDRK QYRMAHRRLK RRCKRRRRAK AAGTAFEEGE KQRLLPGCFK PITCKSIRNK EARFNNRKRP V GWLTPTAN ...文字列: MMAVVYVISK SGKPLMPTTR CGHVRILLKE GKARVVERKP FTIQLTYESA EETQPLVLGI DPGRTNIGMS VVTESGESVF NAQIETRNK DVPKLMKDRK QYRMAHRRLK RRCKRRRRAK AAGTAFEEGE KQRLLPGCFK PITCKSIRNK EARFNNRKRP V GWLTPTAN HLLVTHLNVV KKVQKILPVA KVVLELNRFS FMAMNNPKVQ RWQYQRGPLY GKGSVEEAVS MQQDGHCLFC KH GIDHYHH VVPRRKNGSE TLENRVGLCE EHHRLVHTDK EWEANLASKK SGMNKKYHAL SVLNQIIPYL ADQLADMFPG NFC VTSGQD TYLFREEHGI PKDHYLDAYC IACSALTDAK KVSSPKGRPY MVHQFRRHDR QACHKANLNR SYYMGGKLVA TNRH KAMDQ KTDSLEEYRA AHSAADVSKL TVKHPSAQYK DMSRIMPGSI LVSGEGKLFT LSRSEGRNKG QVNYFVSTEG IKYWA RKCQ YLRNNGGLQI YV |
-分子 #2: RNA (222-MER)
| 分子 | 名称: RNA (222-MER) / タイプ: rna / ID: 2 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種: unidentified (未定義) |
| 分子量 | 理論値: 71.877797 KDa |
| 配列 | 文字列: AAAAGAGUGA ACGAGAGGCU CUUCCAACUU UAUGGUUGCG ACCGUAGGUU GAAAGAGCAC AGGCUGAGAC AUUCGUAAGG CCGAAAGAC CGGACGCACC CUGGGAUUUC CCCAGUCCCC GGAACUGCAU AGCGGAUGCC AGUUGAUGGA GCAAUCUAUC A GAUAAGCC ...文字列: AAAAGAGUGA ACGAGAGGCU CUUCCAACUU UAUGGUUGCG ACCGUAGGUU GAAAGAGCAC AGGCUGAGAC AUUCGUAAGG CCGAAAGAC CGGACGCACC CUGGGAUUUC CCCAGUCCCC GGAACUGCAU AGCGGAUGCC AGUUGAUGGA GCAAUCUAUC A GAUAAGCC AGGGGGAACA AUCACCUCUC UGUAUCAGAG AGAGUUUUAC AAAAGGAGGA ACGG |
-分子 #3: DNA target strand
| 分子 | 名称: DNA target strand / タイプ: dna / ID: 3 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 18.305646 KDa |
| 配列 | 文字列: (DG)(DC)(DC)(DA)(DC)(DG)(DG)(DG)(DC)(DT) (DG)(DA)(DC)(DC)(DT)(DC)(DG)(DA)(DC)(DT) (DT)(DC)(DT)(DA)(DG)(DT)(DC)(DT)(DC) (DG)(DT)(DT)(DC)(DA)(DC)(DT)(DC)(DT)(DT) (DT) (DT)(DG)(DC)(DC)(DG) ...文字列: (DG)(DC)(DC)(DA)(DC)(DG)(DG)(DG)(DC)(DT) (DG)(DA)(DC)(DC)(DT)(DC)(DG)(DA)(DC)(DT) (DT)(DC)(DT)(DA)(DG)(DT)(DC)(DT)(DC) (DG)(DT)(DT)(DC)(DA)(DC)(DT)(DC)(DT)(DT) (DT) (DT)(DG)(DC)(DC)(DG)(DT)(DA)(DC) (DC)(DC)(DT)(DC)(DG)(DT)(DG)(DG)(DG)(DG) (DC)(DC) |
-分子 #4: DNA non-target strand
| 分子 | 名称: DNA non-target strand / タイプ: dna / ID: 4 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: unidentified (未定義) |
| 分子量 | 理論値: 18.557873 KDa |
| 配列 | 文字列: (DC)(DG)(DC)(DC)(DC)(DC)(DA)(DC)(DG)(DA) (DG)(DG)(DG)(DT)(DA)(DC)(DG)(DG)(DC)(DA) (DA)(DA)(DA)(DG)(DA)(DG)(DT)(DT)(DT) (DT)(DT)(DT)(DT)(DT)(DA)(DC)(DT)(DA)(DG) (DA) (DA)(DG)(DT)(DC)(DG) ...文字列: (DC)(DG)(DC)(DC)(DC)(DC)(DA)(DC)(DG)(DA) (DG)(DG)(DG)(DT)(DA)(DC)(DG)(DG)(DC)(DA) (DA)(DA)(DA)(DG)(DA)(DG)(DT)(DT)(DT) (DT)(DT)(DT)(DT)(DT)(DA)(DC)(DT)(DA)(DG) (DA) (DA)(DG)(DT)(DC)(DG)(DA)(DG)(DG) (DT)(DC)(DA)(DG)(DC)(DC)(DC)(DG)(DT)(DG) (DG)(DC) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.5 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.25 構成要素:
| |||||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY / 支持フィルム - Film thickness: 100 | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV / 詳細: blot for 6.5 seconds before plunging. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: NONE |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.1 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 71831 |
| 初期 角度割当 | タイプ: NOT APPLICABLE |
| 最終 角度割当 | タイプ: NOT APPLICABLE |
 ムービー
ムービー コントローラー
コントローラー









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)