+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-23057 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | rFVIIIFc-VWF-XTEN (BIVV001) | |||||||||
 マップデータ マップデータ | Full map from 3D refinement | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Defective F8 accelerates dissociation of the A2 domain / Defective F8 binding to the cell membrane / Defective F8 secretion / Gamma carboxylation, hypusinylation, hydroxylation, and arylsulfatase activation / Defective F8 sulfation at Y1699 / Defective VWF binding to collagen type I / Enhanced cleavage of VWF variant by ADAMTS13 / Defective VWF cleavage by ADAMTS13 variant / Weibel-Palade body / Defective F8 binding to von Willebrand factor ...Defective F8 accelerates dissociation of the A2 domain / Defective F8 binding to the cell membrane / Defective F8 secretion / Gamma carboxylation, hypusinylation, hydroxylation, and arylsulfatase activation / Defective F8 sulfation at Y1699 / Defective VWF binding to collagen type I / Enhanced cleavage of VWF variant by ADAMTS13 / Defective VWF cleavage by ADAMTS13 variant / Weibel-Palade body / Defective F8 binding to von Willebrand factor / Enhanced binding of GP1BA variant to VWF multimer:collagen / Defective binding of VWF variant to GPIb:IX:V / hemostasis / blood coagulation, intrinsic pathway / platelet alpha granule / Platelet Adhesion to exposed collagen / Cargo concentration in the ER / Defective factor IX causes thrombophilia / Defective cofactor function of FVIIIa variant / Defective F9 variant does not activate FX / COPII-mediated vesicle transport / positive regulation of intracellular signal transduction / GP1b-IX-V activation signalling / p130Cas linkage to MAPK signaling for integrins / cell-substrate adhesion / COPII-coated ER to Golgi transport vesicle / Defective F8 cleavage by thrombin / Platelet Aggregation (Plug Formation) / immunoglobulin binding / GRB2:SOS provides linkage to MAPK signaling for Integrins / Integrin cell surface interactions / Common Pathway of Fibrin Clot Formation / collagen binding / Intrinsic Pathway of Fibrin Clot Formation / endoplasmic reticulum-Golgi intermediate compartment membrane / Integrin signaling / extracellular matrix / platelet alpha granule lumen / acute-phase response / Signaling by high-kinase activity BRAF mutants / MAP2K and MAPK activation / platelet activation / response to wounding / Golgi lumen / Signaling by RAF1 mutants / Signaling by moderate kinase activity BRAF mutants / Paradoxical activation of RAF signaling by kinase inactive BRAF / Signaling downstream of RAS mutants / Signaling by BRAF and RAF1 fusions / blood coagulation / integrin binding / Platelet degranulation / signaling receptor activity / protein-folding chaperone binding / collagen-containing extracellular matrix / protease binding / oxidoreductase activity / cell adhesion / copper ion binding / endoplasmic reticulum lumen / endoplasmic reticulum / extracellular space / extracellular exosome / extracellular region / identical protein binding / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
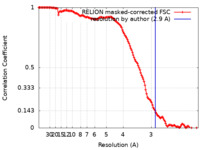
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.9 Å | |||||||||
 データ登録者 データ登録者 | Fuller JR / Batchelor JD | |||||||||
 引用 引用 | ジャーナル: Elife / 年: 2016 タイトル: Automated structure refinement of macromolecular assemblies from cryo-EM maps using Rosetta. 著者: Ray Yu-Ruei Wang / Yifan Song / Benjamin A Barad / Yifan Cheng / James S Fraser / Frank DiMaio /  要旨: Cryo-EM has revealed the structures of many challenging yet exciting macromolecular assemblies at near-atomic resolution (3-4.5Å), providing biological phenomena with molecular descriptions. ...Cryo-EM has revealed the structures of many challenging yet exciting macromolecular assemblies at near-atomic resolution (3-4.5Å), providing biological phenomena with molecular descriptions. However, at these resolutions, accurately positioning individual atoms remains challenging and error-prone. Manually refining thousands of amino acids - typical in a macromolecular assembly - is tedious and time-consuming. We present an automated method that can improve the atomic details in models that are manually built in near-atomic-resolution cryo-EM maps. Applying the method to three systems recently solved by cryo-EM, we are able to improve model geometry while maintaining the fit-to-density. Backbone placement errors are automatically detected and corrected, and the refinement shows a large radius of convergence. The results demonstrate that the method is amenable to structures with symmetry, of very large size, and containing RNA as well as covalently bound ligands. The method should streamline the cryo-EM structure determination process, providing accurate and unbiased atomic structure interpretation of such maps. #1:  ジャーナル: Comput. Cryst. Newsl. / 年: 2013 ジャーナル: Comput. Cryst. Newsl. / 年: 2013タイトル: New tool: phenix.real_space_refine 著者: Afonine PV / Headd JJ / Terwilliger TC / Adams PD | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_23057.map.gz emd_23057.map.gz | 170.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-23057-v30.xml emd-23057-v30.xml emd-23057.xml emd-23057.xml | 34.3 KB 34.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_23057_fsc.xml emd_23057_fsc.xml | 13.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_23057.png emd_23057.png | 112.1 KB | ||
| マスクデータ |  emd_23057_msk_1.map emd_23057_msk_1.map | 216 MB |  マスクマップ マスクマップ | |
| その他 |  emd_23057_additional_1.map.gz emd_23057_additional_1.map.gz emd_23057_half_map_1.map.gz emd_23057_half_map_1.map.gz emd_23057_half_map_2.map.gz emd_23057_half_map_2.map.gz | 125.7 MB 171.4 MB 171.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-23057 http://ftp.pdbj.org/pub/emdb/structures/EMD-23057 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23057 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23057 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_23057_validation.pdf.gz emd_23057_validation.pdf.gz | 706.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_23057_full_validation.pdf.gz emd_23057_full_validation.pdf.gz | 705.7 KB | 表示 | |
| XML形式データ |  emd_23057_validation.xml.gz emd_23057_validation.xml.gz | 21.2 KB | 表示 | |
| CIF形式データ |  emd_23057_validation.cif.gz emd_23057_validation.cif.gz | 28.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23057 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23057 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23057 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23057 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_23057.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_23057.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Full map from 3D refinement | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.06 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
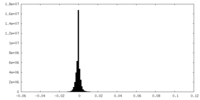
-マスク #1
| ファイル |  emd_23057_msk_1.map emd_23057_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
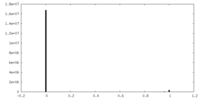
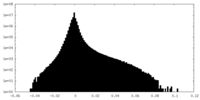
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
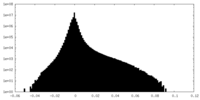
-追加マップ: Map sharpened by an automatically-fit B-factor (-69.3204) then...
| ファイル | emd_23057_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Map sharpened by an automatically-fit B-factor (-69.3204) then filtered to local resolution. Used for model building and refinement. | ||||||||||||
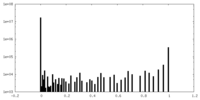
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Unfiltered half map 2
| ファイル | emd_23057_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unfiltered half map 2 | ||||||||||||
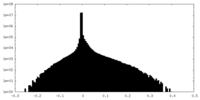
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Unfiltered half map 1
| ファイル | emd_23057_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unfiltered half map 1 | ||||||||||||
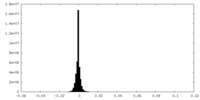
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : rFVIIIFc-VWF-XTEN (BIVV001)
| 全体 | 名称: rFVIIIFc-VWF-XTEN (BIVV001) |
|---|---|
| 要素 |
|
-超分子 #1: rFVIIIFc-VWF-XTEN (BIVV001)
| 超分子 | 名称: rFVIIIFc-VWF-XTEN (BIVV001) / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 詳細: An engineered therapeutic complex between coagulation factor VIII and von Willebrand factor D'D3 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 311.64974 KDa |
-分子 #1: Coagulation factor FVIII-Fc-XTEN
| 分子 | 名称: Coagulation factor FVIII-Fc-XTEN / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 218.874969 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MQIELSTCFF LCLLRFCFSA TRRYYLGAVE LSWDYMQSDL GELPVDARFP PRVPKSFPFN TSVVYKKTLF VEFTDHLFNI AKPRPPWMG LLGPTIQAEV YDTVVITLKN MASHPVSLHA VGVSYWKASE GAEYDDQTSQ REKEDDKVFP GGSHTYVWQV L KENGPMAS ...文字列: MQIELSTCFF LCLLRFCFSA TRRYYLGAVE LSWDYMQSDL GELPVDARFP PRVPKSFPFN TSVVYKKTLF VEFTDHLFNI AKPRPPWMG LLGPTIQAEV YDTVVITLKN MASHPVSLHA VGVSYWKASE GAEYDDQTSQ REKEDDKVFP GGSHTYVWQV L KENGPMAS DPLCLTYSYL SHVDLVKDLN SGLIGALLVC REGSLAKEKT QTLHKFILLF AVFDEGKSWH SETKNSLMQD RD AASARAW PKMHTVNGYV NRSLPGLIGC HRKSVYWHVI GMGTTPEVHS IFLEGHTFLV RNHRQASLEI SPITFLTAQT LLM DLGQFL LFCHISSHQH DGMEAYVKVD SCPEEPQLRM KNNEEAEDYD DDLTDSEMDV VRFDDDNSPS FIQIRSVAKK HPKT WVHYI AAEEEDWDYA PLVLAPDDRS YKSQYLNNGP QRIGRKYKKV RFMAYTDETF KTREAIQHES GILGPLLYGE VGDTL LIIF KNQASRPYNI YPHGITDVRP LYSRRLPKGV KHLKDFPILP GEIFKYKWTV TVEDGPTKSD PRCLTRYYSS FVNMER DLA SGLIGPLLIC YKESVDQRGN QIMSDKRNVI LFSVFDENRS WYLTENIQRF LPNPAGVQLE DPEFQASNIM HSINGYV FD SLQLSVCLHE VAYWYILSIG AQTDFLSVFF SGYTFKHKMV YEDTLTLFPF SGETVFMSME NPGLWILGCH NSDFRNRG M TALLKVSSCD KNTGDYYEDS YEDISAYLLS KNNAIEPRSF SQNGTSESAT PESGPGSEPA TSGSETPGTS ESATPESGP GSEPATSGSE TPGTSESATP ESGPGTSTEP SEGSAPGSPA GSPTSTEEGT SESATPESGP GSEPATSGSE TPGTSESATP ESGPGSPAG SPTSTEEGSP AGSPTSTEEG TSTEPSEGSA PGTSESATPE SGPGTSESAT PESGPGTSES ATPESGPGSE P ATSGSETP GSEPATSGSE TPGSPAGSPT STEEGTSTEP SEGSAPGTST EPSEGSAPGS EPATSGSETP GTSESATPES GP GTSTEPS EGSAPASSEI TRTTLQSDQE EIDYDDTISV EMKKEDFDI(TYS) DEDENQSPRS FQKKTRHYFI AAVERLWDY GMSSSPHVLR NRAQSGSVPQ FKKVVFQEFT DGSFTQPLYR GELNEHLGLL GPYIRAEVED NIMVTFRNQA SRPYSFYSSL ISYEEDQRQ GAEPRKNFVK PNETKTYFWK VQHHMAPTKD EFDCKAWAYF SDVDLEKDVH SGLIGPLLVC HTNTLNPAHG R QVTVQEFA LFFTIFDETK SWYFTENMER NCRAPCNIQM EDPTFKENYR FHAINGYIMD TLPGLVMAQD QRIRWYLLSM GS NENIHSI HFSGHVFTVR KKEEYKMALY NLYPGVFETV EMLPSKAGIW RVECLIGEHL HAGMSTLFLV YSNKCQTPLG MAS GHIRDF QITASGQYGQ WAPKLARLHY SGSINAWSTK EPFSWIKVDL LAPMIIHGIK TQGARQKFSS LYISQFIIMY SLDG KKWQT YRGNSTGTLM VFFGNVDSSG IKHNIFNPPI IARYIRLHPT HYSIRSTLRM ELMGCDLNSC SMPLGMESKA ISDAQ ITAS SYFTNMFATW SPSKARLHLQ GRSNAWRPQV NNPKEWLQVD FQKTMKVTGV TTQGVKSLLT SMYVKEFLIS SSQDGH QWT LFFQNGKVKV FQGNQDSFTP VVNSLDPPLL TRYLRIHPQS WVHQIALRME VLGCEAQDLY DKTHTCPPCP APELLGG PS VFLFPPKPKD TLMISRTPEV TCVVVDVSHE DPEVKFNWYV DGVEVHNAKT KPREEQYNST YRVVSVLTVL HQDWLNGK E YKCKVSNKAL PAPIEKTISK AKGQPREPQV YTLPPSRDEL TKNQVSLTCL VKGFYPSDIA VEWESNGQPE NNYKTTPPV LDSDGSFFLY SKLTVDKSRW QQGNVFSCSV MHEALHNHYT QKSLSLSPG |
-分子 #2: von Willebrand factor-XTEN-Fc
| 分子 | 名称: von Willebrand factor-XTEN-Fc / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 179.048953 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MIPARFAGVL LALALILPGT LCAEGTRGRS STARCSLFGS DFVNTFDGSM YSFAGYCSYL LAGGCQKRSF SIIGDFQNGK RVSLSVYLG EFFDIHLFVN GTVTQGDQRV SMPYASKGLY LETEAGYYKL SGEAYGFVAR IDGSGNFQVL LSDRYFNKTC G LCGNFNIF ...文字列: MIPARFAGVL LALALILPGT LCAEGTRGRS STARCSLFGS DFVNTFDGSM YSFAGYCSYL LAGGCQKRSF SIIGDFQNGK RVSLSVYLG EFFDIHLFVN GTVTQGDQRV SMPYASKGLY LETEAGYYKL SGEAYGFVAR IDGSGNFQVL LSDRYFNKTC G LCGNFNIF AEDDFMTQEG TLTSDPYDFA NSWALSSGEQ WCERASPPSS SCNISSGEMQ KGLWEQCQLL KSTSVFARCH PL VDPEPFV ALCEKTLCEC AGGLECACPA LLEYARTCAQ EGMVLYGWTD HSACSPVCPA GMEYRQCVSP CARTCQSLHI NEM CQERCV DGCSCPEGQL LDEGLCVEST ECPCVHSGKR YPPGTSLSRD CNTCICRNSQ WICSNEECPG ECLVTGQSHF KSFD NRYFT FSGICQYLLA RDCQDHSFSI VIETVQCADD RDAVCTRSVT VRLPGLHNSL VKLKHGAGVA MDGQDIQLPL LKGDL RIQH TVTASVRLSY GEDLQMDWDG RGRLLVKLSP VYAGKTCGLC GNYNGNQGDD FLTPSGLAEP RVEDFGNAWK LHGDCQ DLQ KQHSDPCALN PRMTRFSEEA CAVLTSPTFE ACHRAVSPLP YLRNCRYDVC SCSDGRECLC GALASYAAAC AGRGVRV AW REPGRCELNC PKGQVYLQCG TPCNLTCRSL SYPDEECNEA CLEGCFCPPG LYMDERGDCV PKAQCPCYYD GEIFQPED I FSDHHTMCYC EDGFMHCTMS GVPGSLLPDA VLSSPLSHRS KRSLSCRPPM VKLVCPADNL RAEGLECTKT CQNYDLECM SMGCVSGCLC PPGMVRHENR CVALERCPCF HQGKEYAPGE TVKIGCNTCV CRDRKWNCTD HVCDATCSTI GMAHYLTFDG LKYLFPGEC QYVLVQDYCG SNPGTFRILV GNKGCSHPSV KCKKRVTILV EGGEIELFDG EVNVKRPMKD ETHFEVVESG R YIILLLGK ALSVVWDRHL SISVVLKQTY QEKVCGLCGN FDGIQNNDLT SSNLQVEEDP VDFGNSWKVS SQCADTRKVP LD SSPATCH NNIMKQTMVD SSCRILTSDV FQDCNKLVDP EPYLDVCIYD TCSCESIGDC AAFCDTIAAY AHVCAQHGKV VTW RTATLC PQSCEERNLR ENGYEAEWRY NSCAPACQVT CQHPEPLACP VQCVEGCHAH CPPGKILDEL LQTCVDPEDC PVCE VAGRR FASGKKVTLN PSDPEHCQIC HCDVVNLTCE ACQEPGTSES ATPESGPGSE PATSGSETPG TSESATPESG PGSEP ATSG SETPGTSESA TPESGPGTST EPSEGSAPGS PAGSPTSTEE GTSESATPES GPGSEPATSG SETPGTSESA TPESGP GSP AGSPTSTEEG SPAGSPTSTE EGASSDKNTG DYYEDSYEDI SAYLLSKNNA IEPRSFSDKT HTCPPCPAPE LLGGPSV FL FPPKPKDTLM ISRTPEVTCV VVDVSHEDPE VKFNWYVDGV EVHNAKTKPR EEQYNSTYRV VSVLTVLHQD WLNGKEYK C KVSNKALPAP IEKTISKAKG QPREPQVYTL PPSRDELTKN QVSLTCLVKG FYPSDIAVEW ESNGQPENNY KTTPPVLDS DGSFFLYSKL TVDKSRWQQG NVFSCSVMHE ALHNHYTQKS LSLSPG |
-分子 #4: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 4 / コピー数: 3 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-分子 #5: CALCIUM ION
| 分子 | 名称: CALCIUM ION / タイプ: ligand / ID: 5 / コピー数: 2 / 式: CA |
|---|---|
| 分子量 | 理論値: 40.078 Da |
-分子 #6: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 6 / コピー数: 1 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-分子 #7: COPPER (II) ION
| 分子 | 名称: COPPER (II) ION / タイプ: ligand / ID: 7 / コピー数: 1 / 式: CU |
|---|---|
| 分子量 | 理論値: 63.546 Da |
| Chemical component information |  ChemComp-CU: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製 #1
試料調製 #1
| Preparation ID | 1 | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 濃度 | 0.75 mg/mL | |||||||||||||||
| 緩衝液 | pH: 7.3 構成要素:
詳細: NP-40s detergent was added to samples immediately prior to vitrification, to a final concentration of 0.0038 % (weight/volume). | |||||||||||||||
| グリッド | モデル: UltrAuFoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 支持フィルム - Film type ID: 1 / 支持フィルム - 材質: GOLD / 支持フィルム - トポロジー: HOLEY ARRAY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 291 K / 装置: FEI VITROBOT MARK IV | |||||||||||||||
| 詳細 | Prepared by gel filtration chromatography immediately prior to vitrification. |
- 試料調製 #2
試料調製 #2
| Preparation ID | 2 | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 濃度 | 0.75 mg/mL | |||||||||||||||
| 緩衝液 | pH: 7.3 構成要素:
詳細: NP-40s detergent was added to samples immediately prior to vitrification, to a final concentration of 0.0038 % (weight/volume). | |||||||||||||||
| グリッド | モデル: UltrAuFoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 支持フィルム - Film type ID: 1 / 支持フィルム - 材質: GOLD / 支持フィルム - トポロジー: HOLEY ARRAY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 291 K / 装置: FEI VITROBOT MARK IV | |||||||||||||||
| 詳細 | Prepared by gel filtration chromatography immediately prior to vitrification. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) デジタル化 - サイズ - 横: 11520 pixel / デジタル化 - サイズ - 縦: 8184 pixel / 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.6 µm / 最小 デフォーカス(公称値): 1.2 µm / 倍率(公称値): 81000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー































 Z
Z Y
Y X
X