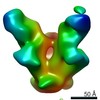
登録情報 データベース : EMDB / ID : EMD-22588タイトル Structure of TFIIH/Rad4-Rad23-Rad33/DNA in DNA opening Composite map after multibody refinement of TFIIH/Rad4-Rad23-Rad33/DNA with separate refinement of TFIIH and Rad4-23-33/DNA bodies 複合体 : 10 component complex of Rad4-23, Rad33 and TFIIH on damaged DNAタンパク質・ペプチド : x 8種DNA : x 2種タンパク質・ペプチド : x 1種リガンド : x 3種機能・相同性 分子機能 ドメイン・相同性 構成要素
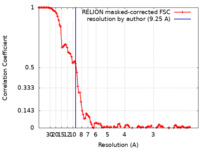
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Saccharomyces cerevisiae (strain ATCC 204508 / S288c) (パン酵母) / Baker's yeast (パン酵母) / synthetic construct (人工物) 手法 / / 解像度 : 9.25 Å van Eeuwen T / Min JH / Murakami K 資金援助 Organization Grant number 国 National Institutes of Health/National Institute of General Medical Sciences (NIH/NIGMS) R01-GM123233
ジャーナル : Nat Commun / 年 : 2021タイトル : Cryo-EM structure of TFIIH/Rad4-Rad23-Rad33 in damaged DNA opening in nucleotide excision repair.著者 : Trevor van Eeuwen / Yoonjung Shim / Hee Jong Kim / Tingting Zhao / Shrabani Basu / Benjamin A Garcia / Craig D Kaplan / Jung-Hyun Min / Kenji Murakami / 要旨 : The versatile nucleotide excision repair (NER) pathway initiates as the XPC-RAD23B-CETN2 complex first recognizes DNA lesions from the genomic DNA and recruits the general transcription factor ... The versatile nucleotide excision repair (NER) pathway initiates as the XPC-RAD23B-CETN2 complex first recognizes DNA lesions from the genomic DNA and recruits the general transcription factor complex, TFIIH, for subsequent lesion verification. Here, we present a cryo-EM structure of an NER initiation complex containing Rad4-Rad23-Rad33 (yeast homologue of XPC-RAD23B-CETN2) and 7-subunit coreTFIIH assembled on a carcinogen-DNA adduct lesion at 3.9-9.2 Å resolution. A ~30-bp DNA duplex could be mapped as it straddles between Rad4 and the Ssl2 (XPB) subunit of TFIIH on the 3' and 5' side of the lesion, respectively. The simultaneous binding with Rad4 and TFIIH was permitted by an unwinding of DNA at the lesion. Translocation coupled with torque generation by Ssl2 and Rad4 would extend the DNA unwinding at the lesion and deliver the damaged strand to Rad3 (XPD) in an open form suitable for subsequent lesion scanning and verification. 履歴 登録 2020年9月3日 - ヘッダ(付随情報) 公開 2021年7月28日 - マップ公開 2021年7月28日 - 更新 2021年7月28日 - 現状 2021年7月28日 処理サイト : RCSB / 状態 : 公開
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 マップデータ
マップデータ 試料
試料 機能・相同性情報
機能・相同性情報

 データ登録者
データ登録者 米国, 1件
米国, 1件  引用
引用 ジャーナル: Nat Commun / 年: 2021
ジャーナル: Nat Commun / 年: 2021
 構造の表示
構造の表示 ムービービューア
ムービービューア SurfView
SurfView Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク emd_22588.map.gz
emd_22588.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-22588-v30.xml
emd-22588-v30.xml emd-22588.xml
emd-22588.xml EMDBヘッダ
EMDBヘッダ emd_22588_fsc.xml
emd_22588_fsc.xml FSCデータファイル
FSCデータファイル emd_22588.png
emd_22588.png emd_22588_additional_1.map.gz
emd_22588_additional_1.map.gz emd_22588_additional_2.map.gz
emd_22588_additional_2.map.gz emd_22588_half_map_1.map.gz
emd_22588_half_map_1.map.gz emd_22588_half_map_2.map.gz
emd_22588_half_map_2.map.gz http://ftp.pdbj.org/pub/emdb/structures/EMD-22588
http://ftp.pdbj.org/pub/emdb/structures/EMD-22588 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22588
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22588 emd_22588_validation.pdf.gz
emd_22588_validation.pdf.gz EMDB検証レポート
EMDB検証レポート emd_22588_full_validation.pdf.gz
emd_22588_full_validation.pdf.gz emd_22588_validation.xml.gz
emd_22588_validation.xml.gz emd_22588_validation.cif.gz
emd_22588_validation.cif.gz https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22588
https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22588 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22588
ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22588 リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
マップ ダウンロード / ファイル: emd_22588.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_22588.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) 試料の構成要素
試料の構成要素 解析
解析 試料調製
試料調製 電子顕微鏡法
電子顕微鏡法 FIELD EMISSION GUN
FIELD EMISSION GUN
 ムービー
ムービー コントローラー
コントローラー






























 Z
Z Y
Y X
X





























 Trichoplusia ni (イラクサキンウワバ)
Trichoplusia ni (イラクサキンウワバ)