+ Open data
Open data
- Basic information
Basic information
| Entry | Database: EMDB / ID: EMD-22391 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Title | Sheep Connexin-50 at 2.5 angstroms resolution, Lipid Class 3 | |||||||||
 Map data Map data | Local resolution-filtered map. | |||||||||
 Sample Sample |
| |||||||||
 Keywords Keywords | Connexin / Gap Junction / Lipid / Nanodisc / MEMBRANE PROTEIN | |||||||||
| Function / homology |  Function and homology information Function and homology informationgap junction-mediated intercellular transport / gap junction hemi-channel activity / connexin complex / gap junction channel activity / visual perception / cell-cell signaling / plasma membrane Similarity search - Function | |||||||||
| Biological species |  | |||||||||
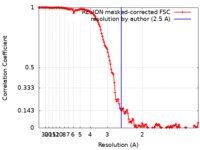
| Method | single particle reconstruction / cryo EM / Resolution: 2.5 Å | |||||||||
 Authors Authors | Flores JA / Haddad BG | |||||||||
| Funding support |  United States, 2 items United States, 2 items
| |||||||||
 Citation Citation | Journal: Acta Crystallogr D Struct Biol / Year: 2019 Title: Macromolecular structure determination using X-rays, neutrons and electrons: recent developments in Phenix. Authors: Dorothee Liebschner / Pavel V Afonine / Matthew L Baker / Gábor Bunkóczi / Vincent B Chen / Tristan I Croll / Bradley Hintze / Li Wei Hung / Swati Jain / Airlie J McCoy / Nigel W Moriarty ...Authors: Dorothee Liebschner / Pavel V Afonine / Matthew L Baker / Gábor Bunkóczi / Vincent B Chen / Tristan I Croll / Bradley Hintze / Li Wei Hung / Swati Jain / Airlie J McCoy / Nigel W Moriarty / Robert D Oeffner / Billy K Poon / Michael G Prisant / Randy J Read / Jane S Richardson / David C Richardson / Massimo D Sammito / Oleg V Sobolev / Duncan H Stockwell / Thomas C Terwilliger / Alexandre G Urzhumtsev / Lizbeth L Videau / Christopher J Williams / Paul D Adams /    Abstract: Diffraction (X-ray, neutron and electron) and electron cryo-microscopy are powerful methods to determine three-dimensional macromolecular structures, which are required to understand biological ...Diffraction (X-ray, neutron and electron) and electron cryo-microscopy are powerful methods to determine three-dimensional macromolecular structures, which are required to understand biological processes and to develop new therapeutics against diseases. The overall structure-solution workflow is similar for these techniques, but nuances exist because the properties of the reduced experimental data are different. Software tools for structure determination should therefore be tailored for each method. Phenix is a comprehensive software package for macromolecular structure determination that handles data from any of these techniques. Tasks performed with Phenix include data-quality assessment, map improvement, model building, the validation/rebuilding/refinement cycle and deposition. Each tool caters to the type of experimental data. The design of Phenix emphasizes the automation of procedures, where possible, to minimize repetitive and time-consuming manual tasks, while default parameters are chosen to encourage best practice. A graphical user interface provides access to many command-line features of Phenix and streamlines the transition between programs, project tracking and re-running of previous tasks. #2:  Journal: BioRxiv / Year: 2020 Journal: BioRxiv / Year: 2020Title: Connexin-46/50 in a dynamic lipid environment resolved by CryoEM at 1.9 angstroms Authors: Flores JA / Haddad BG / Dolan KA / Myers JB / Yoshioka CC / Copperman J / Zuckerman DM / Reichow SL | |||||||||
| History |
|
- Structure visualization
Structure visualization
| Movie |
 Movie viewer Movie viewer |
|---|---|
| Structure viewer | EM map:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| Supplemental images |
- Downloads & links
Downloads & links
-EMDB archive
| Map data |  emd_22391.map.gz emd_22391.map.gz | 153.7 MB |  EMDB map data format EMDB map data format | |
|---|---|---|---|---|
| Header (meta data) |  emd-22391-v30.xml emd-22391-v30.xml emd-22391.xml emd-22391.xml | 30 KB 30 KB | Display Display |  EMDB header EMDB header |
| FSC (resolution estimation) |  emd_22391_fsc.xml emd_22391_fsc.xml | 14.2 KB | Display |  FSC data file FSC data file |
| Images |  emd_22391.png emd_22391.png | 157.7 KB | ||
| Masks |  emd_22391_msk_1.map emd_22391_msk_1.map | 244.1 MB |  Mask map Mask map | |
| Filedesc metadata |  emd-22391.cif.gz emd-22391.cif.gz | 6.8 KB | ||
| Others |  emd_22391_additional_1.map.gz emd_22391_additional_1.map.gz emd_22391_additional_2.map.gz emd_22391_additional_2.map.gz emd_22391_additional_3.map.gz emd_22391_additional_3.map.gz emd_22391_additional_4.map.gz emd_22391_additional_4.map.gz emd_22391_half_map_1.map.gz emd_22391_half_map_1.map.gz emd_22391_half_map_2.map.gz emd_22391_half_map_2.map.gz | 228.3 MB 19.6 MB 225 MB 191.8 MB 192 MB 192 MB | ||
| Archive directory |  http://ftp.pdbj.org/pub/emdb/structures/EMD-22391 http://ftp.pdbj.org/pub/emdb/structures/EMD-22391 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22391 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22391 | HTTPS FTP |
-Related structure data
| Related structure data |  7jmcMC  7jn1MC  7jjpC  7jkcC  7jlwC  7jm9C  7jmdC  7jn0C M: atomic model generated by this map C: citing same article ( |
|---|---|
| Similar structure data | |
| EM raw data |  EMPIAR-10480 (Title: Connexin-46/50 in a dynamic lipid environment resolved by CryoEM at 1.9 Å EMPIAR-10480 (Title: Connexin-46/50 in a dynamic lipid environment resolved by CryoEM at 1.9 ÅData size: 4.1 TB Data #1: Unaligned multiframe micrographs of sheep lens Connexin-46/50 gap junctions embedded in MSP1E1 lipid nanodiscs [micrographs - multiframe]) |
- Links
Links
| EMDB pages |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- Map
Map
| File |  Download / File: emd_22391.map.gz / Format: CCP4 / Size: 244.1 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) Download / File: emd_22391.map.gz / Format: CCP4 / Size: 244.1 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Annotation | Local resolution-filtered map. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Projections & slices | Image control
Images are generated by Spider. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Voxel size | X=Y=Z: 0.649 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Density |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Symmetry | Space group: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Details | EMDB XML:
CCP4 map header:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-Supplemental data
-Mask #1
| File |  emd_22391_msk_1.map emd_22391_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Projections & Slices |
| ||||||||||||
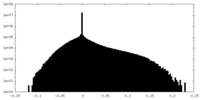
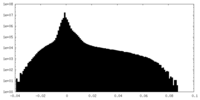
| Density Histograms |
-Additional map: Postprocessed map, unmasked.
| File | emd_22391_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Annotation | Postprocessed map, unmasked. | ||||||||||||
| Projections & Slices |
| ||||||||||||
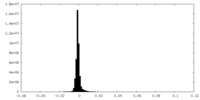
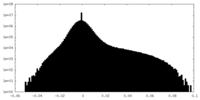
| Density Histograms |
-Additional map: Postprocessed map, masked.
| File | emd_22391_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Annotation | Postprocessed map, masked. | ||||||||||||
| Projections & Slices |
| ||||||||||||
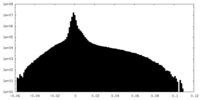
| Density Histograms |
-Additional map: Postprocessed map, lowpass-filtered to 3.5 angstroms resolution.
| File | emd_22391_additional_3.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Annotation | Postprocessed map, lowpass-filtered to 3.5 angstroms resolution. | ||||||||||||
| Projections & Slices |
| ||||||||||||
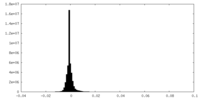
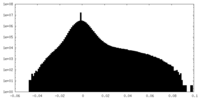
| Density Histograms |
-Additional map: Pre-postprocessed map.
| File | emd_22391_additional_4.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Annotation | Pre-postprocessed map. | ||||||||||||
| Projections & Slices |
| ||||||||||||
| Density Histograms |
-Half map: Half-map (even) used for calculation of pre-postprocessed, postprocessed,...
| File | emd_22391_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Annotation | Half-map (even) used for calculation of pre-postprocessed, postprocessed, and local resolution-filtered maps. | ||||||||||||
| Projections & Slices |
| ||||||||||||
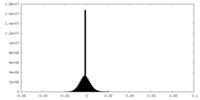
| Density Histograms |
-Half map: Half-map (odd) used for calculation of pre-postprocessed, postprocessed,...
| File | emd_22391_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Annotation | Half-map (odd) used for calculation of pre-postprocessed, postprocessed, and local resolution-filtered maps. | ||||||||||||
| Projections & Slices |
| ||||||||||||
| Density Histograms |
- Sample components
Sample components
-Entire : Dodecameric Connexin-50 Gap Junction Channel Complex
| Entire | Name: Dodecameric Connexin-50 Gap Junction Channel Complex |
|---|---|
| Components |
|
-Supramolecule #1: Dodecameric Connexin-50 Gap Junction Channel Complex
| Supramolecule | Name: Dodecameric Connexin-50 Gap Junction Channel Complex / type: complex / ID: 1 / Parent: 0 / Macromolecule list: #1 |
|---|---|
| Source (natural) | Organism:  |
| Molecular weight | Theoretical: 450 KDa |
-Macromolecule #1: Gap junction alpha-8 protein
| Macromolecule | Name: Gap junction alpha-8 protein / type: protein_or_peptide / ID: 1 / Number of copies: 12 / Enantiomer: LEVO |
|---|---|
| Source (natural) | Organism:  |
| Molecular weight | Theoretical: 49.216809 KDa |
| Sequence | String: MGDWSFLGNI LEEVNEHSTV IGRVWLTVLF IFRILILGTA AEFVWGDEQS DFVCNTQQPG CENVCYDEAF PISHIRLWVL QIIFVSTPS LVYVGHAVHH VRMEEKRKER EAEELSQQSP GNGGERAPLA ADQGSVKKSS SSSKGTKKFR LEGTLLRTYV C HIIFKTLF ...String: MGDWSFLGNI LEEVNEHSTV IGRVWLTVLF IFRILILGTA AEFVWGDEQS DFVCNTQQPG CENVCYDEAF PISHIRLWVL QIIFVSTPS LVYVGHAVHH VRMEEKRKER EAEELSQQSP GNGGERAPLA ADQGSVKKSS SSSKGTKKFR LEGTLLRTYV C HIIFKTLF EVGFIVGHYF LYGFRILPLY RCSRWPCPNV VDCFVSRPTE KTIFILFMLS VASVSLFLNI LEMSHLGLKK IR SAFKRPV EQPLGEIPEK SLHSIAVSSI QKAKGYQLLE EEKIVSHYFP LTEVGMVEAS PLSAKPFSQF EEKVGPGPLG DLS RAYQET LPSYAQVGAQ EGVEEEQPIE AAAEPEVGDK SQEAERVSTE GEETLAVLEE EKVEPPEVEK EAEKEETPPE KVSK QELTP EKAPSLCAEL PGEDTRPLSR LSKASSRARS DDLTV UniProtKB: Gap junction alpha-8 protein |
-Macromolecule #2: 1,2-DIMYRISTOYL-RAC-GLYCERO-3-PHOSPHOCHOLINE
| Macromolecule | Name: 1,2-DIMYRISTOYL-RAC-GLYCERO-3-PHOSPHOCHOLINE / type: ligand / ID: 2 / Number of copies: 144 / Formula: MC3 |
|---|---|
| Molecular weight | Theoretical: 677.933 Da |
| Chemical component information | 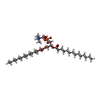 ChemComp-MC3: |
-Macromolecule #3: water
| Macromolecule | Name: water / type: ligand / ID: 3 / Number of copies: 168 / Formula: HOH |
|---|---|
| Molecular weight | Theoretical: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-Experimental details
-Structure determination
| Method | cryo EM |
|---|---|
 Processing Processing | single particle reconstruction |
| Aggregation state | particle |
- Sample preparation
Sample preparation
| Buffer | pH: 7.4 |
|---|---|
| Grid | Model: Quantifoil R1.2/1.3 / Material: COPPER / Support film - Material: CARBON / Support film - topology: HOLEY |
| Vitrification | Cryogen name: ETHANE / Chamber humidity: 100 % / Instrument: FEI VITROBOT MARK IV |
- Electron microscopy
Electron microscopy
| Microscope | FEI TITAN KRIOS |
|---|---|
| Image recording | Film or detector model: FEI FALCON III (4k x 4k) / Detector mode: COUNTING / Digitization - Dimensions - Width: 4096 pixel / Digitization - Dimensions - Height: 4096 pixel / Number grids imaged: 1 / Number real images: 2088 / Average exposure time: 150.0 sec. / Average electron dose: 52.5 e/Å2 |
| Electron beam | Acceleration voltage: 300 kV / Electron source:  FIELD EMISSION GUN FIELD EMISSION GUN |
| Electron optics | Illumination mode: FLOOD BEAM / Imaging mode: BRIGHT FIELD |
| Sample stage | Specimen holder model: FEI TITAN KRIOS AUTOGRID HOLDER / Cooling holder cryogen: NITROGEN |
| Experimental equipment |  Model: Titan Krios / Image courtesy: FEI Company |
 Movie
Movie Controller
Controller












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)