+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-22364 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Helical filaments of plant light-dependent protochlorophyllide oxidoreductase (LPOR) bound to NADPH, Pchlide, and membrane | ||||||||||||
 マップデータ マップデータ | RELION sharpened map (B factor -97) with local symmetry and then helical symmetry applied to the entire map | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | reductase / light-activated / ligand-protein complex / PHOTOSYNTHESIS | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報protochlorophyllide reductase / protochlorophyllide reductase activity / chloroplast outer membrane / response to ethylene / chlorophyll biosynthetic process / chloroplast thylakoid / chloroplast envelope / chloroplast thylakoid membrane / photosynthesis / chloroplast ...protochlorophyllide reductase / protochlorophyllide reductase activity / chloroplast outer membrane / response to ethylene / chlorophyll biosynthetic process / chloroplast thylakoid / chloroplast envelope / chloroplast thylakoid membrane / photosynthesis / chloroplast / protein domain specific binding / mRNA binding / cytosol 類似検索 - 分子機能 | ||||||||||||
| 生物種 |  | ||||||||||||
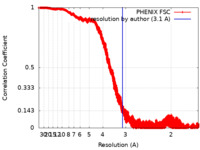
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 3.1 Å | ||||||||||||
 データ登録者 データ登録者 | Nguyen HC / Gabruk M | ||||||||||||
| 資金援助 |  ポーランド, ポーランド,  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Nat Plants / 年: 2021 ジャーナル: Nat Plants / 年: 2021タイトル: Photocatalytic LPOR forms helical lattices that shape membranes for chlorophyll synthesis. 著者: Henry C Nguyen / Arthur A Melo / Jerzy Kruk / Adam Frost / Michal Gabruk /   要旨: Chlorophyll biosynthesis, crucial to life on Earth, is tightly regulated because its precursors are phototoxic. In flowering plants, the enzyme light-dependent protochlorophyllide oxidoreductase ...Chlorophyll biosynthesis, crucial to life on Earth, is tightly regulated because its precursors are phototoxic. In flowering plants, the enzyme light-dependent protochlorophyllide oxidoreductase (LPOR) captures photons to catalyse the penultimate reaction: the reduction of a double bond within protochlorophyllide (Pchlide) to generate chlorophyllide (Chlide). In darkness, LPOR oligomerizes to facilitate photon energy transfer and catalysis. However, the complete three-dimensional structure of LPOR, the higher-order architecture of LPOR oligomers and the implications of these self-assembled states for catalysis, including how LPOR positions Pchlide and the co-factor NADPH, remain unknown. Here, we report the atomic structure of LPOR assemblies by electron cryo-microscopy. LPOR polymerizes with its substrates into helical filaments around constricted lipid bilayer tubes. Portions of LPOR and Pchlide insert into the outer membrane leaflet, targeting the product, Chlide, to the membrane for the final reaction site of chlorophyll biosynthesis. In addition to its crucial photocatalytic role, we show that in darkness LPOR filaments directly shape membranes into high-curvature tubules with the spectral properties of the prolamellar body, whose light-triggered disassembly provides lipids for thylakoid assembly. Moreover, our structure of the catalytic site challenges previously proposed reaction mechanisms. Together, our results reveal a new and unexpected synergy between photosynthetic membrane biogenesis and chlorophyll synthesis in plants, orchestrated by LPOR. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_22364.map.gz emd_22364.map.gz | 48.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-22364-v30.xml emd-22364-v30.xml emd-22364.xml emd-22364.xml | 20.9 KB 20.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_22364_fsc.xml emd_22364_fsc.xml | 3.5 MB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_22364.png emd_22364.png | 468.7 KB | ||
| マスクデータ |  emd_22364_msk_1.map emd_22364_msk_1.map | 216 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-22364.cif.gz emd-22364.cif.gz | 6.1 KB | ||
| その他 |  emd_22364_additional_1.map.gz emd_22364_additional_1.map.gz emd_22364_additional_2.map.gz emd_22364_additional_2.map.gz emd_22364_half_map_1.map.gz emd_22364_half_map_1.map.gz emd_22364_half_map_2.map.gz emd_22364_half_map_2.map.gz | 200.3 MB 170.9 MB 170.8 MB 170.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-22364 http://ftp.pdbj.org/pub/emdb/structures/EMD-22364 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22364 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22364 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_22364_validation.pdf.gz emd_22364_validation.pdf.gz | 1.2 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_22364_full_validation.pdf.gz emd_22364_full_validation.pdf.gz | 1.2 MB | 表示 | |
| XML形式データ |  emd_22364_validation.xml.gz emd_22364_validation.xml.gz | 246.1 KB | 表示 | |
| CIF形式データ |  emd_22364_validation.cif.gz emd_22364_validation.cif.gz | 889.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22364 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22364 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22364 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22364 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_22364.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_22364.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | RELION sharpened map (B factor -97) with local symmetry and then helical symmetry applied to the entire map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.11333 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
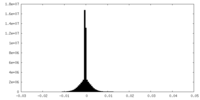
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_22364_msk_1.map emd_22364_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
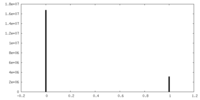
| 投影像・断面図 |
| ||||||||||||
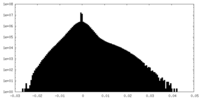
| 密度ヒストグラム |
-追加マップ: RELION sharpened map (B factor -97) with central Z of 40%
| ファイル | emd_22364_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | RELION sharpened map (B factor -97) with central Z of 40% | ||||||||||||
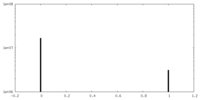
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: RELION Refine3D summed and filtered map with central Z of 40%
| ファイル | emd_22364_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | RELION Refine3D summed and filtered map with central Z of 40% | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: RELION Refine3D unfiltered half map with central Z of 40%
| ファイル | emd_22364_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | RELION Refine3D unfiltered half map with central Z of 40% | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: RELION Refine3D unfiltered half map with central Z of 40%
| ファイル | emd_22364_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | RELION Refine3D unfiltered half map with central Z of 40% | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : light-dependent protochlorophyllide oxidoreductase bound to NADPH...
| 全体 | 名称: light-dependent protochlorophyllide oxidoreductase bound to NADPH, Pchlide, and lipid membrane |
|---|---|
| 要素 |
|
-超分子 #1: light-dependent protochlorophyllide oxidoreductase bound to NADPH...
| 超分子 | 名称: light-dependent protochlorophyllide oxidoreductase bound to NADPH, Pchlide, and lipid membrane タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Protochlorophyllide reductase B, chloroplastic
| 分子 | 名称: Protochlorophyllide reductase B, chloroplastic / タイプ: protein_or_peptide / ID: 1 / コピー数: 40 / 光学異性体: LEVO / EC番号: protochlorophyllide reductase |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 43.415199 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MALQAASLVS SAFSVRKDAK LNASSSSFKD SSLFGASITD QIKSEHGSSS LRFKREQSLR NLAIRAQTAA TSSPTVTKSV DGKKTLRKG NVVVTGASSG LGLATAKALA ETGKWNVIMA CRDFLKAERA AKSVGMPKDS YTVMHLDLAS LDSVRQFVDN F RRTETPLD ...文字列: MALQAASLVS SAFSVRKDAK LNASSSSFKD SSLFGASITD QIKSEHGSSS LRFKREQSLR NLAIRAQTAA TSSPTVTKSV DGKKTLRKG NVVVTGASSG LGLATAKALA ETGKWNVIMA CRDFLKAERA AKSVGMPKDS YTVMHLDLAS LDSVRQFVDN F RRTETPLD VLVCNAAVYF PTAKEPTYSA EGFELSVATN HLGHFLLARL LLDDLKKSDY PSKRLIIVGS ITGNTNTLAG NV PPKANLG DLRGLAGGLN GLNSSAMIDG GDFDGAKAYK DSKVCNMLTM QEFHRRFHEE TGVTFASLYP GCIASTGLFR EHI PLFRAL FPPFQKYITK GYVSETESGK RLAQVVSDPS LTKSGVYWSW NNASASFENQ LSEEASDVEK ARKVWEISEK LVGL A UniProtKB: Protochlorophyllide reductase B, chloroplastic |
-分子 #2: NADPH DIHYDRO-NICOTINAMIDE-ADENINE-DINUCLEOTIDE PHOSPHATE
| 分子 | 名称: NADPH DIHYDRO-NICOTINAMIDE-ADENINE-DINUCLEOTIDE PHOSPHATE タイプ: ligand / ID: 2 / コピー数: 40 / 式: NDP |
|---|---|
| 分子量 | 理論値: 745.421 Da |
| Chemical component information |  ChemComp-NDP: |
-分子 #3: Protochlorophyllide
| 分子 | 名称: Protochlorophyllide / タイプ: ligand / ID: 3 / コピー数: 40 / 式: PMR |
|---|---|
| 分子量 | 理論値: 612.957 Da |
| Chemical component information |  ChemComp-PMR: |
-分子 #4: 1,2-DISTEAROYL-MONOGALACTOSYL-DIGLYCERIDE
| 分子 | 名称: 1,2-DISTEAROYL-MONOGALACTOSYL-DIGLYCERIDE / タイプ: ligand / ID: 4 / コピー数: 40 / 式: LMG |
|---|---|
| 分子量 | 理論値: 787.158 Da |
| Chemical component information |  ChemComp-LMG: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 緩衝液 | pH: 7.1 |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 200 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 298 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 73.5 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー













 Z
Z Y
Y X
X