[日本語] English
 万見
万見- EMDB-21152: Tetradecameric PilQ bound by TsaP heptamer from Pseudomonas aeruginosa -
+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-21152 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
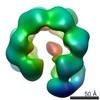
| タイトル | Tetradecameric PilQ bound by TsaP heptamer from Pseudomonas aeruginosa | |||||||||
 マップデータ マップデータ | sharpened map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Type IV pilus / T4P / PilQ / TsaP / secretin / pilotin / type IVa pilus / T4aP / pilus / outer membrane / periplasm / bacterial secretion system / PROTEIN TRANSPORT | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Pseudomonas aeruginosa (strain ATCC 15692 / DSM 22644 / CIP 104116 / JCM 14847 / LMG 12228 / 1C / PRS 101 / PAO1) (緑膿菌) Pseudomonas aeruginosa (strain ATCC 15692 / DSM 22644 / CIP 104116 / JCM 14847 / LMG 12228 / 1C / PRS 101 / PAO1) (緑膿菌) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.3 Å | |||||||||
 データ登録者 データ登録者 | McCallum M / Tammam S | |||||||||
| 資金援助 |  カナダ, 1件 カナダ, 1件
| |||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2021 ジャーナル: Structure / 年: 2021タイトル: CryoEM map of Pseudomonas aeruginosa PilQ enables structural characterization of TsaP. 著者: Matthew McCallum / Stephanie Tammam / John L Rubinstein / Lori L Burrows / P Lynne Howell /  要旨: The type IV pilus machinery is a multi-protein complex that polymerizes and depolymerizes a pilus fiber used for attachment, twitching motility, phage adsorption, natural competence, protein ...The type IV pilus machinery is a multi-protein complex that polymerizes and depolymerizes a pilus fiber used for attachment, twitching motility, phage adsorption, natural competence, protein secretion, and surface-sensing. An outer membrane secretin pore is required for passage of the pilus fiber out of the cell. Herein, the structure of the tetradecameric secretin, PilQ, from the Pseudomonas aeruginosa type IVa pilus system was determined to 4.3 Å and 4.4 Å resolution in the presence and absence of C symmetric spikes, respectively. The heptameric spikes were found to be two tandem C-terminal domains of TsaP. TsaP forms a belt around PilQ and while it is not essential for twitching motility, overexpression of TsaP triggers a signal cascade upstream of PilY1 leading to cyclic di-GMP up-regulation. These results resolve the identity of the spikes identified with Proteobacterial PilQ homologs and may reveal a new component of the surface-sensing cyclic di-GMP signal cascade. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_21152.map.gz emd_21152.map.gz | 59.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-21152-v30.xml emd-21152-v30.xml emd-21152.xml emd-21152.xml | 19.3 KB 19.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_21152.png emd_21152.png | 186.4 KB | ||
| Filedesc metadata |  emd-21152.cif.gz emd-21152.cif.gz | 6.1 KB | ||
| その他 |  emd_21152_additional_1.map.gz emd_21152_additional_1.map.gz emd_21152_half_map_1.map.gz emd_21152_half_map_1.map.gz emd_21152_half_map_2.map.gz emd_21152_half_map_2.map.gz | 31.6 MB 59.3 MB 59.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-21152 http://ftp.pdbj.org/pub/emdb/structures/EMD-21152 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21152 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21152 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_21152_validation.pdf.gz emd_21152_validation.pdf.gz | 874.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_21152_full_validation.pdf.gz emd_21152_full_validation.pdf.gz | 874.1 KB | 表示 | |
| XML形式データ |  emd_21152_validation.xml.gz emd_21152_validation.xml.gz | 12.4 KB | 表示 | |
| CIF形式データ |  emd_21152_validation.cif.gz emd_21152_validation.cif.gz | 14.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21152 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21152 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21152 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21152 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_21152.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_21152.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | sharpened map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.45 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: Unsharpened map
| ファイル | emd_21152_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unsharpened map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half Map A
| ファイル | emd_21152_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half Map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half Map B
| ファイル | emd_21152_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half Map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : PilQ with TsaP
| 全体 | 名称: PilQ with TsaP |
|---|---|
| 要素 |
|
-超分子 #1: PilQ with TsaP
| 超分子 | 名称: PilQ with TsaP / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all / 詳細: Affinity purified PilQ with pulled-down TsaP |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas aeruginosa (strain ATCC 15692 / DSM 22644 / CIP 104116 / JCM 14847 / LMG 12228 / 1C / PRS 101 / PAO1) (緑膿菌) Pseudomonas aeruginosa (strain ATCC 15692 / DSM 22644 / CIP 104116 / JCM 14847 / LMG 12228 / 1C / PRS 101 / PAO1) (緑膿菌)株: ATCC 15692 / DSM 22644 / CIP 104116 / JCM 14847 / LMG 12228 / 1C / PRS 101 / PAO1 |
-分子 #1: Fimbrial assembly protein PilQ
| 分子 | 名称: Fimbrial assembly protein PilQ / タイプ: protein_or_peptide / ID: 1 / コピー数: 14 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas aeruginosa (strain ATCC 15692 / DSM 22644 / CIP 104116 / JCM 14847 / LMG 12228 / 1C / PRS 101 / PAO1) (緑膿菌) Pseudomonas aeruginosa (strain ATCC 15692 / DSM 22644 / CIP 104116 / JCM 14847 / LMG 12228 / 1C / PRS 101 / PAO1) (緑膿菌)株: ATCC 15692 / DSM 22644 / CIP 104116 / JCM 14847 / LMG 12228 / 1C / PRS 101 / PAO1 |
| 分子量 | 理論値: 79.731141 KDa |
| 組換発現 | 生物種:  Pseudomonas aeruginosa PAO1 (緑膿菌) Pseudomonas aeruginosa PAO1 (緑膿菌) |
| 配列 | 文字列: MNSGLSRLGI ALLAAMFAPA LLAADLEKLD VAALPGDRVE LKLQFDEPVA APRGYTIEQP ARIALDLPGV QNKLGTKNRE LSVGNTRSV TVVEAKDRTR LIINLTALSS YTTRVEGNNL FVVVGNSPHH HHHHHHAGAS VASAAPVKAS PAPASYAQPI K PKPYVPAG ...文字列: MNSGLSRLGI ALLAAMFAPA LLAADLEKLD VAALPGDRVE LKLQFDEPVA APRGYTIEQP ARIALDLPGV QNKLGTKNRE LSVGNTRSV TVVEAKDRTR LIINLTALSS YTTRVEGNNL FVVVGNSPHH HHHHHHAGAS VASAAPVKAS PAPASYAQPI K PKPYVPAG RAIRNIDFQR GEKGEGNVVI DLSDPTLSPD IQEQGGKIRL DFAKTQLPDA LRVRLDVKDF ATPVQFVNAS AQ SDRTSIT IEPSGLYDYL VYQTDNRLTV SIKPMTTEDA ERRKKDNFAY TGEKLSLNFQ DIDVRSVLQL IADFTDLNLV ASD TVQGNI TLRLQNVPWD QALDLVLKTK GLDKRKLGNV LLVAPADEIA ARERQELEAQ KQIAELAPLR RELIQVNYAK AADI AKLFQ SVTSDGGQEG KEGGRGSITV DDRTNSIIAY QPQERLDELR RIVSQLDIPV RQVMIEARIV EANVGYDKSL GVRWG GAYH KGNWSGYGKD GNIGIKDEDG MNCGPIAGSC TFPTTGTSKS PSPFVDLGAK DATSGIGIGF ITDNIILDLQ LSAMEK TGN GEIVSQPKVV TSDKETAKIL KGSEVPYQEA SSSGATSTSF KEAALSLEVT PQITPDNRII VEVKVTKDAP DYQNMLN GV PPINKNEVNA KILVNDGETI VIGGVFSNEQ SKSVEKVPFL GELPYLGRLF RRDTVTDRKN ELLVFLTPRI MNNQAIAI G RGHHHHHHHH UniProtKB: Fimbrial assembly protein PilQ |
-分子 #2: LysM domain-containing protein
| 分子 | 名称: LysM domain-containing protein / タイプ: protein_or_peptide / ID: 2 / コピー数: 7 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas aeruginosa (strain ATCC 15692 / DSM 22644 / CIP 104116 / JCM 14847 / LMG 12228 / 1C / PRS 101 / PAO1) (緑膿菌) Pseudomonas aeruginosa (strain ATCC 15692 / DSM 22644 / CIP 104116 / JCM 14847 / LMG 12228 / 1C / PRS 101 / PAO1) (緑膿菌)株: ATCC 15692 / DSM 22644 / CIP 104116 / JCM 14847 / LMG 12228 / 1C / PRS 101 / PAO1 |
| 分子量 | 理論値: 37.632902 KDa |
| 組換発現 | 生物種:  Pseudomonas aeruginosa PAO1 (緑膿菌) Pseudomonas aeruginosa PAO1 (緑膿菌) |
| 配列 | 文字列: MRKSLVALLL LAASGLAQAQ VDLREGHPDR YTVVRGDTLW DISGKFLRQP WKWPELWHAN PQIQNPHLIY PGDTLSLVYV DGQPRLVLN RGESRGTIKL SPKIRSTPIA EAIPTIPLDK INSFLLANRI VDDEKTFTSA PYIVAGNAER IVSGTGDRIY A RGKFADGQ ...文字列: MRKSLVALLL LAASGLAQAQ VDLREGHPDR YTVVRGDTLW DISGKFLRQP WKWPELWHAN PQIQNPHLIY PGDTLSLVYV DGQPRLVLN RGESRGTIKL SPKIRSTPIA EAIPTIPLDK INSFLLANRI VDDEKTFTSA PYIVAGNAER IVSGTGDRIY A RGKFADGQ PAYGIFRQGK VYIDPKTKEV LGINADDIGG GEVVATEGDV ATLALTRTTQ EVRLGDRLFP TEERAVNSTF MP GEPSREV KGEIIDVPRG VTQIGQFDVV TLNRGQRDGL AEGNVLAIYK VGETVRDRVT GESVKIPDER AGLLMVFRTY KKL SYALVL MASRPLSVTD RVQNP UniProtKB: LysM domain-containing protein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| グリッド | 詳細: unspecified |
| 凍結 | 凍結剤: ETHANE-PROPANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F20 |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 35.7 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: INSILICO MODEL In silico モデル: Starting models generated in Phyre2 and I-TASSER |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: C7 (7回回転対称) / 解像度のタイプ: BY AUTHOR / 解像度: 4.3 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: cryoSPARC / 使用した粒子像数: 47468 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: cryoSPARC |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: cryoSPARC |
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT / 温度因子: 98 |
|---|---|
| 得られたモデル |  PDB-6ve2: |
 ムービー
ムービー コントローラー
コントローラー












 Z
Z Y
Y X
X