+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-21019 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM Structure of the Hyperpolarization-Activated Potassium Channel KAT1: Octamer | |||||||||
 マップデータ マップデータ | Structure of the Hyperpolarization-Activated Potassium Channel KAT1 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | membrane protein / voltage-gated ion channel / potassium channel / TRANSPORT PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報inward rectifier potassium channel activity / monoatomic ion channel complex / identical protein binding / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
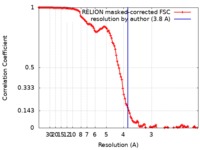
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.8 Å | |||||||||
 データ登録者 データ登録者 | Clark MD / Contreras GF | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2020 ジャーナル: Nature / 年: 2020タイトル: Electromechanical coupling in the hyperpolarization-activated K channel KAT1. 著者: Michael David Clark / Gustavo F Contreras / Rong Shen / Eduardo Perozo /  要旨: Voltage-gated potassium (K) channels coordinate electrical signalling and control cell volume by gating in response to membrane depolarization or hyperpolarization. However, although voltage-sensing ...Voltage-gated potassium (K) channels coordinate electrical signalling and control cell volume by gating in response to membrane depolarization or hyperpolarization. However, although voltage-sensing domains transduce transmembrane electric field changes by a common mechanism involving the outward or inward translocation of gating charges, the general determinants of channel gating polarity remain poorly understood. Here we suggest a molecular mechanism for electromechanical coupling and gating polarity in non-domain-swapped K channels on the basis of the cryo-electron microscopy structure of KAT1, the hyperpolarization-activated K channel from Arabidopsis thaliana. KAT1 displays a depolarized voltage sensor, which interacts with a closed pore domain directly via two interfaces and indirectly via an intercalated phospholipid. Functional evaluation of KAT1 structure-guided mutants at the sensor-pore interfaces suggests a mechanism in which direct interaction between the sensor and the C-linker hairpin in the adjacent pore subunit is the primary determinant of gating polarity. We suggest that an inward motion of the S4 sensor helix of approximately 5-7 Å can underlie a direct-coupling mechanism, driving a conformational reorientation of the C-linker and ultimately opening the activation gate formed by the S6 intracellular bundle. This direct-coupling mechanism contrasts with allosteric mechanisms proposed for hyperpolarization-activated cyclic nucleotide-gated channels, and may represent an unexpected link between depolarization- and hyperpolarization-activated channels. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_21019.map.gz emd_21019.map.gz | 15.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-21019-v30.xml emd-21019-v30.xml emd-21019.xml emd-21019.xml | 14.5 KB 14.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_21019_fsc.xml emd_21019_fsc.xml | 12.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_21019.png emd_21019.png | 36.9 KB | ||
| Filedesc metadata |  emd-21019.cif.gz emd-21019.cif.gz | 6.1 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-21019 http://ftp.pdbj.org/pub/emdb/structures/EMD-21019 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21019 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21019 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_21019_validation.pdf.gz emd_21019_validation.pdf.gz | 420.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_21019_full_validation.pdf.gz emd_21019_full_validation.pdf.gz | 420.5 KB | 表示 | |
| XML形式データ |  emd_21019_validation.xml.gz emd_21019_validation.xml.gz | 12.7 KB | 表示 | |
| CIF形式データ |  emd_21019_validation.cif.gz emd_21019_validation.cif.gz | 17.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21019 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21019 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21019 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21019 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6v1yMC  6v1xC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-11054 (タイトル: Cryo-EM Structure of the Hyperpolarization-Activated Potassium Channel KAT1 EMPIAR-11054 (タイトル: Cryo-EM Structure of the Hyperpolarization-Activated Potassium Channel KAT1Data size: 3.1 TB Data #1: unaligned non-gain-ref movies [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_21019.map.gz / 形式: CCP4 / 大きさ: 149.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_21019.map.gz / 形式: CCP4 / 大きさ: 149.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Structure of the Hyperpolarization-Activated Potassium Channel KAT1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.064 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
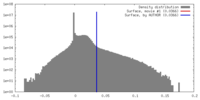
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Hyperpolarization-Activated Potassium Channel KAT1
| 全体 | 名称: Hyperpolarization-Activated Potassium Channel KAT1 |
|---|---|
| 要素 |
|
-超分子 #1: Hyperpolarization-Activated Potassium Channel KAT1
| 超分子 | 名称: Hyperpolarization-Activated Potassium Channel KAT1 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Potassium channel KAT1
| 分子 | 名称: Potassium channel KAT1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 8 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 59.639594 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSISWTRNFF ERFCVEEYNI DTIKQSSFLS ADLLPSLGAR INQSTKLRKH IISPFNPRYR AWEMWLVLLV IYSAWICPFQ FAFITYKKD AIFIIDNIVN GFFAIDIILT FFVAYLDSHS YLLVDSPKKI AIRYLSTWFA FDVCSTAPFQ PLSLLFNYNG S ELGFRILS ...文字列: MSISWTRNFF ERFCVEEYNI DTIKQSSFLS ADLLPSLGAR INQSTKLRKH IISPFNPRYR AWEMWLVLLV IYSAWICPFQ FAFITYKKD AIFIIDNIVN GFFAIDIILT FFVAYLDSHS YLLVDSPKKI AIRYLSTWFA FDVCSTAPFQ PLSLLFNYNG S ELGFRILS MLRLWRLRRV SSLFARLEKD IRFNYFWIRC TKLISVTLFA IHCAGCFNYL IADRYPNPRK TWIGAVYPNF KE ASLWNRY VTALYWSITT LTTTGYGDFH AENPREMLFD IFFMMFNLGL TAYLIGNMTN LVVHWTSRTR TFRDSVRAAS EFA SRNQLP HDIQDQMLSH ICLKFKTEGL KQQETLNNLP KAIRSSIANY LFFPIVHNIY LFQGVSRNFL FQLVSDIDAE YFPP KEDII LQNEAPTDLY ILVSGAVDFT VYVDGHDQFQ GKAVIGETFG EVGVLYYRPQ PFTVRTTELS QILRISRTSL MSAMH AHAD DGRVIMNNLF MKLRGQQSSR GSLEVLFQ UniProtKB: Potassium channel KAT1 |
-分子 #2: (2S)-1-(nonanoyloxy)-3-(phosphonooxy)propan-2-yl tetradecanoate
| 分子 | 名称: (2S)-1-(nonanoyloxy)-3-(phosphonooxy)propan-2-yl tetradecanoate タイプ: ligand / ID: 2 / コピー数: 8 / 式: QNP |
|---|---|
| 分子量 | 理論値: 522.652 Da |
| Chemical component information |  ChemComp-QNP: |
-分子 #3: (3beta,5beta,14beta,17alpha)-cholestan-3-ol
| 分子 | 名称: (3beta,5beta,14beta,17alpha)-cholestan-3-ol / タイプ: ligand / ID: 3 / コピー数: 8 / 式: QNJ |
|---|---|
| 分子量 | 理論値: 388.669 Da |
| Chemical component information | 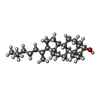 ChemComp-QNJ: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 5 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.4 構成要素:
| |||||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 200 / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 時間: 30 sec. / 前処理 - 雰囲気: AIR | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 295 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / デジタル化 - 画像ごとのフレーム数: 1-40 / 撮影したグリッド数: 1 / 実像数: 1502 / 平均露光時間: 12.0 sec. / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 130000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL |
|---|---|
| 得られたモデル |  PDB-6v1y: |
 ムービー
ムービー コントローラー
コントローラー
















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)