+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-20504 | |||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | 10 Angstrom structure of the asymmetric flagellar filament purified from Leptospira biflexa Patoc WT cells resolved via subtomogram averaging | |||||||||||||||||||||||||||||||||
 マップデータ マップデータ | Masked full EM map resulting from subtomogram averaging in emClarity of flagellar filaments purified from wildtype Leptospira biflexa serovar Patoc strain Patoc 1. | |||||||||||||||||||||||||||||||||
 試料 試料 |
| |||||||||||||||||||||||||||||||||
 キーワード キーワード | bacterial flagella / FcpA / FcpB / FlaA / FlaB / Leptospira / STRUCTURAL PROTEIN | |||||||||||||||||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報periplasmic flagellum / bacterial-type flagellum filament / bacterial-type flagellum-dependent cell motility / structural molecule activity 類似検索 - 分子機能 | |||||||||||||||||||||||||||||||||
| 生物種 |  Leptospira biflexa serovar Patoc strain 'Patoc 1 (Paris)' (バクテリア) / Leptospira biflexa serovar Patoc strain 'Patoc 1 (Paris)' (バクテリア) /  Leptospira biflexa serovar Patoc (strain Patoc 1 / ATCC 23582 / Paris) (バクテリア) Leptospira biflexa serovar Patoc (strain Patoc 1 / ATCC 23582 / Paris) (バクテリア) | |||||||||||||||||||||||||||||||||
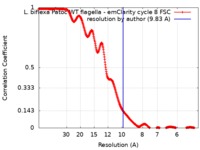
| 手法 | サブトモグラム平均法 / クライオ電子顕微鏡法 / 解像度: 9.83 Å | |||||||||||||||||||||||||||||||||
 データ登録者 データ登録者 | Gibson KH / Sindelar CV | |||||||||||||||||||||||||||||||||
| 資金援助 |  米国, ウルグアイ, 10件 米国, ウルグアイ, 10件
| |||||||||||||||||||||||||||||||||
 引用 引用 | ジャーナル: Front Cell Infect Microbiol / 年: 2018 タイトル: FcpB Is a Surface Filament Protein of the Endoflagellum Required for the Motility of the Spirochete . 著者: Elsio A Wunder / Leyla Slamti / David N Suwondo / Kimberley H Gibson / Zhiguo Shang / Charles V Sindelar / Felipe Trajtenberg / Alejandro Buschiazzo / Albert I Ko / Mathieu Picardeau /    要旨: The spirochete endoflagellum is a unique motility apparatus among bacteria. Despite its critical importance for pathogenesis, the full composition of the flagellum remains to be determined. We have ...The spirochete endoflagellum is a unique motility apparatus among bacteria. Despite its critical importance for pathogenesis, the full composition of the flagellum remains to be determined. We have recently reported that FcpA is a novel flagellar protein and a major component of the sheath of the filament of the spirochete . By screening a library of random transposon mutants in the spirochete , we found a motility-deficient mutant harboring a disruption in a hypothetical gene of unknown function. Here, we show that this gene encodes a surface component of the endoflagellar filament and is required for typical hook- and spiral-shaped ends of the cell body, coiled structure of the endoflagella, and high velocity phenotype. We therefore named the gene for flagellar-coiling protein B. is conserved in all members of the genus, but not present in other organisms including other spirochetes. Complementation of the mutant restored the wild-type morphology and motility phenotypes. Immunoblotting with anti-FcpA and anti-FcpB antisera and cryo-electron microscopy of the filament indicated that FcpB assembled onto the surface of the sheath of the filament and mostly located on the outer (convex) side of the coiled filament. We provide evidence that FcpB, together with FcpA, are -specific novel components of the sheath of the filament, key determinants of the coiled and asymmetric structure of the endoflagella and are essential for high velocity. Defining the components of the endoflagella and their functions in these atypical bacteria should greatly enhance our understanding of the mechanisms by which these bacteria produce motility. | |||||||||||||||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_20504.map.gz emd_20504.map.gz | 25.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-20504-v30.xml emd-20504-v30.xml emd-20504.xml emd-20504.xml | 40.4 KB 40.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_20504_fsc.xml emd_20504_fsc.xml | 34.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_20504.png emd_20504.png | 97.9 KB | ||
| Filedesc metadata |  emd-20504.cif.gz emd-20504.cif.gz | 9.1 KB | ||
| その他 |  emd_20504_additional_1.map.gz emd_20504_additional_1.map.gz emd_20504_additional_2.map.gz emd_20504_additional_2.map.gz emd_20504_additional_3.map.gz emd_20504_additional_3.map.gz emd_20504_additional_4.map.gz emd_20504_additional_4.map.gz emd_20504_half_map_1.map.gz emd_20504_half_map_1.map.gz emd_20504_half_map_2.map.gz emd_20504_half_map_2.map.gz | 25.5 MB 25.1 MB 25.5 MB 25.5 MB 25.1 MB 25.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-20504 http://ftp.pdbj.org/pub/emdb/structures/EMD-20504 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20504 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20504 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_20504_validation.pdf.gz emd_20504_validation.pdf.gz | 804.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_20504_full_validation.pdf.gz emd_20504_full_validation.pdf.gz | 804.4 KB | 表示 | |
| XML形式データ |  emd_20504_validation.xml.gz emd_20504_validation.xml.gz | 23.2 KB | 表示 | |
| CIF形式データ |  emd_20504_validation.cif.gz emd_20504_validation.cif.gz | 32.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20504 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20504 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20504 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20504 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_20504.map.gz / 形式: CCP4 / 大きさ: 27 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_20504.map.gz / 形式: CCP4 / 大きさ: 27 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Masked full EM map resulting from subtomogram averaging in emClarity of flagellar filaments purified from wildtype Leptospira biflexa serovar Patoc strain Patoc 1. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
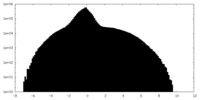
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.604 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: Masked half map 1 resulting from subtomogram averaging...
| ファイル | emd_20504_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Masked half map 1 resulting from subtomogram averaging in emClarity of flagellar filaments purified from wildtype Leptospira biflexa serovar Patoc strain Patoc 1. | ||||||||||||
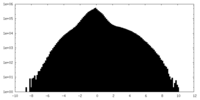
| 投影像・断面図 |
| ||||||||||||
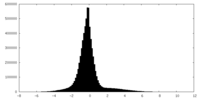
| 密度ヒストグラム |
-追加マップ: Unmasked full EM map resulting from subtomogram averaging...
| ファイル | emd_20504_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unmasked full EM map resulting from subtomogram averaging in emClarity of flagellar filaments purified from wildtype Leptospira biflexa serovar Patoc strain Patoc 1. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
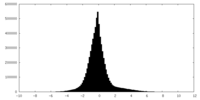
| 密度ヒストグラム |
-追加マップ: Masked half map 2 resulting from subtomogram averaging...
| ファイル | emd_20504_additional_3.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Masked half map 2 resulting from subtomogram averaging in emClarity of flagellar filaments purified from wildtype Leptospira biflexa serovar Patoc strain Patoc 1. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Symmetrised map generated by rotational symmetrization of the...
| ファイル | emd_20504_additional_4.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Symmetrised map generated by rotational symmetrization of the L. biflexa Patoc WT filament core resulting from subtomogram averaging. Map used to calculate helical parameters for FlaB core model. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Unmasked-half map 1 resulting from subtomogram averaging in...
| ファイル | emd_20504_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unmasked-half map 1 resulting from subtomogram averaging in emClarity of flagellar filaments purified from wildtype Leptospira biflexa serovar Patoc strain Patoc 1. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Unmasked-half map 2 resulting from subtomogram averaging in...
| ファイル | emd_20504_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unmasked-half map 2 resulting from subtomogram averaging in emClarity of flagellar filaments purified from wildtype Leptospira biflexa serovar Patoc strain Patoc 1. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Flagellar filament purified from the periplasm of the Spirochete ...
| 全体 | 名称: Flagellar filament purified from the periplasm of the Spirochete bacterium, Leptospira biflexa |
|---|---|
| 要素 |
|
-超分子 #1: Flagellar filament purified from the periplasm of the Spirochete ...
| 超分子 | 名称: Flagellar filament purified from the periplasm of the Spirochete bacterium, Leptospira biflexa タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: Filaments were purified from Leptospira biflexa wild type cells. |
|---|---|
| 由来(天然) | 生物種:  Leptospira biflexa serovar Patoc strain 'Patoc 1 (Paris)' (バクテリア) Leptospira biflexa serovar Patoc strain 'Patoc 1 (Paris)' (バクテリア)Organelle: flagellar filament / 細胞中の位置: periplasm |
-分子 #1: Flagellin B1 (FlaB1)
| 分子 | 名称: Flagellin B1 (FlaB1) / タイプ: protein_or_peptide / ID: 1 / コピー数: 84 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Leptospira biflexa serovar Patoc (strain Patoc 1 / ATCC 23582 / Paris) (バクテリア) Leptospira biflexa serovar Patoc (strain Patoc 1 / ATCC 23582 / Paris) (バクテリア)株: Patoc 1 / ATCC 23582 / Paris / 細胞: bacteria |
| 分子量 | 理論値: 29.467033 KDa |
| 配列 | 文字列: AAINSHRVLK FQNEEVSKNM EKLSSGMRIN RAGDDASGLA VSEKMRTQVN GLRQAERNTE DGMSLIQTTE GFLQESNDII QRIRTLAIQ SSNGIYTEED RQMIQVEVSQ LIDEVDRIAS QAEFNKMNLL QGDFARGSRA TSMWFHIGPN MHQRERVFIA T MTARSLNL ...文字列: AAINSHRVLK FQNEEVSKNM EKLSSGMRIN RAGDDASGLA VSEKMRTQVN GLRQAERNTE DGMSLIQTTE GFLQESNDII QRIRTLAIQ SSNGIYTEED RQMIQVEVSQ LIDEVDRIAS QAEFNKMNLL QGDFARGSRA TSMWFHIGPN MHQRERVFIA T MTARSLNL KGQSGELLSL STADKSNDAI GTLDAALTRI SKQRANLGAY FNRLEHAAKG LMNAYENTQA SESRIRDADM AE ETVAFTK NQILVQSGTA MLAQANVR UniProtKB: Flagellin |
-分子 #2: Flagellar coiling protein A (FcpA)
| 分子 | 名称: Flagellar coiling protein A (FcpA) / タイプ: protein_or_peptide / ID: 2 / コピー数: 47 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Leptospira biflexa serovar Patoc (strain Patoc 1 / ATCC 23582 / Paris) (バクテリア) Leptospira biflexa serovar Patoc (strain Patoc 1 / ATCC 23582 / Paris) (バクテリア)株: Patoc 1 / ATCC 23582 / Paris / 細胞: bacteria |
| 分子量 | 理論値: 28.931002 KDa |
| 配列 | 文字列: LTEDQKKKKK EIMEQESLWK NPDFKGYNKT FQELHQLSKT FANNQFRLAL SNYQSGVNTI MKNRDWVEQY RKEEAEKKRL DEKWYWQKV DRKAREERVV YREKMKAKQD ALNYFSKAIN HLDEIKNPDL RERPEFKRLL SDVYRSWIMA EYDLQNLPQT I PILELYIE ...文字列: LTEDQKKKKK EIMEQESLWK NPDFKGYNKT FQELHQLSKT FANNQFRLAL SNYQSGVNTI MKNRDWVEQY RKEEAEKKRL DEKWYWQKV DRKAREERVV YREKMKAKQD ALNYFSKAIN HLDEIKNPDL RERPEFKRLL SDVYRSWIMA EYDLQNLPQT I PILELYIE IDDNEKEYPA HKYLASAYSF EENMIKKTKG PDDMLFKYRY KKNVHLLRAT ELKYGKDSPE YKHIVNVIN UniProtKB: Uncharacterized protein |
-分子 #3: Flagellar coiling protein B (FcpB)
| 分子 | 名称: Flagellar coiling protein B (FcpB) / タイプ: protein_or_peptide / ID: 3 / コピー数: 32 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Leptospira biflexa serovar Patoc (strain Patoc 1 / ATCC 23582 / Paris) (バクテリア) Leptospira biflexa serovar Patoc (strain Patoc 1 / ATCC 23582 / Paris) (バクテリア)株: Patoc 1 / ATCC 23582 / Paris / 細胞: bacteria |
| 分子量 | 理論値: 25.539666 KDa |
| 配列 | 文字列: SGKSMADTEK ELDDNISEVN KRLRLHTVLF KMKVRTLPHK TVLYKGKPSA DGERCEAADK QEAQDNTCLH LEVFDFVGSE DGKSSKNLG AKFKKMELFF EGSNNADPDP RKEQPRNLTK IRTYIYQNNF LLEDKVISVI ADVAPNGEPA HNDKIELFYQ H DDYPVWGT ...文字列: SGKSMADTEK ELDDNISEVN KRLRLHTVLF KMKVRTLPHK TVLYKGKPSA DGERCEAADK QEAQDNTCLH LEVFDFVGSE DGKSSKNLG AKFKKMELFF EGSNNADPDP RKEQPRNLTK IRTYIYQNNF LLEDKVISVI ADVAPNGEPA HNDKIELFYQ H DDYPVWGT PETPSEKGVG KYILSNVENT KSNPIRNNFK KQFYFKNLDY FDKLFTKIFD YND UniProtKB: Uncharacterized protein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 濃度 | 0.5 mg/mL |
|---|---|
| 緩衝液 | pH: 6.8 / 詳細: Tris-HCl or ddH20, pH6.8 with <1% sodium azide as a preservative. FIDUCIAL MARKERS: Prior to vitrification, 1 uL of 6x concentrated Gold Tracer beads (10 nm colloidal gold) were mixed with 2 uL of purified flagella. To each grid, 3 uL of this mixture were applied. |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 支持フィルム - Film thickness: 600 / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 時間: 30 sec. / 前処理 - 雰囲気: OTHER 詳細: Model 950 Solarus Advanced Plasma System manufactured by GATAN. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 291 K / 装置: FEI VITROBOT MARK III 詳細: 2 minute incubation time; blot time of 6-7.5 seconds; and blot offset of -2 mm. |
| 詳細 | Leptospira biflexa serovar Patoc strain Patoc I wild type, fcpA, and fcpB mutant cells cultured in Ellinghausen-McCullough-Johnson-Harris liquid medium until they reached logarithmic phase at 30C. The cells were pelleted and the periplasmic flagellar filaments were purified as described in Wunder et al., 2016; Wunder et al., 2018. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI POLARA 300 |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 3710 pixel / デジタル化 - サイズ - 縦: 3838 pixel / デジタル化 - 画像ごとのフレーム数: 1-12 / 撮影したグリッド数: 1 / 平均露光時間: 1.2 sec. / 平均電子線量: 1.7 e/Å2 詳細: Tilt series acquisition. A total of ~35 tilt angles per tilt stack were acquired. The total dose was ~ 60 e/Angstrom2. |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 19230 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.0 mm / 最大 デフォーカス(公称値): 5.0 µm / 最小 デフォーカス(公称値): 3.0 µm / 倍率(公称値): 15000 |
| 試料ステージ | 試料ホルダーモデル: SIDE ENTRY, EUCENTRIC / ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Tecnai Polara / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル |
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 詳細 | Eliminate minor clashes between FlaB1, FcpA, and FcpB | ||||||||
| 精密化 | 空間: RECIPROCAL / プロトコル: RIGID BODY FIT / 当てはまり具合の基準: Correlation coefficient | ||||||||
| 得られたモデル |  PDB-6pwb: |
 ムービー
ムービー コントローラー
コントローラー











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)