+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-20267 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of LbCas12a-crRNA: AcrVA4 (2:2 complex) | |||||||||
 マップデータ マップデータ | 3D Reconstruction (State II) | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | CRISPR-Cas / anti-CRISPR / Cas12a / Cpf1 / LbCas12a / AcrVA4 / RNA BINDING PROTEIN-RNA complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Lachnospiraceae bacterium ND2006 (バクテリア) / Lachnospiraceae bacterium ND2006 (バクテリア) /  Moraxella bovoculi (バクテリア) Moraxella bovoculi (バクテリア) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.9 Å | |||||||||
 データ登録者 データ登録者 | Knott GJ / Liu JJ | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 | ジャーナル: Acta Crystallogr D Biol Crystallogr / 年: 2010 タイトル: PHENIX: a comprehensive Python-based system for macromolecular structure solution. 著者: Paul D Adams / Pavel V Afonine / Gábor Bunkóczi / Vincent B Chen / Ian W Davis / Nathaniel Echols / Jeffrey J Headd / Li-Wei Hung / Gary J Kapral / Ralf W Grosse-Kunstleve / Airlie J McCoy ...著者: Paul D Adams / Pavel V Afonine / Gábor Bunkóczi / Vincent B Chen / Ian W Davis / Nathaniel Echols / Jeffrey J Headd / Li-Wei Hung / Gary J Kapral / Ralf W Grosse-Kunstleve / Airlie J McCoy / Nigel W Moriarty / Robert Oeffner / Randy J Read / David C Richardson / Jane S Richardson / Thomas C Terwilliger / Peter H Zwart /  要旨: Macromolecular X-ray crystallography is routinely applied to understand biological processes at a molecular level. However, significant time and effort are still required to solve and complete many ...Macromolecular X-ray crystallography is routinely applied to understand biological processes at a molecular level. However, significant time and effort are still required to solve and complete many of these structures because of the need for manual interpretation of complex numerical data using many software packages and the repeated use of interactive three-dimensional graphics. PHENIX has been developed to provide a comprehensive system for macromolecular crystallographic structure solution with an emphasis on the automation of all procedures. This has relied on the development of algorithms that minimize or eliminate subjective input, the development of algorithms that automate procedures that are traditionally performed by hand and, finally, the development of a framework that allows a tight integration between the algorithms. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_20267.map.gz emd_20267.map.gz | 141.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-20267-v30.xml emd-20267-v30.xml emd-20267.xml emd-20267.xml | 21.9 KB 21.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_20267.png emd_20267.png | 80.5 KB | ||
| その他 |  emd_20267_half_map_1.map.gz emd_20267_half_map_1.map.gz emd_20267_half_map_2.map.gz emd_20267_half_map_2.map.gz | 139 MB 139 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-20267 http://ftp.pdbj.org/pub/emdb/structures/EMD-20267 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20267 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20267 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_20267_validation.pdf.gz emd_20267_validation.pdf.gz | 1.2 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_20267_full_validation.pdf.gz emd_20267_full_validation.pdf.gz | 1.2 MB | 表示 | |
| XML形式データ |  emd_20267_validation.xml.gz emd_20267_validation.xml.gz | 14.4 KB | 表示 | |
| CIF形式データ |  emd_20267_validation.cif.gz emd_20267_validation.cif.gz | 17.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20267 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20267 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20267 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20267 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_20267.map.gz / 形式: CCP4 / 大きさ: 149.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_20267.map.gz / 形式: CCP4 / 大きさ: 149.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 3D Reconstruction (State II) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.9 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-ハーフマップ: 3D Reconstruction (State II) Half Map A
| ファイル | emd_20267_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 3D Reconstruction (State II) Half Map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
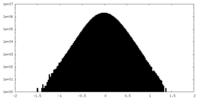
| 密度ヒストグラム |
-ハーフマップ: 3D Reconstruction (State II) Half Map B
| ファイル | emd_20267_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 3D Reconstruction (State II) Half Map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
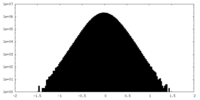
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : LbCas12a-crRNA-AcrVA4 Complex (2:2, State I)
| 全体 | 名称: LbCas12a-crRNA-AcrVA4 Complex (2:2, State I) |
|---|---|
| 要素 |
|
-超分子 #1: LbCas12a-crRNA-AcrVA4 Complex (2:2, State I)
| 超分子 | 名称: LbCas12a-crRNA-AcrVA4 Complex (2:2, State I) / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 詳細: Homodimeric AcrVA4 bound to two copies of the LbCas12a-crRNA complex. |
|---|
-超分子 #2: LbCas12a
| 超分子 | 名称: LbCas12a / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #2 |
|---|---|
| 由来(天然) | 生物種:  Lachnospiraceae bacterium ND2006 (バクテリア) Lachnospiraceae bacterium ND2006 (バクテリア) |
-超分子 #3: crRNA
| 超分子 | 名称: crRNA / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #3 |
|---|---|
| 由来(天然) | 生物種:  Lachnospiraceae bacterium ND2006 (バクテリア) Lachnospiraceae bacterium ND2006 (バクテリア) |
-超分子 #4: AcrVA4
| 超分子 | 名称: AcrVA4 / タイプ: complex / ID: 4 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Moraxella bovoculi (バクテリア) Moraxella bovoculi (バクテリア) |
-分子 #1: anti-CRISPR VA4
| 分子 | 名称: anti-CRISPR VA4 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Moraxella bovoculi (バクテリア) Moraxella bovoculi (バクテリア) |
| 分子量 | 理論値: 27.641422 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: SNAMYEIKLN DTLIHQTDDR VNAFVAYRYL LRRGDLPKCE NIARMYYDGK VIKTDVIDHD SVHSDEQAKV SNNDIIKMAI SELGVNNFK SLIKKQGYPF SNGHINSWFT DDPVKSKTMH NDEMYLVVQA LIRACIIKEI DLYTEQLYNI IKSLPYDKRP N VVYSDQPL ...文字列: SNAMYEIKLN DTLIHQTDDR VNAFVAYRYL LRRGDLPKCE NIARMYYDGK VIKTDVIDHD SVHSDEQAKV SNNDIIKMAI SELGVNNFK SLIKKQGYPF SNGHINSWFT DDPVKSKTMH NDEMYLVVQA LIRACIIKEI DLYTEQLYNI IKSLPYDKRP N VVYSDQPL DPNNLDLSEP ELWAEQVGEC MRYAHNDQPC FYIGSTKREL RVNYIVPVIG VRDEIERVMT LEEVRNLHK UniProtKB: Uncharacterized protein |
-分子 #2: Cas12a
| 分子 | 名称: Cas12a / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Lachnospiraceae bacterium ND2006 (バクテリア) Lachnospiraceae bacterium ND2006 (バクテリア) |
| 分子量 | 理論値: 144.160609 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: SNAMSKLEKF TNCYSLSKTL RFKAIPVGKT QENIDNKRLL VEDEKRAEDY KGVKKLLDRY YLSFINDVLH SIKLKNLNNY ISLFRKKTR TEKENKELEN LEINLRKEIA KAFKGNEGYK SLFKKDIIET ILPEFLDDKD EIALVNSFNG FTTAFTGFFD N RENMFSEE ...文字列: SNAMSKLEKF TNCYSLSKTL RFKAIPVGKT QENIDNKRLL VEDEKRAEDY KGVKKLLDRY YLSFINDVLH SIKLKNLNNY ISLFRKKTR TEKENKELEN LEINLRKEIA KAFKGNEGYK SLFKKDIIET ILPEFLDDKD EIALVNSFNG FTTAFTGFFD N RENMFSEE AKSTSIAFRC INENLTRYIS NMDIFEKVDA IFDKHEVQEI KEKILNSDYD VEDFFEGEFF NFVLTQEGID VY NAIIGGF VTESGEKIKG LNEYINLYNQ KTKQKLPKFK PLYKQVLSDR ESLSFYGEGY TSDEEVLEVF RNTLNKNSEI FSS IKKLEK LFKNFDEYSS AGIFVKNGPA ISTISKDIFG EWNVIRDKWN AEYDDIHLKK KAVVTEKYED DRRKSFKKIG SFSL EQLQE YADADLSVVE KLKEIIIQKV DEIYKVYGSS EKLFDADFVL EKSLKKNDAV VAIMKDLLDS VKSFENYIKA FFGEG KETN RDESFYGDFV LAYDILLKVD HIYDAIRNYV TQKPYSKDKF KLYFQNPQFM GGWDKDKETD YRATILRYGS KYYLAI MDK KYAKCLQKID KDDVNGNYEK INYKLLPGPN KMLPKVFFSK KWMAYYNPSE DIQKIYKNGT FKKGDMFNLN DCHKLID FF KDSISRYPKW SNAYDFNFSE TEKYKDIAGF YREVEEQGYK VSFESASKKE VDKLVEEGKL YMFQIYNKDF SDKSHGTP N LHTMYFKLLF DENNHGQIRL SGGAELFMRR ASLKKEELVV HPANSPIANK NPDNPKKTTT LSYDVYKDKR FSEDQYELH IPIAINKCPK NIFKINTEVR VLLKHDDNPY VIGIDRGERN LLYIVVVDGK GNIVEQYSLN EIINNFNGIR IKTDYHSLLD KKEKERFEA RQNWTSIENI KELKAGYISQ VVHKICELVE KYDAVIALED LNSGFKNSRV KVEKQVYQKF EKMLIDKLNY M VDKKSNPC ATGGALKGYQ ITNKFESFKS MSTQNGFIFY IPAWLTSKID PSTGFVNLLK TKYTSIADSK KFISSFDRIM YV PEEDLFE FALDYKNFSR TDADYIKKWK LYSYGNRIRI FRNPKKNNVF DWEEVCLTSA YKELFNKYGI NYQQGDIRAL LCE QSDKAF YSSFMALMSL MLQMRNSITG RTDVDFLISP VKNSDGIFYD SRNYEAQENA ILPKNADANG AYNIARKVLW AIGQ FKKAE DEKLDKVKIA ISNKEWLEYA QTSVKH |
-分子 #3: mature crRNA
| 分子 | 名称: mature crRNA / タイプ: rna / ID: 3 / コピー数: 2 |
|---|---|
| 由来(天然) | 生物種:  Lachnospiraceae bacterium ND2006 (バクテリア) Lachnospiraceae bacterium ND2006 (バクテリア) |
| 分子量 | 理論値: 12.815634 KDa |
| 配列 | 文字列: AAUUUCUACU AAGUGUAGAU AAAGUGCUCA UCAUUGGAAA |
-分子 #4: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 4 / コピー数: 4 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 構成要素:
| |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| グリッド | 詳細: unspecified | |||||||||||||||
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 平均電子線量: 42.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: PDB ENTRY PDBモデル - PDB ID: |
|---|---|
| 最終 再構成 | 使用したクラス数: 1 / 想定した対称性 - 点群: C1 (非対称) / 解像度のタイプ: BY AUTHOR / 解像度: 4.9 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 79787 |
| 初期 角度割当 | タイプ: NOT APPLICABLE |
| 最終 角度割当 | タイプ: NOT APPLICABLE |
 ムービー
ムービー コントローラー
コントローラー















 Z
Z Y
Y X
X