+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | cryoEM structure of Acs1 filament determined by FilamentID | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | metabolic enzyme / filament / cryoEM / CYTOSOLIC PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報acetate-CoA ligase / acetate-CoA ligase activity / acetyl-CoA biosynthetic process from acetate / AMP binding / mitochondrion / ATP binding / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |   | |||||||||
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 3.5 Å | |||||||||
 データ登録者 データ登録者 | Hugener J / Xu J / Wettstein R / Ioannidi L / Velikov D / Wollweber F / Henggeler A / Matos J / Pilhofer M | |||||||||
| 資金援助 |  スイス, European Union, 2件 スイス, European Union, 2件
| |||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2024 ジャーナル: Cell / 年: 2024タイトル: FilamentID reveals the composition and function of metabolic enzyme polymers during gametogenesis. 著者: Jannik Hugener / Jingwei Xu / Rahel Wettstein / Lydia Ioannidi / Daniel Velikov / Florian Wollweber / Adrian Henggeler / Joao Matos / Martin Pilhofer /   要旨: Gamete formation and subsequent offspring development often involve extended phases of suspended cellular development or even dormancy. How cells adapt to recover and resume growth remains poorly ...Gamete formation and subsequent offspring development often involve extended phases of suspended cellular development or even dormancy. How cells adapt to recover and resume growth remains poorly understood. Here, we visualized budding yeast cells undergoing meiosis by cryo-electron tomography (cryoET) and discovered elaborate filamentous assemblies decorating the nucleus, cytoplasm, and mitochondria. To determine filament composition, we developed a "filament identification" (FilamentID) workflow that combines multiscale cryoET/cryo-electron microscopy (cryoEM) analyses of partially lysed cells or organelles. FilamentID identified the mitochondrial filaments as being composed of the conserved aldehyde dehydrogenase Ald4 and the nucleoplasmic/cytoplasmic filaments as consisting of acetyl-coenzyme A (CoA) synthetase Acs1. Structural characterization further revealed the mechanism underlying polymerization and enabled us to genetically perturb filament formation. Acs1 polymerization facilitates the recovery of chronologically aged spores and, more generally, the cell cycle re-entry of starved cells. FilamentID is broadly applicable to characterize filaments of unknown identity in diverse cellular contexts. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_19548.map.gz emd_19548.map.gz | 9.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-19548-v30.xml emd-19548-v30.xml emd-19548.xml emd-19548.xml | 17.2 KB 17.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_19548.png emd_19548.png | 60.3 KB | ||
| マスクデータ |  emd_19548_msk_1.map emd_19548_msk_1.map | 52.7 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-19548.cif.gz emd-19548.cif.gz | 5.8 KB | ||
| その他 |  emd_19548_additional_1.map.gz emd_19548_additional_1.map.gz emd_19548_half_map_1.map.gz emd_19548_half_map_1.map.gz emd_19548_half_map_2.map.gz emd_19548_half_map_2.map.gz | 46.9 MB 40.7 MB 40.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-19548 http://ftp.pdbj.org/pub/emdb/structures/EMD-19548 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-19548 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-19548 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_19548_validation.pdf.gz emd_19548_validation.pdf.gz | 1002.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_19548_full_validation.pdf.gz emd_19548_full_validation.pdf.gz | 1002.4 KB | 表示 | |
| XML形式データ |  emd_19548_validation.xml.gz emd_19548_validation.xml.gz | 11.8 KB | 表示 | |
| CIF形式データ |  emd_19548_validation.cif.gz emd_19548_validation.cif.gz | 13.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-19548 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-19548 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-19548 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-19548 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8rwjMC  8rwkC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_19548.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_19548.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.07 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_19548_msk_1.map emd_19548_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
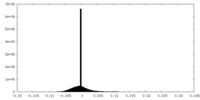
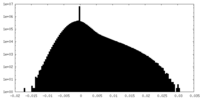
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: cryoEM map further improved by deepEMhancer
| ファイル | emd_19548_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | cryoEM map further improved by deepEMhancer | ||||||||||||
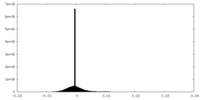
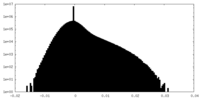
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_19548_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_19548_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Acetyl-CoA synthetase 1 filament from spread meiotic yeast sphero...
| 全体 | 名称: Acetyl-CoA synthetase 1 filament from spread meiotic yeast spheroplasts |
|---|---|
| 要素 |
|
-超分子 #1: Acetyl-CoA synthetase 1 filament from spread meiotic yeast sphero...
| 超分子 | 名称: Acetyl-CoA synthetase 1 filament from spread meiotic yeast spheroplasts タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Acetyl-coenzyme A synthetase
| 分子 | 名称: Acetyl-coenzyme A synthetase / タイプ: protein_or_peptide / ID: 1 / コピー数: 9 / 光学異性体: LEVO / EC番号: acetate-CoA ligase |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 79.27057 KDa |
| 配列 | 文字列: MSPSAVQSSK LEEQSSEIDK LKAKMSQSAS TAQQKKEHEY EHLTSVKIVP QRPISDRLQP AIATHYSPHL DGLQDYQRLH KESIEDPAK FFGSKATQFL NWSKPFDKVF IPDSKTGRPS FQNNAWFLNG QLNACYNCVD RHALKTPNKK AIIFEGDEPG Q GYSITYKE ...文字列: MSPSAVQSSK LEEQSSEIDK LKAKMSQSAS TAQQKKEHEY EHLTSVKIVP QRPISDRLQP AIATHYSPHL DGLQDYQRLH KESIEDPAK FFGSKATQFL NWSKPFDKVF IPDSKTGRPS FQNNAWFLNG QLNACYNCVD RHALKTPNKK AIIFEGDEPG Q GYSITYKE LLEEVCQVAQ VLTYSMGVRK GDTVAVYMPM VPEAIITLLA ISRIGAIHSV VFAGFSSNSL RDRINDGDSK VV ITTDESN RGGKVIETKR IVDDALRETP GVRHVLVYRK TNNPSVAFHA PRDLDWATEK KKYKTYYPCT PVDSEDPLFL LYT SGSTGA PKGVQHSTAG YLLGALLTMR YTFDTHQEDV FFTAGDIGWI TGHTYVVYGP LLYGCATLVF EGTPAYPNYS RYWD IIDEH KVTQFYVAPT ALRLLKRAGD SYIENHSLKS LRCLGSVGEP IAAEVWEWYS EKIGKNEIPI VDTYWQTESG SHLVT PLAG GVTPMKPGSA SFPFFGIDAV VLDPNTGEEL NTSHAEGVLA VKAAWPSFAR TIWKNHDRYL DTYLNPYPGY YFTGDG AAK DKDGYIWILG RVDDVVNVSG HRLSTAEIEA AIIEDPIVAE CAVVGFNDDL TGQAVAAFVV LKNKSNWSTA TDDELQD IK KHLVFTVRKD IGPFAAPKLI ILVDDLPKTR SGKIMRRILR KILAGESDQL GDVSTLSNPG IVRHLIDSVK L UniProtKB: Acetyl-coenzyme A synthetase |
-分子 #2: [[(2~{R},3~{S},4~{R},5~{R})-5-(6-aminopurin-9-yl)-3,4-bis(oxidany...
| 分子 | 名称: [[(2~{R},3~{S},4~{R},5~{R})-5-(6-aminopurin-9-yl)-3,4-bis(oxidanyl)oxolan-2-yl]methoxy-oxidanyl-phosphoryl] ethanoate タイプ: ligand / ID: 2 / コピー数: 9 / 式: 6R9 |
|---|---|
| 分子量 | 理論値: 389.258 Da |
| Chemical component information | 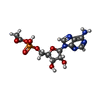 ChemComp-6R9: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 緩衝液 | pH: 6.4 |
|---|---|
| 凍結 | 凍結剤: ETHANE-PROPANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD 最大 デフォーカス(公称値): 2.8000000000000003 µm 最小 デフォーカス(公称値): 1.2 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 最終 再構成 | 想定した対称性 - らせんパラメータ - Δz: 53.61 Å 想定した対称性 - らせんパラメータ - ΔΦ: 13.03 ° 想定した対称性 - らせんパラメータ - 軸対称性: C3 (3回回転対称) 解像度のタイプ: BY AUTHOR / 解像度: 3.5 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: RELION / 使用した粒子像数: 17169 |
|---|---|
| 初期モデル | モデルのタイプ: OTHER 詳細: the initial model is determined from sub-tomogram averaged volume of spread spheroplast. |
| 最終 角度割当 | タイプ: NOT APPLICABLE |
 ムービー
ムービー コントローラー
コントローラー















 Z
Z Y
Y X
X