+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM reconstruction of VP5*/VP8* assembly from SA11 Rotavirus Tripsinized Triple Layered Particle | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Rotavirus / dsRNA virus / VIRUS | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報host cell rough endoplasmic reticulum / host cytoskeleton / viral outer capsid / permeabilization of host organelle membrane involved in viral entry into host cell / host cell endoplasmic reticulum-Golgi intermediate compartment / virion attachment to host cell / host cell plasma membrane / membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Rotavirus (ウイルス) / Rotavirus (ウイルス) /  Rotavirus A (ロタウイルス A) Rotavirus A (ロタウイルス A) | |||||||||
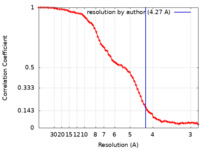
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.27 Å | |||||||||
 データ登録者 データ登録者 | Asensio-Cob D / Perez-Mata C / Gomez-Blanco J / Vargas J / Rodriguez JM / Luque D | |||||||||
| 資金援助 |  スペイン, 1件 スペイン, 1件
| |||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Structural basis of rotavirus spike proteolytic activation 著者: Asensio-Cob D / Perez-Mata C / Gomez-Blanco J / Vargas J / Rodriguez JM / Luque D | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_18655.map.gz emd_18655.map.gz | 665.8 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-18655-v30.xml emd-18655-v30.xml emd-18655.xml emd-18655.xml | 15.7 KB 15.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_18655_fsc.xml emd_18655_fsc.xml | 9.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_18655.png emd_18655.png | 64.1 KB | ||
| Filedesc metadata |  emd-18655.cif.gz emd-18655.cif.gz | 5.9 KB | ||
| その他 |  emd_18655_half_map_1.map.gz emd_18655_half_map_1.map.gz emd_18655_half_map_2.map.gz emd_18655_half_map_2.map.gz | 23.3 MB 23.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-18655 http://ftp.pdbj.org/pub/emdb/structures/EMD-18655 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18655 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18655 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_18655_validation.pdf.gz emd_18655_validation.pdf.gz | 630 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_18655_full_validation.pdf.gz emd_18655_full_validation.pdf.gz | 629.6 KB | 表示 | |
| XML形式データ |  emd_18655_validation.xml.gz emd_18655_validation.xml.gz | 13.1 KB | 表示 | |
| CIF形式データ |  emd_18655_validation.cif.gz emd_18655_validation.cif.gz | 18 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18655 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18655 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18655 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18655 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_18655.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_18655.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.43 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Rotavirus A
| 全体 | 名称:  Rotavirus A (ロタウイルス A) Rotavirus A (ロタウイルス A) |
|---|---|
| 要素 |
|
-超分子 #1: Rotavirus A
| 超分子 | 名称: Rotavirus A / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all / 詳細: Rotavirus SA11 Non-tripsinized spike / NCBI-ID: 28875 / 生物種: Rotavirus A / Sci species strain: SA11 / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:  Chlorocebus aethiops (ミドリザル) Chlorocebus aethiops (ミドリザル) |
| 分子量 | 理論値: 92 MDa |
| ウイルス殻 | Shell ID: 1 / 名称: TLP / 直径: 700.0 Å |
-分子 #1: Outer capsid protein VP4
| 分子 | 名称: Outer capsid protein VP4 / タイプ: protein_or_peptide / ID: 1 / 詳細: Outer capsid protein VP4 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Rotavirus (ウイルス) Rotavirus (ウイルス) |
| 分子量 | 理論値: 86.749891 KDa |
| 組換発現 | 生物種:  Chlorocebus aethiops (ミドリザル) Chlorocebus aethiops (ミドリザル) |
| 配列 | 文字列: MASLIYRQLL TNSYTVDLSD EIQEIGSTKS QNVTINPGPF AQTGYAPVNW GPGEINDSTT VEPLLDGPYQ PTTFNPPVDY WMLLAPTTP GVIVEGTNNT DRWLATILIE PNVQSENRTY TIFGIQEQLT VSNTSQDQWK FIDVVKTTAN GSIGQYGSLL S SPKLYAVM ...文字列: MASLIYRQLL TNSYTVDLSD EIQEIGSTKS QNVTINPGPF AQTGYAPVNW GPGEINDSTT VEPLLDGPYQ PTTFNPPVDY WMLLAPTTP GVIVEGTNNT DRWLATILIE PNVQSENRTY TIFGIQEQLT VSNTSQDQWK FIDVVKTTAN GSIGQYGSLL S SPKLYAVM KHNEKLYTYE GQTPNARTGH YSTTNYDSVN MTAFCDFYII PRSEESKCTE YINNGLPPIQ NTRNVVPLSL TA RDVIHYR AQANEDIVIS KTSLWKEMQY NRDITIRFKF ANTIIKSGGL GYKWSEISFK PANYQYTYTR DGEEVTAHTT CSV NGVNDF SFNGGSLPTD FVVSKFEVIK ENSYVYIDYW DDSQAFRNVV YVRSLAANLN SVMCTGGSYN FSLPVGQWPV LTGG AVSLH SAGVTLSTQF TDFVSLNSLR FRFRLAVEEP HFKLTRTRLD RLYGLPAADP NNGKEYYEIA GRFSLISLVP SNDDY QTPI ANSVTVRQDL ERQLGELREE FNALSQEIAM SQLIDLALLP LDMFSMFSGI KSTIDAAKSM ATNVMKKFKK SGLANS VST LTDSLSDAAS SISRGSSIRS IGSSASAWTD VSTQITDISS SVSSVSTQTS TISRRLRLKE MATQTEGMNF DDISAAV LK TKIDKSTQIS PNTIPDIVTE ASEKFIPNRA YRVINNDDVF EAGIDGKFFA YKVDTFEEIP FDVQKFADLV TDSPVISA I IDFKTLKNLN DNYGITKQQA FNLLRSDPRV LREFINQDNP IIRNRIEQLI MQCRL UniProtKB: Outer capsid protein VP4 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 295 K / 装置: LEICA EM GP |
| 詳細 | Rotavirus SA11 Tripsinized spike |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: INTEGRATING / 実像数: 2368 / 平均電子線量: 39.9 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 0.75 µm / 倍率(公称値): 58000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)