+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Catalytic module of human CTLH E3 ligase bound to multiphosphorylated UBE2H~ubiquitin | ||||||||||||
 マップデータ マップデータ | |||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | E3 ubiquitin ligase / E2 ubiquitin-conjugating enzyme / phosphorylation / CTLH / GID / UBE2H / LIGASE | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報GID complex / actomyosin contractile ring / enucleate erythrocyte development / negative regulation of myeloid cell apoptotic process / (E3-independent) E2 ubiquitin-conjugating enzyme / protein K11-linked ubiquitination / E2 ubiquitin-conjugating enzyme / erythrocyte maturation / ubiquitin conjugating enzyme activity / ubiquitin ligase complex ...GID complex / actomyosin contractile ring / enucleate erythrocyte development / negative regulation of myeloid cell apoptotic process / (E3-independent) E2 ubiquitin-conjugating enzyme / protein K11-linked ubiquitination / E2 ubiquitin-conjugating enzyme / erythrocyte maturation / ubiquitin conjugating enzyme activity / ubiquitin ligase complex / protein K48-linked ubiquitination / Maturation of protein E / Maturation of protein E / ER Quality Control Compartment (ERQC) / cytoskeleton organization / Myoclonic epilepsy of Lafora / FLT3 signaling by CBL mutants / Prevention of phagosomal-lysosomal fusion / IRAK2 mediated activation of TAK1 complex / Alpha-protein kinase 1 signaling pathway / Glycogen synthesis / IRAK1 recruits IKK complex / IRAK1 recruits IKK complex upon TLR7/8 or 9 stimulation / Membrane binding and targetting of GAG proteins / Endosomal Sorting Complex Required For Transport (ESCRT) / Regulation of TBK1, IKKε (IKBKE)-mediated activation of IRF3, IRF7 / Negative regulation of FLT3 / PTK6 Regulates RTKs and Their Effectors AKT1 and DOK1 / Regulation of TBK1, IKKε-mediated activation of IRF3, IRF7 upon TLR3 ligation / Constitutive Signaling by NOTCH1 HD Domain Mutants / IRAK2 mediated activation of TAK1 complex upon TLR7/8 or 9 stimulation / NOTCH2 Activation and Transmission of Signal to the Nucleus / TICAM1,TRAF6-dependent induction of TAK1 complex / TICAM1-dependent activation of IRF3/IRF7 / APC/C:Cdc20 mediated degradation of Cyclin B / Regulation of FZD by ubiquitination / Downregulation of ERBB4 signaling / APC-Cdc20 mediated degradation of Nek2A / p75NTR recruits signalling complexes / InlA-mediated entry of Listeria monocytogenes into host cells / TRAF6 mediated IRF7 activation in TLR7/8 or 9 signaling / TRAF6-mediated induction of TAK1 complex within TLR4 complex / Regulation of pyruvate metabolism / Regulation of innate immune responses to cytosolic DNA / NF-kB is activated and signals survival / Downregulation of ERBB2:ERBB3 signaling / Pexophagy / NRIF signals cell death from the nucleus / VLDLR internalisation and degradation / Regulation of PTEN localization / regulation of mitotic cell cycle / Activated NOTCH1 Transmits Signal to the Nucleus / Synthesis of active ubiquitin: roles of E1 and E2 enzymes / Regulation of BACH1 activity / MAP3K8 (TPL2)-dependent MAPK1/3 activation / TICAM1, RIP1-mediated IKK complex recruitment / Translesion synthesis by REV1 / Activation of IRF3, IRF7 mediated by TBK1, IKKε (IKBKE) / Translesion synthesis by POLK / InlB-mediated entry of Listeria monocytogenes into host cell / Downregulation of TGF-beta receptor signaling / Josephin domain DUBs / JNK (c-Jun kinases) phosphorylation and activation mediated by activated human TAK1 / Regulation of activated PAK-2p34 by proteasome mediated degradation / Translesion synthesis by POLI / IKK complex recruitment mediated by RIP1 / Gap-filling DNA repair synthesis and ligation in GG-NER / PINK1-PRKN Mediated Mitophagy / TGF-beta receptor signaling in EMT (epithelial to mesenchymal transition) / TNFR1-induced NF-kappa-B signaling pathway / Autodegradation of Cdh1 by Cdh1:APC/C / APC/C:Cdc20 mediated degradation of Securin / N-glycan trimming in the ER and Calnexin/Calreticulin cycle / TCF dependent signaling in response to WNT / Asymmetric localization of PCP proteins / Regulation of NF-kappa B signaling / Ubiquitin-dependent degradation of Cyclin D / SCF-beta-TrCP mediated degradation of Emi1 / NIK-->noncanonical NF-kB signaling / activated TAK1 mediates p38 MAPK activation / Negative regulators of DDX58/IFIH1 signaling / TNFR2 non-canonical NF-kB pathway / AUF1 (hnRNP D0) binds and destabilizes mRNA / Regulation of signaling by CBL / NOTCH3 Activation and Transmission of Signal to the Nucleus / Vpu mediated degradation of CD4 / Assembly of the pre-replicative complex / Ubiquitin-Mediated Degradation of Phosphorylated Cdc25A / Degradation of DVL / Deactivation of the beta-catenin transactivating complex / Negative regulation of FGFR3 signaling / Cdc20:Phospho-APC/C mediated degradation of Cyclin A / Dectin-1 mediated noncanonical NF-kB signaling / Fanconi Anemia Pathway / Peroxisomal protein import / Negative regulation of FGFR2 signaling / Negative regulation of FGFR4 signaling / Regulation of TNFR1 signaling / Degradation of AXIN / Stabilization of p53 類似検索 - 分子機能 | ||||||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||||||||
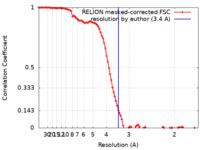
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.4 Å | ||||||||||||
 データ登録者 データ登録者 | Chrustowicz J / Sherpa D / Prabu RJ / Schulman BA | ||||||||||||
| 資金援助 |  ドイツ, European Union, 3件 ドイツ, European Union, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Mol Cell / 年: 2024 ジャーナル: Mol Cell / 年: 2024タイトル: Multisite phosphorylation dictates selective E2-E3 pairing as revealed by Ubc8/UBE2H-GID/CTLH assemblies. 著者: Jakub Chrustowicz / Dawafuti Sherpa / Jerry Li / Christine R Langlois / Eleftheria C Papadopoulou / D Tung Vu / Laura A Hehl / Özge Karayel / Viola Beier / Susanne von Gronau / Judith ...著者: Jakub Chrustowicz / Dawafuti Sherpa / Jerry Li / Christine R Langlois / Eleftheria C Papadopoulou / D Tung Vu / Laura A Hehl / Özge Karayel / Viola Beier / Susanne von Gronau / Judith Müller / J Rajan Prabu / Matthias Mann / Gary Kleiger / Arno F Alpi / Brenda A Schulman /   要旨: Ubiquitylation is catalyzed by coordinated actions of E3 and E2 enzymes. Molecular principles governing many important E3-E2 partnerships remain unknown, including those for RING-family GID/CTLH E3 ...Ubiquitylation is catalyzed by coordinated actions of E3 and E2 enzymes. Molecular principles governing many important E3-E2 partnerships remain unknown, including those for RING-family GID/CTLH E3 ubiquitin ligases and their dedicated E2, Ubc8/UBE2H (yeast/human nomenclature). GID/CTLH-Ubc8/UBE2H-mediated ubiquitylation regulates biological processes ranging from yeast metabolic signaling to human development. Here, cryoelectron microscopy (cryo-EM), biochemistry, and cell biology reveal this exquisitely specific E3-E2 pairing through an unconventional catalytic assembly and auxiliary interactions 70-100 Å away, mediated by E2 multisite phosphorylation. Rather than dynamic polyelectrostatic interactions reported for other ubiquitylation complexes, multiple Ubc8/UBE2H phosphorylation sites within acidic CK2-targeted sequences specifically anchor the E2 C termini to E3 basic patches. Positions of phospho-dependent interactions relative to the catalytic domains correlate across evolution. Overall, our data show that phosphorylation-dependent multivalency establishes a specific E3-E2 partnership, is antagonistic with dephosphorylation, rigidifies the catalytic centers within a flexing GID E3-substrate assembly, and facilitates substrate collision with ubiquitylation active sites. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_17713.map.gz emd_17713.map.gz | 6.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-17713-v30.xml emd-17713-v30.xml emd-17713.xml emd-17713.xml | 27.4 KB 27.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_17713_fsc.xml emd_17713_fsc.xml | 9.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_17713.png emd_17713.png | 106.6 KB | ||
| マスクデータ |  emd_17713_msk_1.map emd_17713_msk_1.map | 76.8 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-17713.cif.gz emd-17713.cif.gz | 7.8 KB | ||
| その他 |  emd_17713_additional_1.map.gz emd_17713_additional_1.map.gz emd_17713_half_map_1.map.gz emd_17713_half_map_1.map.gz emd_17713_half_map_2.map.gz emd_17713_half_map_2.map.gz | 66.3 MB 59.9 MB 59.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-17713 http://ftp.pdbj.org/pub/emdb/structures/EMD-17713 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17713 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17713 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_17713_validation.pdf.gz emd_17713_validation.pdf.gz | 720.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_17713_full_validation.pdf.gz emd_17713_full_validation.pdf.gz | 719.9 KB | 表示 | |
| XML形式データ |  emd_17713_validation.xml.gz emd_17713_validation.xml.gz | 16.8 KB | 表示 | |
| CIF形式データ |  emd_17713_validation.cif.gz emd_17713_validation.cif.gz | 22.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17713 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17713 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17713 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17713 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8pjnMC  8pmqC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_17713.map.gz / 形式: CCP4 / 大きさ: 76.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_17713.map.gz / 形式: CCP4 / 大きさ: 76.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.851 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_17713_msk_1.map emd_17713_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
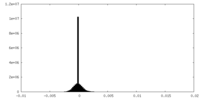
| 密度ヒストグラム |
-追加マップ: #1
| ファイル | emd_17713_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
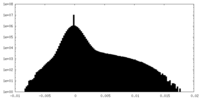
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_17713_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
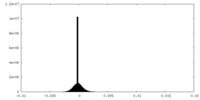
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_17713_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
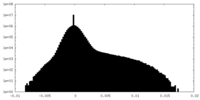
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Complex of human CTLHSR4 E3 ligase bound to engineered ARMC8-spec...
| 全体 | 名称: Complex of human CTLHSR4 E3 ligase bound to engineered ARMC8-specific VH and multiphosphorylated UBE2H~ubiquitin |
|---|---|
| 要素 |
|
-超分子 #1: Complex of human CTLHSR4 E3 ligase bound to engineered ARMC8-spec...
| 超分子 | 名称: Complex of human CTLHSR4 E3 ligase bound to engineered ARMC8-specific VH and multiphosphorylated UBE2H~ubiquitin タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 詳細: Map obtained by focus refinement over the catalytic module (RMND5A, MAEA) and UBE2H~ubiquitin |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 600 KDa |
-分子 #1: E3 ubiquitin-protein transferase RMND5A
| 分子 | 名称: E3 ubiquitin-protein transferase RMND5A / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO / EC番号: RING-type E3 ubiquitin transferase |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 44.043449 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MDQCVTVERE LEKVLHKFSG YGQLCERGLE ELIDYTGGLK HEILQSHGQD AELSGTLSLV LTQCCKRIKD TVQKLASDHK DIHSSVSRV GKAIDKNFDS DISSVGIDGC WQADSQRLLN EVMVEHFFRQ GMLDVAEELC QESGLSVDPS QKEPFVELNR I LEALKVRV ...文字列: MDQCVTVERE LEKVLHKFSG YGQLCERGLE ELIDYTGGLK HEILQSHGQD AELSGTLSLV LTQCCKRIKD TVQKLASDHK DIHSSVSRV GKAIDKNFDS DISSVGIDGC WQADSQRLLN EVMVEHFFRQ GMLDVAEELC QESGLSVDPS QKEPFVELNR I LEALKVRV LRPALEWAVS NREMLIAQNS SLEFKLHRLY FISLLMGGTT NQREALQYAK NFQPFALNHQ KDIQVLMGSL VY LRQGIEN SPYVHLLDAN QWADICDIFT RDACALLGLS VESPLSVSFS AGCVALPALI NIKAVIEQRQ CTGVWNQKDE LPI EVDLGK KCWYHSIFAC PILRQQTTDN NPPMKLVCGH IISRDALNKM FNGSKLKCPY CPMEQSPGDA KQIFF UniProtKB: E3 ubiquitin-protein transferase RMND5A |
-分子 #2: Ubiquitin-conjugating enzyme E2 H
| 分子 | 名称: Ubiquitin-conjugating enzyme E2 H / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO / EC番号: E2 ubiquitin-conjugating enzyme |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 21.260986 KDa |
| 組換発現 | 生物種:  Trichoplusia (蝶・蛾) Trichoplusia (蝶・蛾) |
| 配列 | 文字列: MSSPSPGKRR MDTDVVKLIE SKHEVTILGG LNEFVVKFYG PQGTPYEGGV WKVRVDLPDK YPFKSPSIGF MNKIFHPNID EASGTVKLD VINQTWTALY DLTNIFESFL PQLLAYPNPI DPLNGDAAAM YLHRPEEYKQ KIKEYIQKYA TEEALKEQEE G TGD(SEP) ...文字列: MSSPSPGKRR MDTDVVKLIE SKHEVTILGG LNEFVVKFYG PQGTPYEGGV WKVRVDLPDK YPFKSPSIGF MNKIFHPNID EASGTVKLD VINQTWTALY DLTNIFESFL PQLLAYPNPI DPLNGDAAAM YLHRPEEYKQ KIKEYIQKYA TEEALKEQEE G TGD(SEP)(SEP)(SEP)E(SEP) (SEP)M(SEP)DF(SEP)EDEA QDMEL UniProtKB: Ubiquitin-conjugating enzyme E2 H |
-分子 #3: E3 ubiquitin-protein transferase MAEA
| 分子 | 名称: E3 ubiquitin-protein transferase MAEA / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO / EC番号: RING-type E3 ubiquitin transferase |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 45.356332 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MAVQESAAQL SMTLKVQEYP TLKVPYETLN KRFRAAQKNI DRETSHVTMV VAELEKTLSG CPAVDSVVSL LDGVVEKLSV LKRKAVESI QAEDESAKLC KRRIEHLKEH SSDQPAAASV WKRKRMDRMM VEHLLRCGYY NTAVKLARQS GIEDLVNIEM F LTAKEVEE ...文字列: MAVQESAAQL SMTLKVQEYP TLKVPYETLN KRFRAAQKNI DRETSHVTMV VAELEKTLSG CPAVDSVVSL LDGVVEKLSV LKRKAVESI QAEDESAKLC KRRIEHLKEH SSDQPAAASV WKRKRMDRMM VEHLLRCGYY NTAVKLARQS GIEDLVNIEM F LTAKEVEE SLERRETATC LAWCHDNKSR LRKMKSCLEF SLRIQEFIEL IRQNKRLDAV RHARKHFSQA EGSQLDEVRQ AM GMLAFPP DTHISPYKDL LDPARWRMLI QQFRYDNYRL HQLGNNSVFT LTLQAGLSAI KTPQCYKEDG SSKSPDCPVC SRS LNKLAQ PLPMAHCANS RLVCKISGDV MNENNPPMML PNGYVYGYNS LLSIRQDDKV VCPRTKEVFH FSQAEKVYIM UniProtKB: E3 ubiquitin-protein transferase MAEA |
-分子 #4: Ubiquitin
| 分子 | 名称: Ubiquitin / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 8.576831 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MQIFVKTLTG KTITLEVEPS DTIENVKAKI QDKEGIPPDQ QRLIFAGKQL EDGRTLSDYN IQKESTLHLV LRLRGG UniProtKB: Polyubiquitin-C |
-分子 #5: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 5 / コピー数: 2 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 68.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD 最大 デフォーカス(公称値): 2.3000000000000003 µm 最小 デフォーカス(公称値): 0.8 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー






























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)