+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | In situ cryoEM structure of the Prototype Foamy Virus capsid, hexamer 2 localised reconstruction | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | capsid / Gag / foamy virus / hexamer / VIRAL PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報host cytoskeleton / microtubule-dependent intracellular transport of viral material towards nucleus / viral release from host cell / host cell / viral nucleocapsid / host cell cytoplasm / symbiont entry into host cell / host cell nucleus 類似検索 - 分子機能 | |||||||||
| 生物種 |  Eastern chimpanzee simian foamy virus (ウイルス) Eastern chimpanzee simian foamy virus (ウイルス) | |||||||||
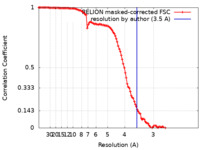
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.5 Å | |||||||||
 データ登録者 データ登録者 | Calcraft T / Nans A / Rosenthal PB | |||||||||
| 資金援助 |  英国, 1件 英国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2024 ジャーナル: Cell / 年: 2024タイトル: Integrated cryoEM structure of a spumaretrovirus reveals cross-kingdom evolutionary relationships and the molecular basis for assembly and virus entry. 著者: Thomas Calcraft / Nicole Stanke-Scheffler / Andrea Nans / Dirk Lindemann / Ian A Taylor / Peter B Rosenthal /   要旨: Foamy viruses (FVs) are an ancient lineage of retroviruses, with an evolutionary history spanning over 450 million years. Vector systems based on Prototype Foamy Virus (PFV) are promising candidates ...Foamy viruses (FVs) are an ancient lineage of retroviruses, with an evolutionary history spanning over 450 million years. Vector systems based on Prototype Foamy Virus (PFV) are promising candidates for gene and oncolytic therapies. Structural studies of PFV contribute to the understanding of the mechanisms of FV replication, cell entry and infection, and retroviral evolution. Here we combine cryoEM and cryoET to determine high-resolution in situ structures of the PFV icosahedral capsid (CA) and envelope glycoprotein (Env), including its type III transmembrane anchor and membrane-proximal external region (MPER), and show how they are organized in an integrated structure of assembled PFV particles. The atomic models reveal an ancient retroviral capsid architecture and an unexpected relationship between Env and other class 1 fusion proteins of the Mononegavirales. Our results represent the de novo structure determination of an assembled retrovirus particle. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_17315.map.gz emd_17315.map.gz | 116.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-17315-v30.xml emd-17315-v30.xml emd-17315.xml emd-17315.xml | 18.7 KB 18.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_17315_fsc.xml emd_17315_fsc.xml | 11.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_17315.png emd_17315.png | 107.4 KB | ||
| マスクデータ |  emd_17315_msk_1.map emd_17315_msk_1.map | 125 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-17315.cif.gz emd-17315.cif.gz | 6.1 KB | ||
| その他 |  emd_17315_half_map_1.map.gz emd_17315_half_map_1.map.gz emd_17315_half_map_2.map.gz emd_17315_half_map_2.map.gz | 97.6 MB 97.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-17315 http://ftp.pdbj.org/pub/emdb/structures/EMD-17315 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17315 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17315 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_17315_validation.pdf.gz emd_17315_validation.pdf.gz | 983.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_17315_full_validation.pdf.gz emd_17315_full_validation.pdf.gz | 982.9 KB | 表示 | |
| XML形式データ |  emd_17315_validation.xml.gz emd_17315_validation.xml.gz | 18.7 KB | 表示 | |
| CIF形式データ |  emd_17315_validation.cif.gz emd_17315_validation.cif.gz | 24.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17315 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17315 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17315 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17315 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8oznMC  8ozhC  8ozjC  8ozkC  8ozlC  8ozmC  8ozpC  8ozqC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_17315.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_17315.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.08 Å | ||||||||||||||||||||||||||||||||||||
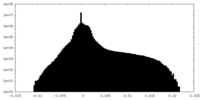
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_17315_msk_1.map emd_17315_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_17315_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_17315_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Eastern chimpanzee simian foamy virus
| 全体 | 名称:  Eastern chimpanzee simian foamy virus (ウイルス) Eastern chimpanzee simian foamy virus (ウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Eastern chimpanzee simian foamy virus
| 超分子 | 名称: Eastern chimpanzee simian foamy virus / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all / NCBI-ID: 2170195 / 生物種: Eastern chimpanzee simian foamy virus / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: Yes / ウイルス・中空状態: No |
|---|
-分子 #1: Gag polyprotein
| 分子 | 名称: Gag polyprotein / タイプ: protein_or_peptide / ID: 1 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Eastern chimpanzee simian foamy virus (ウイルス) Eastern chimpanzee simian foamy virus (ウイルス) |
| 分子量 | 理論値: 70.693414 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MASGSNVEEY ELDVEALVVI LRDRNIPRNP LHGEVIGLRL TEGWWGQIER FQMVRLILQN DDNEPLQRPR YEVIQRAVNP HTMFMISGP LAELQLAFQD LDLPEGPLRF GPLANGHYVQ GDPYSSSYRP VTMAETAQMT RDELEDVLNT QSEIEIQMIN L LELYEVET ...文字列: MASGSNVEEY ELDVEALVVI LRDRNIPRNP LHGEVIGLRL TEGWWGQIER FQMVRLILQN DDNEPLQRPR YEVIQRAVNP HTMFMISGP LAELQLAFQD LDLPEGPLRF GPLANGHYVQ GDPYSSSYRP VTMAETAQMT RDELEDVLNT QSEIEIQMIN L LELYEVET RALRRQLAER SSTGQGGISP GAPRSRPPVS SFSGLPSLPS IPGIHPRAPS PPRATSTPGN IPWSLGDDNP PS SSFPGPS QPRVSFHPGN PFVEEEGHRP RSQSRERRRE ILPAPVPSAP PMIQYIPVPP PPPIGTVIPI QHIRSVTGEP PRN PREIPI WLGRNAPAID GVFPVTTPDL RCRIINAILG GNIGLSLTPG DCLTWDSAVA TLFIRTHGTF PMHQLGNVIK GIVD QEGVA TAYTLGMMLS GQNYQLVSGI IRGYLPGQAV VTALQQRLDQ EIDDQTRAET FIQHLNAVYE ILGLNARGQS IRASV TPQP RPSRGRGRGQ NTSRPSQGPA NSGRGRQRPA SGQSNRGSST QNQNQDNLNQ GGYNLRPRTY QPQRYGGGRG RRWNDN TNN QESRPSDQGS QTPRPNQAGS GVRGNQSQTP RPAAGRGGRG NHNRNQRSSG AGDSRAVNTV TQSATSSTDE SSSAVTA AS GGDQRD UniProtKB: Gag polyprotein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY |
| 凍結 | 凍結剤: ETHANE / 装置: FEI VITROBOT MARK III |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum LS / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 3838 pixel / デジタル化 - サイズ - 縦: 3710 pixel / 平均電子線量: 48.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 4.0 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: RECIPROCAL / プロトコル: FLEXIBLE FIT |
|---|---|
| 得られたモデル |  PDB-8ozn: |
 ムービー
ムービー コントローラー
コントローラー















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)