+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
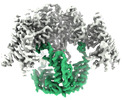
| タイトル | Core-binding domain of fungal E3-binding domain bound to the native pyruvate dehydrogenase E2 core | |||||||||
 マップデータ マップデータ | N. crassa native PDC core with E3BP interior trimer | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Complex / metabolism / mitochondria / TRANSFERASE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報dihydrolipoyllysine-residue acetyltransferase / dihydrolipoyllysine-residue acetyltransferase activity / acetyl-CoA biosynthetic process from pyruvate / : 類似検索 - 分子機能 | |||||||||
| 生物種 |  Neurospora crassa (菌類) Neurospora crassa (菌類) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.1 Å | |||||||||
 データ登録者 データ登録者 | Forsberg BO | |||||||||
| 資金援助 |  スウェーデン, 1件 スウェーデン, 1件
| |||||||||
 引用 引用 |  ジャーナル: Commun Biol / 年: 2023 ジャーナル: Commun Biol / 年: 2023タイトル: The structure and evolutionary diversity of the fungal E3-binding protein. 著者: Bjoern O Forsberg /   要旨: The pyruvate dehydrogenase complex (PDC) is a central metabolic enzyme in all living cells composed majorly of E1, E2, and E3. Tight coupling of their reactions makes each component essential, so ...The pyruvate dehydrogenase complex (PDC) is a central metabolic enzyme in all living cells composed majorly of E1, E2, and E3. Tight coupling of their reactions makes each component essential, so that any loss impacts oxidative metabolism pathologically. E3 retention is mediated by the E3-binding protein (E3BP), which is here resolved within the PDC core from N.crassa, resolved to 3.2Å. Fungal and mammalian E3BP are shown to be orthologs, arguing E3BP as a broadly eukaryotic gene. Fungal E3BP architectures predicted from sequence data and computational models further bridge the evolutionary distance between N.crassa and humans, and suggest discriminants for E3-specificity. This is confirmed by similarities in their respective E3-binding domains, where an interaction previously not described is also predicted. This provides evolutionary parallels for a crucial interaction human metabolism, an interaction specific to fungi that can be targeted, and an example of protein evolution following gene neofunctionalization. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_16884.map.gz emd_16884.map.gz | 19.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-16884-v30.xml emd-16884-v30.xml emd-16884.xml emd-16884.xml | 15.4 KB 15.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_16884.png emd_16884.png | 183.6 KB | ||
| Filedesc metadata |  emd-16884.cif.gz emd-16884.cif.gz | 5.6 KB | ||
| その他 |  emd_16884_half_map_1.map.gz emd_16884_half_map_1.map.gz emd_16884_half_map_2.map.gz emd_16884_half_map_2.map.gz | 96.7 MB 96.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-16884 http://ftp.pdbj.org/pub/emdb/structures/EMD-16884 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16884 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16884 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_16884_validation.pdf.gz emd_16884_validation.pdf.gz | 763.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_16884_full_validation.pdf.gz emd_16884_full_validation.pdf.gz | 763.3 KB | 表示 | |
| XML形式データ |  emd_16884_validation.xml.gz emd_16884_validation.xml.gz | 12 KB | 表示 | |
| CIF形式データ |  emd_16884_validation.cif.gz emd_16884_validation.cif.gz | 14.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16884 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16884 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16884 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16884 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8ohsMC  7r5mC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_16884.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_16884.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | N. crassa native PDC core with E3BP interior trimer | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.25 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #1
| ファイル | emd_16884_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
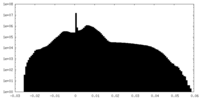
| 投影像・断面図 |
| ||||||||||||
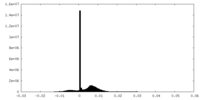
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_16884_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
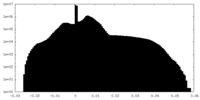
| 投影像・断面図 |
| ||||||||||||
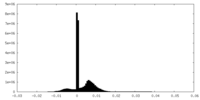
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Native pyruvate dehydrogenase complex
| 全体 | 名称: Native pyruvate dehydrogenase complex |
|---|---|
| 要素 |
|
-超分子 #1: Native pyruvate dehydrogenase complex
| 超分子 | 名称: Native pyruvate dehydrogenase complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Neurospora crassa (菌類) Neurospora crassa (菌類) |
-分子 #1: Dihydrolipoyllysine-residue acetyltransferase component of pyruva...
| 分子 | 名称: Dihydrolipoyllysine-residue acetyltransferase component of pyruvate dehydrogenase complex, mitochondrial タイプ: protein_or_peptide / ID: 1 / コピー数: 6 / 光学異性体: LEVO / EC番号: dihydrolipoyllysine-residue acetyltransferase |
|---|---|
| 由来(天然) | 生物種:  Neurospora crassa (菌類) Neurospora crassa (菌類) |
| 分子量 | 理論値: 48.677395 KDa |
| 配列 | 文字列: MIVPVLSRQA LRHASVARVA LPSLTRWYAS YPPHTVVKMP ALSPTMTSGG IGAWQKKPGD KIEPGEVLVE IETDKAQMDF EFQEEGVLA KILKDSGEKD VAVGNPIAIL VEEGTDVNAF KDFTLKDAGG ETSPAVPKDE PKNESTASAP TPAPTPAPEP E NTSFTGRF ...文字列: MIVPVLSRQA LRHASVARVA LPSLTRWYAS YPPHTVVKMP ALSPTMTSGG IGAWQKKPGD KIEPGEVLVE IETDKAQMDF EFQEEGVLA KILKDSGEKD VAVGNPIAIL VEEGTDVNAF KDFTLKDAGG ETSPAVPKDE PKNESTASAP TPAPTPAPEP E NTSFTGRF QTALEREPNA LPAAKRLARE KGIDLRNVKG SGPGGKITEE DVKKALASAP AAGAAAAAYT DVPISGMRKT IA ARLKESV TENPHFFVST NLSVSKLLKL RQALNSSADG RYKLSVNDFL IKAMGIASKR VPTVNSSWRD GVIRQFETVD VSV AVATPN GLITPIVKGV EGKGLESISA AVKELAKKAR DGKLKPEEYQ GGSISISNMG MNPAVQSFTA IINPPQAAIL AVGA PQKVA VPVENEDGTT GVSWDEQIIV TASFDHKVVD GAVGAEWIRE LKKVIENPLE LLL UniProtKB: Dihydrolipoyllysine-residue acetyltransferase component of pyruvate dehydrogenase complex, mitochondrial |
-分子 #2: Pyruvate dehydrogenase X component
| 分子 | 名称: Pyruvate dehydrogenase X component / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Neurospora crassa (菌類) Neurospora crassa (菌類) |
| 分子量 | 理論値: 44.880066 KDa |
| 配列 | 文字列: MASLTAACRI SARMAGRSVR GFRTSAAALA AQNFTMPALS PTMTEGNIAT WRVKEGDKFS AGDVLLEIET DKATMDVEAQ DDGVMVKIM KNDGAKGVAV GARIAVIAEE GDDISSLEIP ADAAPQSKPA ESAPSAPPPP TTADQSNVAV PESAPQNASS K SAPKPPKR ...文字列: MASLTAACRI SARMAGRSVR GFRTSAAALA AQNFTMPALS PTMTEGNIAT WRVKEGDKFS AGDVLLEIET DKATMDVEAQ DDGVMVKIM KNDGAKGVAV GARIAVIAEE GDDISSLEIP ADAAPQSKPA ESAPSAPPPP TTADQSNVAV PESAPQNASS K SAPKPPKR QYPHYPSVAH LLKVNGIDAA AVKDITPTGP GGRLLKGDVL AYLGKINAQT PSTVSERFEK QSHLDLSNIK VA KSTEAVK ATTEKAQSKK LDAPAPPPVA VVTAPISLSA AIDVQNKLHK TIGVFLPLST FITRATEIAN QKLPLPANYQ PTA DELFNQ VLGLDKVTRK ESRGSYTPTF GSFVAPQRAA RKADIIDILA APSTRVAASA QSKSAAPGLT TSGPNVFSLQ VPKS EEKRA QAFLQKMKLV LEQEPDKLVR A UniProtKB: Pyruvate dehydrogenase X component |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 平均電子線量: 35.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 4.0 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: EMDB MAP EMDB ID: |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 4.1 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 604402 |
| 初期 角度割当 | タイプ: RANDOM ASSIGNMENT |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
-原子モデル構築 1
| 初期モデル |
| ||||||
|---|---|---|---|---|---|---|---|
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT | ||||||
| 得られたモデル |  PDB-8ohs: |
 ムービー
ムービー コントローラー
コントローラー






 Z
Z Y
Y X
X