+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of yeast Elp456 subcomplex | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | wobble uridine modification / TRANSLATION | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報elongator holoenzyme complex / protein urmylation / tRNA wobble uridine modification / tRNA modification / transcription elongation factor complex / regulation of translation / tRNA binding / regulation of transcription by RNA polymerase II / nucleoplasm / identical protein binding ...elongator holoenzyme complex / protein urmylation / tRNA wobble uridine modification / tRNA modification / transcription elongation factor complex / regulation of translation / tRNA binding / regulation of transcription by RNA polymerase II / nucleoplasm / identical protein binding / nucleus / cytoplasm / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
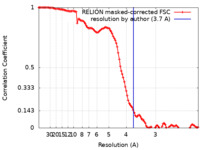
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.7 Å | |||||||||
 データ登録者 データ登録者 | Jaciuk M / Scherf D / Kaszuba K / Gaik M / Koscielniak A / Krutyholowa R / Rawski M / Indyka P / Biela A / Dobosz D ...Jaciuk M / Scherf D / Kaszuba K / Gaik M / Koscielniak A / Krutyholowa R / Rawski M / Indyka P / Biela A / Dobosz D / Lin T-Y / Abbassi N / Hammermeister A / Chramiec-Glabik A / Kosinski J / Schaffrath R / Glatt S | |||||||||
| 資金援助 |  ポーランド, European Union, 2件 ポーランド, European Union, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nucleic Acids Res / 年: 2023 ジャーナル: Nucleic Acids Res / 年: 2023タイトル: Cryo-EM structure of the fully assembled Elongator complex. 著者: Marcin Jaciuk / David Scherf / Karol Kaszuba / Monika Gaik / Alexander Rau / Anna Kościelniak / Rościsław Krutyhołowa / Michał Rawski / Paulina Indyka / Andrea Graziadei / Andrzej ...著者: Marcin Jaciuk / David Scherf / Karol Kaszuba / Monika Gaik / Alexander Rau / Anna Kościelniak / Rościsław Krutyhołowa / Michał Rawski / Paulina Indyka / Andrea Graziadei / Andrzej Chramiec-Głąbik / Anna Biela / Dominika Dobosz / Ting-Yu Lin / Nour-El-Hana Abbassi / Alexander Hammermeister / Juri Rappsilber / Jan Kosinski / Raffael Schaffrath / Sebastian Glatt /    要旨: Transfer RNA (tRNA) molecules are essential to decode messenger RNA codons during protein synthesis. All known tRNAs are heavily modified at multiple positions through post-transcriptional addition ...Transfer RNA (tRNA) molecules are essential to decode messenger RNA codons during protein synthesis. All known tRNAs are heavily modified at multiple positions through post-transcriptional addition of chemical groups. Modifications in the tRNA anticodons are directly influencing ribosome decoding and dynamics during translation elongation and are crucial for maintaining proteome integrity. In eukaryotes, wobble uridines are modified by Elongator, a large and highly conserved macromolecular complex. Elongator consists of two subcomplexes, namely Elp123 containing the enzymatically active Elp3 subunit and the associated Elp456 hetero-hexamer. The structure of the fully assembled complex and the function of the Elp456 subcomplex have remained elusive. Here, we show the cryo-electron microscopy structure of yeast Elongator at an overall resolution of 4.3 Å. We validate the obtained structure by complementary mutational analyses in vitro and in vivo. In addition, we determined various structures of the murine Elongator complex, including the fully assembled mouse Elongator complex at 5.9 Å resolution. Our results confirm the structural conservation of Elongator and its intermediates among eukaryotes. Furthermore, we complement our analyses with the biochemical characterization of the assembled human Elongator. Our results provide the molecular basis for the assembly of Elongator and its tRNA modification activity in eukaryotes. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_15635.map.gz emd_15635.map.gz | 7.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-15635-v30.xml emd-15635-v30.xml emd-15635.xml emd-15635.xml | 23.7 KB 23.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_15635_fsc.xml emd_15635_fsc.xml | 11.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_15635.png emd_15635.png | 135.3 KB | ||
| Filedesc metadata |  emd-15635.cif.gz emd-15635.cif.gz | 7.3 KB | ||
| その他 |  emd_15635_half_map_1.map.gz emd_15635_half_map_1.map.gz emd_15635_half_map_2.map.gz emd_15635_half_map_2.map.gz | 104.7 MB 104.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-15635 http://ftp.pdbj.org/pub/emdb/structures/EMD-15635 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15635 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15635 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_15635_validation.pdf.gz emd_15635_validation.pdf.gz | 630.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_15635_full_validation.pdf.gz emd_15635_full_validation.pdf.gz | 630.3 KB | 表示 | |
| XML形式データ |  emd_15635_validation.xml.gz emd_15635_validation.xml.gz | 18.6 KB | 表示 | |
| CIF形式データ |  emd_15635_validation.cif.gz emd_15635_validation.cif.gz | 24.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15635 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15635 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15635 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15635 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_15635.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_15635.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.1 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #1
| ファイル | emd_15635_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
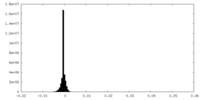
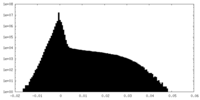
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_15635_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
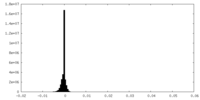
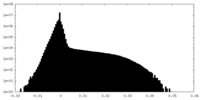
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Yeast Elp456 subcomplex
| 全体 | 名称: Yeast Elp456 subcomplex |
|---|---|
| 要素 |
|
-超分子 #1: Yeast Elp456 subcomplex
| 超分子 | 名称: Yeast Elp456 subcomplex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 233.8 KDa |
-分子 #1: Elongator complex protein 4
| 分子 | 名称: Elongator complex protein 4 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 51.232469 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSFRKRGEIL NDRGSGLRGP LLRGPPRTSS TPLRTGNRRA PGNVPLSDTT ARLKKLNIAD ESKTKMGLDS SHVGVRPSPA TSQPTTSTG SADLDSILGH MGLPLGNSVL VEEQSTTEFH SILGKLFAAQ GIVHNRISDS SADKTRNGDT HVIVLSLNQM F AKELPGIY ...文字列: MSFRKRGEIL NDRGSGLRGP LLRGPPRTSS TPLRTGNRRA PGNVPLSDTT ARLKKLNIAD ESKTKMGLDS SHVGVRPSPA TSQPTTSTG SADLDSILGH MGLPLGNSVL VEEQSTTEFH SILGKLFAAQ GIVHNRISDS SADKTRNGDT HVIVLSLNQM F AKELPGIY KGSRKQMKKN LISEEESKVT VQNLNETQRS TPSRYKDLKI AWKYKLADEK RLGSPDRDDI QQNSEYKDYN HQ FDITTRL MPAPIASELT FIAPTQPVST ILSQIEQTIK RNDKKLIRIV IPSLLHPAMY PPKMFESSEI IGLMHGVRSL VKK YYERVV LFASISIDII TPPLLVLLRN MFDSVINLEP FNQEMTEFLE RVYKSQPGKI QHGLVHILKL PVFTDRGEMR VLKS EWAFK NGRKKFEIEQ WGIPVDDAEG SAASEQSHSH SHSDEISHNI PAKKTKISLD Y UniProtKB: Elongator complex protein 4 |
-分子 #2: Elongator complex protein 5
| 分子 | 名称: Elongator complex protein 5 / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 35.252496 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MASSSHNPVI LLKRILSLTE SSPFILCLDS IAQTSYKLIQ EFVHQSKSKG NEYPIVYISF ETVNKPSYCT QFIDATQMDF VHLVKQIIS YLPAATATQA KKHMVIIDSL NYISTEYITR FLSEIASPHC TMVATYHKDI KDENRTVIPD WNNNYPDKLT L LQFMATTI ...文字列: MASSSHNPVI LLKRILSLTE SSPFILCLDS IAQTSYKLIQ EFVHQSKSKG NEYPIVYISF ETVNKPSYCT QFIDATQMDF VHLVKQIIS YLPAATATQA KKHMVIIDSL NYISTEYITR FLSEIASPHC TMVATYHKDI KDENRTVIPD WNNNYPDKLT L LQFMATTI VDIDVVLTGT LDTEEVSELL NEFRIPRGLN NDIFQLRLVN KRKSGRSLEY DFIVNSNTHE YELLSTTKQE EE SSSNGLE TPEMLQGLTT FNLGTSNKQK LAKDQVALPF LEAQSFGQGG AIVYEYEKDD DYDEEDPYED PF UniProtKB: Elongator complex protein 5 |
-分子 #3: Elongator complex protein 6
| 分子 | 名称: Elongator complex protein 6 / タイプ: protein_or_peptide / ID: 3 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 30.602611 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGSVQRQDLV LFSDQSVLPA HFFQDSNSHN LFFITHQSCT QPLWMINALV ETHVLGSPSS LNESSSSMLP SSTRSHAVLA SFIHEQNYF TNSLNKLKIP SNNYNVLDFL SDFIVNNIHN KPRDKILSDV LAKFSAAIQN NPTDTIVIIE QPELLLSLVS G LTCSELNN ...文字列: MGSVQRQDLV LFSDQSVLPA HFFQDSNSHN LFFITHQSCT QPLWMINALV ETHVLGSPSS LNESSSSMLP SSTRSHAVLA SFIHEQNYF TNSLNKLKIP SNNYNVLDFL SDFIVNNIHN KPRDKILSDV LAKFSAAIQN NPTDTIVIIE QPELLLSLVS G LTCSELNN KFITPLLRQC KVLIIVSNSD IFNIDEYDAS VHSSNLQNFY KSSFIKSMIN LNLNPLKTGF AKDVTGSLHV CR GGAPIAT SNTSLHVVEN EYLYLNEKES TKLFYR UniProtKB: Elongator complex protein 6 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.4 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 200 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. / 前処理 - 雰囲気: AIR | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV / 詳細: 15 s wait time, blot force 5, 5 s blot time. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) デジタル化 - サイズ - 横: 5760 pixel / デジタル化 - サイズ - 縦: 4092 pixel / 実像数: 4716 / 平均露光時間: 1.82 sec. / 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 0.9 µm / 倍率(公称値): 81000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー













 Z
Z Y
Y X
X