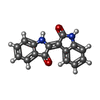
登録情報 データベース : EMDB / ID : EMD-14971タイトル Cryo-EM structure of the indirubin-bound Hsp90-XAP2-AHR complex 複合体 : Hsp90-XAP2-AHR complexタンパク質・ペプチド : Heat shock protein HSP 90-betaタンパク質・ペプチド : AH receptor-interacting proteinタンパク質・ペプチド : Aryl hydrocarbon receptorリガンド : ADENOSINE-5'-DIPHOSPHATEリガンド : MAGNESIUM IONリガンド : MOLYBDATE IONリガンド : (3~{Z})-3-(3-oxidanylidene-1~{H}-indol-2-ylidene)-1~{H}-indol-2-one機能・相同性 分子機能 ドメイン・相同性 構成要素
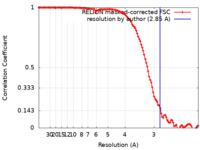
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Homo sapiens (ヒト)手法 / / 解像度 : 2.85 Å Gruszczyk J / Savva CG / Lai-Kee-Him J / Bous J / Ancelin A / Kwong HS / Grandvuillemin L / Bourguet W 資金援助 European Union, Organization Grant number 国 European Union (EU) 825489 European Union ATIP-Avenir R20059SP Montpellier University of Excellence (MUSE) ANR-16-IDEX-0006
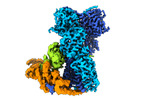
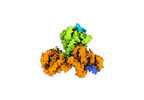
ジャーナル : Nat Commun / 年 : 2022タイトル : Cryo-EM structure of the agonist-bound Hsp90-XAP2-AHR cytosolic complex.著者: Jakub Gruszczyk / Loïc Grandvuillemin / Josephine Lai-Kee-Him / Matteo Paloni / Christos G Savva / Pierre Germain / Marina Grimaldi / Abdelhay Boulahtouf / Hok-Sau Kwong / Julien Bous / ... 著者 : Jakub Gruszczyk / Loïc Grandvuillemin / Josephine Lai-Kee-Him / Matteo Paloni / Christos G Savva / Pierre Germain / Marina Grimaldi / Abdelhay Boulahtouf / Hok-Sau Kwong / Julien Bous / Aurélie Ancelin / Cherine Bechara / Alessandro Barducci / Patrick Balaguer / William Bourguet / 要旨 : The aryl hydrocarbon receptor (AHR) is a ligand-dependent transcription factor that mediates a broad spectrum of (patho)physiological processes in response to numerous substances including ... The aryl hydrocarbon receptor (AHR) is a ligand-dependent transcription factor that mediates a broad spectrum of (patho)physiological processes in response to numerous substances including pollutants, natural products and metabolites. However, the scarcity of structural data precludes understanding of how AHR is activated by such diverse compounds. Our 2.85 Å structure of the human indirubin-bound AHR complex with the chaperone Hsp90 and the co-chaperone XAP2, reported herein, reveals a closed conformation Hsp90 dimer with AHR threaded through its lumen and XAP2 serving as a brace. Importantly, we disclose the long-awaited structure of the AHR PAS-B domain revealing a unique organisation of the ligand-binding pocket and the structural determinants of ligand-binding specificity and promiscuity of the receptor. By providing structural details of the molecular initiating event leading to AHR activation, our study rationalises almost forty years of biochemical data and provides a framework for future mechanistic studies and structure-guided drug design. 履歴 登録 2022年5月12日 - ヘッダ(付随情報) 公開 2022年11月23日 - マップ公開 2022年11月23日 - 更新 2022年12月7日 - 現状 2022年12月7日 処理サイト : PDBe / 状態 : 公開
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報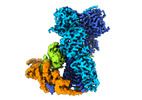
 マップデータ
マップデータ 試料
試料 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) データ登録者
データ登録者 フランス, 3件
フランス, 3件  引用
引用 ジャーナル: Nat Commun / 年: 2022
ジャーナル: Nat Commun / 年: 2022


 構造の表示
構造の表示 ダウンロードとリンク
ダウンロードとリンク emd_14971.map.gz
emd_14971.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-14971-v30.xml
emd-14971-v30.xml emd-14971.xml
emd-14971.xml EMDBヘッダ
EMDBヘッダ emd_14971_fsc.xml
emd_14971_fsc.xml FSCデータファイル
FSCデータファイル emd_14971.png
emd_14971.png emd_14971_msk_1.map
emd_14971_msk_1.map マスクマップ
マスクマップ emd_14971_half_map_1.map.gz
emd_14971_half_map_1.map.gz emd_14971_half_map_2.map.gz
emd_14971_half_map_2.map.gz http://ftp.pdbj.org/pub/emdb/structures/EMD-14971
http://ftp.pdbj.org/pub/emdb/structures/EMD-14971 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14971
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14971 emd_14971_validation.pdf.gz
emd_14971_validation.pdf.gz EMDB検証レポート
EMDB検証レポート emd_14971_full_validation.pdf.gz
emd_14971_full_validation.pdf.gz emd_14971_validation.xml.gz
emd_14971_validation.xml.gz emd_14971_validation.cif.gz
emd_14971_validation.cif.gz https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14971
https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14971 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14971
ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14971
 F&H 検索
F&H 検索 リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
マップ ダウンロード / ファイル: emd_14971.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_14971.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) emd_14971_msk_1.map
emd_14971_msk_1.map 試料の構成要素
試料の構成要素 Homo sapiens (ヒト)
Homo sapiens (ヒト) Homo sapiens (ヒト)
Homo sapiens (ヒト)
 Homo sapiens (ヒト)
Homo sapiens (ヒト)
 Homo sapiens (ヒト)
Homo sapiens (ヒト)

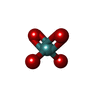
 解析
解析 試料調製
試料調製 電子顕微鏡法
電子顕微鏡法 FIELD EMISSION GUN
FIELD EMISSION GUN
 ムービー
ムービー コントローラー
コントローラー































 Z
Z Y
Y X
X