+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | PucA-LH2 complex from Rps. palustris | |||||||||
 マップデータ マップデータ | LH2 from Rps. palustris. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | light harvesting complex 2 / LH2 / Rps. palustris / photosynthesis / purple bacteria / cryo-EM / single particle analysis | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報organelle inner membrane / plasma membrane light-harvesting complex / bacteriochlorophyll binding / photosynthesis, light reaction / electron transporter, transferring electrons within the cyclic electron transport pathway of photosynthesis activity / membrane => GO:0016020 / metal ion binding / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Rhodopseudomonas palustris ATCC 17001 (光合成細菌) Rhodopseudomonas palustris ATCC 17001 (光合成細菌) | |||||||||
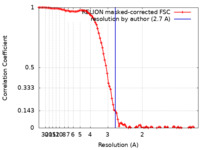
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.7 Å | |||||||||
 データ登録者 データ登録者 | Qian P / Cogdell RJ | |||||||||
| 資金援助 |  英国, 1件 英国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2022 ジャーナル: Proc Natl Acad Sci U S A / 年: 2022タイトル: Cryo-EM structures of light-harvesting 2 complexes from reveal the molecular origin of absorption tuning. 著者: Pu Qian / Cam T Nguyen-Phan / Alastair T Gardiner / Tristan I Croll / Aleksander W Roszak / June Southall / Philip J Jackson / Cvetelin Vasilev / Pablo Castro-Hartmann / Kasim Sader / C Neil ...著者: Pu Qian / Cam T Nguyen-Phan / Alastair T Gardiner / Tristan I Croll / Aleksander W Roszak / June Southall / Philip J Jackson / Cvetelin Vasilev / Pablo Castro-Hartmann / Kasim Sader / C Neil Hunter / Richard J Cogdell /   要旨: The genomes of some purple photosynthetic bacteria contain a multigene family encoding a series of α- and β-polypeptides that together form a heterogeneous antenna of light-harvesting 2 (LH2) ...The genomes of some purple photosynthetic bacteria contain a multigene family encoding a series of α- and β-polypeptides that together form a heterogeneous antenna of light-harvesting 2 (LH2) complexes. To unravel this complexity, we generated four sets of deletion mutants in , each encoding a single type of gene pair and enabling the purification of complexes designated as PucA-LH2, PucB-LH2, PucD-LH2, and PucE-LH2. The structures of all four purified LH2 complexes were determined by cryogenic electron microscopy (cryo-EM) at resolutions ranging from 2.7 to 3.6 Å. Uniquely, each of these complexes contains a hitherto unknown polypeptide, γ, that forms an extended undulating ribbon that lies in the plane of the membrane and that encloses six of the nine LH2 αβ-subunits. The γ-subunit, which is located near to the cytoplasmic side of the complex, breaks the C9 symmetry of the LH2 complex and binds six extra bacteriochlorophylls (BChls) that enhance the 800-nm absorption of each complex. The structures show that all four complexes have two complete rings of BChls, conferring absorption bands centered at 800 and 850 nm on the PucA-LH2, PucB-LH2, and PucE-LH2 complexes, but, unusually, the PucD-LH2 antenna has only a single strong near-infared (NIR) absorption peak at 803 nm. Comparison of the cryo-EM structures of these LH2 complexes reveals altered patterns of hydrogen bonds between LH2 αβ-side chains and the bacteriochlorin rings, further emphasizing the major role that H bonds play in spectral tuning of bacterial antenna complexes. #1:  ジャーナル: Acta Crystallogr., Sect. D: Biol. Crystallogr. ジャーナル: Acta Crystallogr., Sect. D: Biol. Crystallogr.年: 2018 タイトル: Real-space refinement in PHENIX for cryo-EM and crystallography 著者: Qian P / Cogdell RJ #2:  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Cryo-EM structures of light harvesting complex 2 from Rhodopseudomonas palustris: a molecular origin of spectroscopic variation 著者: Qian P / Cogdell R | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_14633.map.gz emd_14633.map.gz | 9.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-14633-v30.xml emd-14633-v30.xml emd-14633.xml emd-14633.xml | 22.4 KB 22.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_14633_fsc.xml emd_14633_fsc.xml | 9.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_14633.png emd_14633.png | 81.3 KB | ||
| Filedesc metadata |  emd-14633.cif.gz emd-14633.cif.gz | 6.5 KB | ||
| その他 |  emd_14633_half_map_1.map.gz emd_14633_half_map_1.map.gz emd_14633_half_map_2.map.gz emd_14633_half_map_2.map.gz | 58.5 MB 58.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-14633 http://ftp.pdbj.org/pub/emdb/structures/EMD-14633 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14633 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14633 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_14633_validation.pdf.gz emd_14633_validation.pdf.gz | 853.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_14633_full_validation.pdf.gz emd_14633_full_validation.pdf.gz | 853.1 KB | 表示 | |
| XML形式データ |  emd_14633_validation.xml.gz emd_14633_validation.xml.gz | 16.7 KB | 表示 | |
| CIF形式データ |  emd_14633_validation.cif.gz emd_14633_validation.cif.gz | 21.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14633 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14633 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14633 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14633 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_14633.map.gz / 形式: CCP4 / 大きさ: 75.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_14633.map.gz / 形式: CCP4 / 大きさ: 75.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | LH2 from Rps. palustris. | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.65 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: The first half map, z-flipped.
| ファイル | emd_14633_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | The first half map, z-flipped. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: The second half map, z-flipped.
| ファイル | emd_14633_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | The second half map, z-flipped. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : PucA-LH2 from Rps. palustris
| 全体 | 名称: PucA-LH2 from Rps. palustris |
|---|---|
| 要素 |
|
-超分子 #1: PucA-LH2 from Rps. palustris
| 超分子 | 名称: PucA-LH2 from Rps. palustris / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 / 詳細: Delt-pucBA(bced) |
|---|---|
| 由来(天然) | 生物種:  Rhodopseudomonas palustris ATCC 17001 (光合成細菌) Rhodopseudomonas palustris ATCC 17001 (光合成細菌) |
-分子 #1: Light-harvesting protein B-800-850 alpha chain
| 分子 | 名称: Light-harvesting protein B-800-850 alpha chain / タイプ: protein_or_peptide / ID: 1 / コピー数: 9 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Rhodopseudomonas palustris ATCC 17001 (光合成細菌) Rhodopseudomonas palustris ATCC 17001 (光合成細菌)株: ATCC 17001 |
| 分子量 | 理論値: 6.443515 KDa |
| 配列 | 文字列: (CXM)NQARIWTVV KPTVGLPLLL GSVTVIAILV HFAVLSHTTW FSKYWNGKAA AIESSVNVG UniProtKB: Light-harvesting protein B-800-850 alpha chain |
-分子 #2: Light-harvesting protein B-800-850 beta chain
| 分子 | 名称: Light-harvesting protein B-800-850 beta chain / タイプ: protein_or_peptide / ID: 2 / コピー数: 9 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Rhodopseudomonas palustris ATCC 17001 (光合成細菌) Rhodopseudomonas palustris ATCC 17001 (光合成細菌)株: ATCC 17001 |
| 分子量 | 理論値: 5.288061 KDa |
| 配列 | 文字列: MADKTLTGLT VEESEELHKH VIDGTRIFGA IAIVAHFLAY VYSPWLH UniProtKB: Light-harvesting protein B-800-850 beta chain |
-分子 #3: PucA-LH2-gamma
| 分子 | 名称: PucA-LH2-gamma / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Rhodopseudomonas palustris ATCC 17001 (光合成細菌) Rhodopseudomonas palustris ATCC 17001 (光合成細菌)株: ATCC 17001 |
| 分子量 | 理論値: 10.848179 KDa |
| 配列 | 文字列: MSEEYKGHSG HPLILKQEGE YKGYSGEPLI LKQEGEYKGY SGTPLILEQK GEYQSFSGTP LILKQEGEYR GFSGAPLILK QDGEYKSFS GYPLLLNI UniProtKB: Uncharacterized protein |
-分子 #4: BACTERIOCHLOROPHYLL A
| 分子 | 名称: BACTERIOCHLOROPHYLL A / タイプ: ligand / ID: 4 / コピー数: 33 / 式: BCL |
|---|---|
| 分子量 | 理論値: 911.504 Da |
| Chemical component information |  ChemComp-BCL: |
-分子 #5: 1,2-Dihydro-psi,psi-caroten-1-ol
| 分子 | 名称: 1,2-Dihydro-psi,psi-caroten-1-ol / タイプ: ligand / ID: 5 / コピー数: 9 / 式: IRM |
|---|---|
| 分子量 | 理論値: 554.888 Da |
| Chemical component information |  ChemComp-IRM: |
-分子 #6: water
| 分子 | 名称: water / タイプ: ligand / ID: 6 / コピー数: 9 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 7.0 mg/mL |
|---|---|
| 緩衝液 | pH: 8 / 構成要素 - 濃度: 20.0 mM / 構成要素 - 式: C4H11NO3 / 構成要素 - 名称: Tris.HCl / 詳細: 0.1% LDAO in 20 mM Tris.Cl pH 8.0 |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV 詳細: Chamber humidity: 100%; Chamber temperature: 4 oC; Blotting time: 2.5; Blotting force: 3; Wait time: 30 sec. |
| 詳細 | protein was purified using LDAO detergent. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 温度 | 最低: 80.0 K |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 撮影したグリッド数: 1 / 実像数: 4865 / 平均露光時間: 1.4 sec. / 平均電子線量: 42.03 e/Å2 / 詳細: Images were collected in AFIS mode. |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.4 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 130000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)