+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
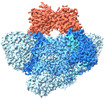
| タイトル | E. coli C-P lyase bound to a PhnK ABC dimer and ATP | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | protein complex / transferase / ABC / hydrolase / lyase / carbon phosphorus | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報alpha-D-ribose 1-methylphosphonate 5-phosphate C-P-lyase / alpha-D-ribose 1-methylphosphonate 5-phosphate C-P-lyase activity / alpha-D-ribose 1-methylphosphonate 5-triphosphate synthase / alpha-D-ribose 1-methylphosphonate 5-triphosphate synthase activity / alpha-D-ribose 1-methylphosphonate 5-triphosphate synthase complex / carbon phosphorus lyase complex / organic phosphonate metabolic process / organic phosphonate transport / organic phosphonate catabolic process / peptide transport ...alpha-D-ribose 1-methylphosphonate 5-phosphate C-P-lyase / alpha-D-ribose 1-methylphosphonate 5-phosphate C-P-lyase activity / alpha-D-ribose 1-methylphosphonate 5-triphosphate synthase / alpha-D-ribose 1-methylphosphonate 5-triphosphate synthase activity / alpha-D-ribose 1-methylphosphonate 5-triphosphate synthase complex / carbon phosphorus lyase complex / organic phosphonate metabolic process / organic phosphonate transport / organic phosphonate catabolic process / peptide transport / 4 iron, 4 sulfur cluster binding / lyase activity / protein homodimerization activity / ATP hydrolysis activity / ATP binding / identical protein binding / metal ion binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
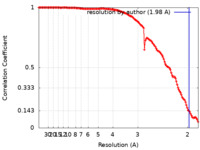
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 1.98 Å | |||||||||
 データ登録者 データ登録者 | Amstrup SK / Sofos N | |||||||||
| 資金援助 |  デンマーク, 1件 デンマーク, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: Structural remodelling of the carbon-phosphorus lyase machinery by a dual ABC ATPase. 著者: Søren K Amstrup / Sui Ching Ong / Nicholas Sofos / Jesper L Karlsen / Ragnhild B Skjerning / Thomas Boesen / Jan J Enghild / Bjarne Hove-Jensen / Ditlev E Brodersen /  要旨: In Escherichia coli, the 14-cistron phn operon encoding carbon-phosphorus lyase allows for utilisation of phosphorus from a wide range of stable phosphonate compounds containing a C-P bond. As part ...In Escherichia coli, the 14-cistron phn operon encoding carbon-phosphorus lyase allows for utilisation of phosphorus from a wide range of stable phosphonate compounds containing a C-P bond. As part of a complex, multi-step pathway, the PhnJ subunit was shown to cleave the C-P bond via a radical mechanism, however, the details of the reaction could not immediately be reconciled with the crystal structure of a 220 kDa PhnGHIJ C-P lyase core complex, leaving a significant gap in our understanding of phosphonate breakdown in bacteria. Here, we show using single-particle cryogenic electron microscopy that PhnJ mediates binding of a double dimer of the ATP-binding cassette proteins, PhnK and PhnL, to the core complex. ATP hydrolysis induces drastic structural remodelling leading to opening of the core complex and reconfiguration of a metal-binding and putative active site located at the interface between the PhnI and PhnJ subunits. #1:  ジャーナル: Biorxiv / 年: 2022 ジャーナル: Biorxiv / 年: 2022タイトル: Structural remodelling of the carbon-phosphorus lyase machinery by a dual ABC ATPase 著者: Amstrup SK / Sofos N / Karlsen JL / Skjerning RB / Boesen T / Enghild JJ / Hove-Jensen B / Brodersen DE | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_14444.map.gz emd_14444.map.gz | 110.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-14444-v30.xml emd-14444-v30.xml emd-14444.xml emd-14444.xml | 29.5 KB 29.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_14444_fsc.xml emd_14444_fsc.xml | 13.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_14444.png emd_14444.png | 207.1 KB | ||
| Filedesc metadata |  emd-14444.cif.gz emd-14444.cif.gz | 7.9 KB | ||
| その他 |  emd_14444_additional_1.map.gz emd_14444_additional_1.map.gz emd_14444_half_map_1.map.gz emd_14444_half_map_1.map.gz emd_14444_half_map_2.map.gz emd_14444_half_map_2.map.gz | 4.4 MB 203.4 MB 203.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-14444 http://ftp.pdbj.org/pub/emdb/structures/EMD-14444 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14444 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14444 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_14444_validation.pdf.gz emd_14444_validation.pdf.gz | 965.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_14444_full_validation.pdf.gz emd_14444_full_validation.pdf.gz | 964.9 KB | 表示 | |
| XML形式データ |  emd_14444_validation.xml.gz emd_14444_validation.xml.gz | 21.6 KB | 表示 | |
| CIF形式データ |  emd_14444_validation.cif.gz emd_14444_validation.cif.gz | 28.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14444 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14444 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14444 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14444 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_14444.map.gz / 形式: CCP4 / 大きさ: 219.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_14444.map.gz / 形式: CCP4 / 大きさ: 219.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.92189 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: Map filtered to local resolution
| ファイル | emd_14444_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Map filtered to local resolution | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
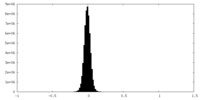
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_14444_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
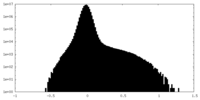
| 密度ヒストグラム |
-ハーフマップ: Half map A
| ファイル | emd_14444_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
+全体 : E. coli C-P lyase bound to a PhnK ABC dimer and ATP
+超分子 #1: E. coli C-P lyase bound to a PhnK ABC dimer and ATP
+分子 #1: Alpha-D-ribose 1-methylphosphonate 5-triphosphate synthase subuni...
+分子 #2: Alpha-D-ribose 1-methylphosphonate 5-triphosphate synthase subuni...
+分子 #3: Alpha-D-ribose 1-methylphosphonate 5-triphosphate synthase subuni...
+分子 #4: Alpha-D-ribose 1-methylphosphonate 5-phosphate C-P lyase
+分子 #5: Putative phosphonates utilization ATP-binding protein PhnK
+分子 #6: ZINC ION
+分子 #7: alpha-D-ribose-1,2-cyclic-phosphate-5-phosphate
+分子 #8: ADENOSINE-5'-TRIPHOSPHATE
+分子 #9: MAGNESIUM ION
+分子 #10: water
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2.5 mg/mL | |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
詳細: ATP up from a concentration from 0.25 to 1.5 and MgCl2 to a concentration of 6 mM was added 15 seconds before the sample was plunge frozen | |||||||||||||||||||||
| グリッド | モデル: UltrAuFoil R0.6/1 / 材質: GOLD / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 90 sec. / 前処理 - 雰囲気: AIR / 詳細: 10 mA for 90 seconds in a GloQube, Quorum | |||||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 310 K / 装置: LEICA EM GP / 詳細: Blotting for 6-9 seconds before plunging. | |||||||||||||||||||||
| 詳細 | Sample was produced under ATP to ADP+Pi turnover conditions. Sample was monodisperse. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 1 / 実像数: 6637 / 平均電子線量: 62.0 e/Å2 詳細: Images were collected in movie-mode with a total of 56 frames |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm 最大 デフォーカス(公称値): 1.4000000000000001 µm 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 135000 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー














 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)