+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | SARS-CoV-2 S Omicron Spike B.1.1.529 - RBD up - 1-P2G3 and 1-P5C3 Fabs (Local) | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 | Surface glycoprotein 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
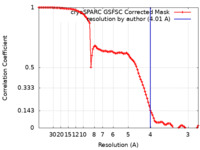
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.01 Å | |||||||||
 データ登録者 データ登録者 | Ni D / Lau K / Turelli P / Fenwick C / Perez L / Pojer F / Stahlberg H / Pantaleo G / Trono D | |||||||||
| 資金援助 |  スイス, 1件 スイス, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Microbiol / 年: 2022 ジャーナル: Nat Microbiol / 年: 2022タイトル: Patient-derived monoclonal antibody neutralizes SARS-CoV-2 Omicron variants and confers full protection in monkeys. 著者: Craig Fenwick / Priscilla Turelli / Dongchun Ni / Laurent Perez / Kelvin Lau / Cécile Herate / Romain Marlin / Erica Lana / Céline Pellaton / Charlène Raclot / Line Esteves-Leuenberger / ...著者: Craig Fenwick / Priscilla Turelli / Dongchun Ni / Laurent Perez / Kelvin Lau / Cécile Herate / Romain Marlin / Erica Lana / Céline Pellaton / Charlène Raclot / Line Esteves-Leuenberger / Jérémy Campos / Alex Farina / Flurin Fiscalini / Nathalie Dereuddre-Bosquet / Francis Relouzat / Rana Abdelnabi / Caroline S Foo / Johan Neyts / Pieter Leyssen / Yves Lévy / Florence Pojer / Henning Stahlberg / Roger LeGrand / Didier Trono / Giuseppe Pantaleo /    要旨: The SARS-CoV-2 Omicron variant has very high levels of transmission, is resistant to neutralization by authorized therapeutic human monoclonal antibodies (mAb) and is less sensitive to vaccine- ...The SARS-CoV-2 Omicron variant has very high levels of transmission, is resistant to neutralization by authorized therapeutic human monoclonal antibodies (mAb) and is less sensitive to vaccine-mediated immunity. To provide additional therapies against Omicron, we isolated a mAb named P2G3 from a previously infected vaccinated donor and showed that it has picomolar-range neutralizing activity against Omicron BA.1, BA.1.1, BA.2 and all other variants tested. We solved the structure of P2G3 Fab in complex with the Omicron spike using cryo-electron microscopy at 3.04 Å resolution to identify the P2G3 epitope as a Class 3 mAb that is different from mAb-binding spike epitopes reported previously. Using a SARS-CoV-2 Omicron monkey challenge model, we show that P2G3 alone, or in combination with P5C3 (a broadly active Class 1 mAb previously identified), confers complete prophylactic or therapeutic protection. Although we could select for SARS-CoV-2 mutants escaping neutralization by P2G3 or by P5C3 in vitro, they had low infectivity and 'escape' mutations are extremely rare in public sequence databases. We conclude that this combination of mAbs has potential as an anti-Omicron drug. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_14142.map.gz emd_14142.map.gz | 97.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-14142-v30.xml emd-14142-v30.xml emd-14142.xml emd-14142.xml | 17.8 KB 17.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_14142_fsc.xml emd_14142_fsc.xml | 9.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_14142.png emd_14142.png | 44.6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-14142 http://ftp.pdbj.org/pub/emdb/structures/EMD-14142 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14142 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14142 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_14142_validation.pdf.gz emd_14142_validation.pdf.gz | 496.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_14142_full_validation.pdf.gz emd_14142_full_validation.pdf.gz | 496.1 KB | 表示 | |
| XML形式データ |  emd_14142_validation.xml.gz emd_14142_validation.xml.gz | 11.2 KB | 表示 | |
| CIF形式データ |  emd_14142_validation.cif.gz emd_14142_validation.cif.gz | 15.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14142 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14142 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14142 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14142 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_14142.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_14142.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.3833 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Spike Omicron trimer with Fabs
| 全体 | 名称: Spike Omicron trimer with Fabs |
|---|---|
| 要素 |
|
-超分子 #1: Spike Omicron trimer with Fabs
| 超分子 | 名称: Spike Omicron trimer with Fabs / タイプ: complex / キメラ: Yes / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
-分子 #1: P2G3 Heavy Chain
| 分子 | 名称: P2G3 Heavy Chain / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 24.562475 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: EVQLVESGGG LVQPGRSLRL SCAASGFRFD DYALHWVRQA PEKGLEWVSG ISWNSNNIGY AESVKGRFTI SRDTAKKSLY LQMNDLRAE DTALYYCVKD RHYDSSGYFV NGFDIWGQGT LVTVSAASTK GPSVFPLAPS SKSTSGGTAA LGCLVKDYFP E PVTVSWNS ...文字列: EVQLVESGGG LVQPGRSLRL SCAASGFRFD DYALHWVRQA PEKGLEWVSG ISWNSNNIGY AESVKGRFTI SRDTAKKSLY LQMNDLRAE DTALYYCVKD RHYDSSGYFV NGFDIWGQGT LVTVSAASTK GPSVFPLAPS SKSTSGGTAA LGCLVKDYFP E PVTVSWNS GALTSGVHTF PAVLQSSGLY SLSSVVTVPS SSLGTQTYIC NVNHKPSNTK VDKRVEPKSC |
-分子 #2: P2G3 Light Chain
| 分子 | 名称: P2G3 Light Chain / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 23.321771 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: DIQLTQSPSF LSASVGDRVT VTCRASQGIS SYVAWYQQKA GKAPTLLIYT ASTLQSGVPS RFSGSGSGTE FTLTISSLQP EDFATYYCQ QLHSYPVTFG QGTRLDIERT VAAPSVFIFP PSDEQLKSGT ASVVCLLNNF YPREAKVQWK VDNALQSGNS Q ESVTEQDS ...文字列: DIQLTQSPSF LSASVGDRVT VTCRASQGIS SYVAWYQQKA GKAPTLLIYT ASTLQSGVPS RFSGSGSGTE FTLTISSLQP EDFATYYCQ QLHSYPVTFG QGTRLDIERT VAAPSVFIFP PSDEQLKSGT ASVVCLLNNF YPREAKVQWK VDNALQSGNS Q ESVTEQDS KDSTYSLSST LTLSKADYEK HKVYACEVTH QGLSSPVTKS FNRGEC |
-分子 #3: Surface glycoprotein
| 分子 | 名称: Surface glycoprotein / タイプ: protein_or_peptide / ID: 3 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 142.558094 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MFVFLVLLPL VSSQCVNLTT RTQLPPAYTN SFTRGVYYPD KVFRSSVLHS TQDLFLPFFS NVTWFHVISG TNGTKRFDNP VLPFNDGVY FASIEKSNII RGWIFGTTLD SKTQSLLIVN NATNVVIKVC EFQFCNDPFL DHKNNKSWME SEFRVYSSAN N CTFEYVSQ ...文字列: MFVFLVLLPL VSSQCVNLTT RTQLPPAYTN SFTRGVYYPD KVFRSSVLHS TQDLFLPFFS NVTWFHVISG TNGTKRFDNP VLPFNDGVY FASIEKSNII RGWIFGTTLD SKTQSLLIVN NATNVVIKVC EFQFCNDPFL DHKNNKSWME SEFRVYSSAN N CTFEYVSQ PFLMDLEGKQ GNFKNLREFV FKNIDGYFKI YSKHTPIIVR EPEDLPQGFS ALEPLVDLPI GINITRFQTL LA LHRSYLT PGDSSSGWTA GAAAYYVGYL QPRTFLLKYN ENGTITDAVD CALDPLSETK CTLKSFTVEK GIYQTSNFRV QPT ESIVRF PNITNLCPFD EVFNATRFAS VYAWNRKRIS NCVADYSVLY NLAPFFTFKC YGVSPTKLND LCFTNVYADS FVIR GDEVR QIAPGQTGNI ADYNYKLPDD FTGCVIAWNS NKLDSKVSGN YNYLYRLFRK SNLKPFERDI STEIYQAGNK PCNGV AGFN CYFPLRSYSF RPTYGVGHQP YRVVVLSFEL LHAPATVCGP KKSTNLVKNK CVNFNFNGLK GTGVLTESNK KFLPFQ QFG RDIADTTDAV RDPQTLEILD ITPCSFGGVS VITPGTNTSN QVAVLYQGVN CTEVPVAIHA DQLTPTWRVY STGSNVF QT RAGCLIGAEY VNNSYECDIP IGAGICASYQ TQTKSHGSAS SVASQSIIAY TMSLGAENSV AYSNNSIAIP TNFTISVT T EILPVSMTKT SVDCTMYICG DSTECSNLLL QYGSFCTQLK RALTGIAVEQ DKNTQEVFAQ VKQIYKTPPI KYFGGFNFS QILPDPSKPS KRSFIEDLLF NKVTLADAGF IKQYGDCLGD IAARDLICAQ KFKGLTVLPP LLTDEMIAQY TSALLAGTIT SGWTFGAGA ALQIPFAMQM AYRFNGIGVT QNVLYENQKL IANQFNSAIG KIQDSLSSTA SALGKLQDVV NHNAQALNTL V KQLSSKFG AISSVLNDIF SRLDPPEAEV QIDRLITGRL QSLQTYVTQQ LIRAAEIRAS ANLAATKMSE CVLGQSKRVD FC GKGYHLM SFPQSAPHGV VFLHVTYVPA QEKNFTTAPA ICHDGKAHFP REGVFVSNGT HWFVTQRNFY EPQIITTDNT FVS GNCDVV IGIVNNTVYD PLQPELDSFK EELDKYFKNH TSPDVDLGDI SGINASVVNI QKEIDRLNEV AKNLNESLID LQEL GKYEQ GSGYIPEAPR DGQAYVRKDG EWVLLSTFLG RSLEVLFQGP GHHHHHHHHS AWSHPQFEKG GGSGGGGSGG SAWSH PQFE K |
-分子 #4: P5C3 Light Chain
| 分子 | 名称: P5C3 Light Chain / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 23.388875 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: EIVLTQSPGT LSLSPGERAT LSCRGSQSVR SSYLGWYQQK PGQAPRLLIY GASSRATGIP DRFSGSGSGT DFTLTISRLE PEDFAVYYC QQYGSSPWTF GQGTKVEIKG TVAAPSVFIF PPSDEQLKSG TASVVCLLNN FYPREAKVQW KVDNALQSGN S QESVTEQD ...文字列: EIVLTQSPGT LSLSPGERAT LSCRGSQSVR SSYLGWYQQK PGQAPRLLIY GASSRATGIP DRFSGSGSGT DFTLTISRLE PEDFAVYYC QQYGSSPWTF GQGTKVEIKG TVAAPSVFIF PPSDEQLKSG TASVVCLLNN FYPREAKVQW KVDNALQSGN S QESVTEQD SKDSTYSLSS TLTLSKADYE KHKVYACEVT HQGLSSPVTK SFNRGEC |
-分子 #5: P5C3 Heavy Chain
| 分子 | 名称: P5C3 Heavy Chain / タイプ: protein_or_peptide / ID: 5 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 23.362133 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: QMQLVQSGPE VKKPGTSVKV SCKASGFTFT SSAVQWVRQA RGQRLEWIGW IVVGSGNTDY AQQFQERVTI TRDMSTSTAY MELSSLGSE DTAVYYCAAP NCSGGSCYDG FDLWGQGTMV TVSSASTKGP SVFPLAPSSK STSGGTAALG CLVKDYFPEP V TVSWNSGA ...文字列: QMQLVQSGPE VKKPGTSVKV SCKASGFTFT SSAVQWVRQA RGQRLEWIGW IVVGSGNTDY AQQFQERVTI TRDMSTSTAY MELSSLGSE DTAVYYCAAP NCSGGSCYDG FDLWGQGTMV TVSSASTKGP SVFPLAPSSK STSGGTAALG CLVKDYFPEP V TVSWNSGA LTSGVHTFPA VLQSSGLYSL SSVVTVPSSS LGTQTYICNV NHKPSNTKVD KKV |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.7 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 / 詳細: PBS |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 400 |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)