+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | human Connexin 26 dodecamer at 20mmHg PCO2, pH7.4 | |||||||||
 マップデータ マップデータ | dodecameric assembly of human connexin 26 in 20mmHg PCO2 at pH7.4 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Gap junction / Ion Channel / carbon dioxide sensitive / MEMBRANE PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Transport of connexons to the plasma membrane / response to human chorionic gonadotropin / gap junction-mediated intercellular transport / gap junction channel activity involved in cell communication by electrical coupling / epididymis development / Oligomerization of connexins into connexons / Transport of connexins along the secretory pathway / gap junction assembly / connexin complex / gap junction ...Transport of connexons to the plasma membrane / response to human chorionic gonadotropin / gap junction-mediated intercellular transport / gap junction channel activity involved in cell communication by electrical coupling / epididymis development / Oligomerization of connexins into connexons / Transport of connexins along the secretory pathway / gap junction assembly / connexin complex / gap junction / astrocyte projection / Gap junction assembly / gap junction channel activity / endoplasmic reticulum-Golgi intermediate compartment / inner ear development / decidualization / lateral plasma membrane / response to retinoic acid / cellular response to glucagon stimulus / cellular response to dexamethasone stimulus / response to ischemia / response to progesterone / sensory perception of sound / transmembrane transport / cell-cell signaling / response to estradiol / cellular response to oxidative stress / cell body / response to lipopolysaccharide / calcium ion binding / perinuclear region of cytoplasm / identical protein binding / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
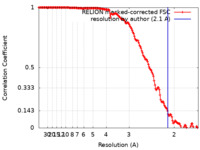
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.1 Å | |||||||||
 データ登録者 データ登録者 | Brotherton DH / Cameron AD / Savva CG / Ragan TJ | |||||||||
| 資金援助 |  英国, 2件 英国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2022 ジャーナル: Structure / 年: 2022タイトル: Conformational changes and CO-induced channel gating in connexin26. 著者: Deborah H Brotherton / Christos G Savva / Timothy J Ragan / Nicholas Dale / Alexander D Cameron /  要旨: Connexins form large-pore channels that function either as dodecameric gap junctions or hexameric hemichannels to allow the regulated movement of small molecules and ions across cell membranes. ...Connexins form large-pore channels that function either as dodecameric gap junctions or hexameric hemichannels to allow the regulated movement of small molecules and ions across cell membranes. Opening or closing of the channels is controlled by a variety of stimuli, and dysregulation leads to multiple diseases. An increase in the partial pressure of carbon dioxide (PCO) has been shown to cause connexin26 (Cx26) gap junctions to close. Here, we use cryoelectron microscopy (cryo-EM) to determine the structure of human Cx26 gap junctions under increasing levels of PCO. We show a correlation between the level of PCO and the size of the aperture of the pore, governed by the N-terminal helices that line the pore. This indicates that CO alone is sufficient to cause conformational changes in the protein. Analysis of the conformational states shows that movements at the N terminus are linked to both subunit rotation and flexing of the transmembrane helices. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_13940.map.gz emd_13940.map.gz | 14.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-13940-v30.xml emd-13940-v30.xml emd-13940.xml emd-13940.xml | 20.3 KB 20.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_13940_fsc.xml emd_13940_fsc.xml | 15.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_13940.png emd_13940.png | 113.3 KB | ||
| Filedesc metadata |  emd-13940.cif.gz emd-13940.cif.gz | 6.5 KB | ||
| その他 |  emd_13940_half_map_1.map.gz emd_13940_half_map_1.map.gz emd_13940_half_map_2.map.gz emd_13940_half_map_2.map.gz | 250.5 MB 250.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-13940 http://ftp.pdbj.org/pub/emdb/structures/EMD-13940 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13940 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13940 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_13940_validation.pdf.gz emd_13940_validation.pdf.gz | 686.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_13940_full_validation.pdf.gz emd_13940_full_validation.pdf.gz | 686.3 KB | 表示 | |
| XML形式データ |  emd_13940_validation.xml.gz emd_13940_validation.xml.gz | 23.1 KB | 表示 | |
| CIF形式データ |  emd_13940_validation.cif.gz emd_13940_validation.cif.gz | 30.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13940 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13940 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13940 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13940 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7qetMC  7qeoC  7qeqC  7qerC  7qesC  7qeuC  7qevC  7qewC  7qeyC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_13940.map.gz / 形式: CCP4 / 大きさ: 316.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_13940.map.gz / 形式: CCP4 / 大きさ: 316.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | dodecameric assembly of human connexin 26 in 20mmHg PCO2 at pH7.4 | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.85 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: half map 1 from D6 refinement of full data
| ファイル | emd_13940_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 1 from D6 refinement of full data | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map 2 from D6 refinement of full dataset
| ファイル | emd_13940_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 2 from D6 refinement of full dataset | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : dodecameric assembly of human connexion 26
| 全体 | 名称: dodecameric assembly of human connexion 26 |
|---|---|
| 要素 |
|
-超分子 #1: dodecameric assembly of human connexion 26
| 超分子 | 名称: dodecameric assembly of human connexion 26 / タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Gap junction beta-2 protein
| 分子 | 名称: Gap junction beta-2 protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 12 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 26.713674 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MDWGTLQTIL GGVNKHSTSI GKIWLTVLFI FRIMILVVAA KEVWGDEQAD FVCNTLQPGC KNVCYDHYFP ISHIRLWALQ LIFVSTPAL LVAMHVAYRR HEKKRKFIKG EIKSEFKDIE EIKTQKVRIE GSLWWTYTSS IFFRVIFEAA FMYVFYVMYD G FSMQRLVK ...文字列: MDWGTLQTIL GGVNKHSTSI GKIWLTVLFI FRIMILVVAA KEVWGDEQAD FVCNTLQPGC KNVCYDHYFP ISHIRLWALQ LIFVSTPAL LVAMHVAYRR HEKKRKFIKG EIKSEFKDIE EIKTQKVRIE GSLWWTYTSS IFFRVIFEAA FMYVFYVMYD G FSMQRLVK CNAWPCPNTV DCFVSRPTEK TVFTVFMIAV SGICILLNVT ELCYLLIRYC SGKSKKPVLV PR UniProtKB: Gap junction beta-2 protein |
-分子 #2: DODECYL-BETA-D-MALTOSIDE
| 分子 | 名称: DODECYL-BETA-D-MALTOSIDE / タイプ: ligand / ID: 2 / コピー数: 48 / 式: LMT |
|---|---|
| 分子量 | 理論値: 510.615 Da |
| Chemical component information |  ChemComp-LMT: |
-分子 #3: PHOSPHATIDYLETHANOLAMINE
| 分子 | 名称: PHOSPHATIDYLETHANOLAMINE / タイプ: ligand / ID: 3 / コピー数: 12 / 式: PTY |
|---|---|
| 分子量 | 理論値: 734.039 Da |
| Chemical component information |  ChemComp-PTY: |
-分子 #4: water
| 分子 | 名称: water / タイプ: ligand / ID: 4 / コピー数: 305 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 3.5 mg/mL | ||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.4 構成要素:
詳細: The buffer, except DDM and DTT, was prepared fresh from 10x stock on day of use. The basal buffer was filtered and de-gassed, and DDM and DTT added. The buffer was pH corrected at point of ...詳細: The buffer, except DDM and DTT, was prepared fresh from 10x stock on day of use. The basal buffer was filtered and de-gassed, and DDM and DTT added. The buffer was pH corrected at point of use to 7.4 using 2.5% CO2 in 97.5% N2. | ||||||||||||||||||||||||||||||
| グリッド | モデル: Quantifoil R0.6/1 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: GOLD / 支持フィルム - トポロジー: HOLEY / 支持フィルム - Film thickness: 50 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. / 前処理 - 雰囲気: AIR | ||||||||||||||||||||||||||||||
| 凍結 | 凍結剤: ETHANE-PROPANE / チャンバー内湿度: 95 % / チャンバー内温度: 277 K / 装置: LEICA EM GP 詳細: 3 microlitres protein applied to grid, blot time 6 seconds, in 2.5% CO2/97.5%N2 atmosphere. | ||||||||||||||||||||||||||||||
| 詳細 | This sample monodisperse |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 1 / 実像数: 3305 / 平均露光時間: 3.0 sec. / 平均電子線量: 45.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)