+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-13638 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of pentameric S-layer protein from Halofaerax volcanii | ||||||||||||
 マップデータ マップデータ | Non-postprocessed, non-sharpened full map. | ||||||||||||
 試料 試料 |
| ||||||||||||
| 機能・相同性 | Surface glycoprotein signal peptide / Major cell surface glycoprotein / PGF-CTERM archaeal protein-sorting signal / PGF-CTERM motif / S-layer / cell wall organization / extracellular region / plasma membrane / Cell surface glycoprotein 機能・相同性情報 機能・相同性情報 | ||||||||||||
| 生物種 |  Haloferax volcanii DS2 (古細菌) Haloferax volcanii DS2 (古細菌) | ||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.87 Å | ||||||||||||
 データ登録者 データ登録者 | von Kuegelgen A / Bharat TAM | ||||||||||||
| 資金援助 |  英国, 3件 英国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Cell Rep / 年: 2021 ジャーナル: Cell Rep / 年: 2021タイトル: Complete atomic structure of a native archaeal cell surface. 著者: Andriko von Kügelgen / Vikram Alva / Tanmay A M Bharat /   要旨: Many prokaryotic cells are covered by an ordered, proteinaceous, sheet-like structure called a surface layer (S-layer). S-layer proteins (SLPs) are usually the highest copy number macromolecules in ...Many prokaryotic cells are covered by an ordered, proteinaceous, sheet-like structure called a surface layer (S-layer). S-layer proteins (SLPs) are usually the highest copy number macromolecules in prokaryotes, playing critical roles in cellular physiology such as blocking predators, scaffolding membranes, and facilitating environmental interactions. Using electron cryomicroscopy of two-dimensional sheets, we report the atomic structure of the S-layer from the archaeal model organism Haloferax volcanii. This S-layer consists of a hexagonal array of tightly interacting immunoglobulin-like domains, which are also found in SLPs across several classes of archaea. Cellular tomography reveal that the S-layer is nearly continuous on the cell surface, completed by pentameric defects in the hexagonal lattice. We further report the atomic structure of the SLP pentamer, which shows markedly different relative arrangements of SLP domains needed to complete the S-layer. Our structural data provide a framework for understanding cell surfaces of archaea at the atomic level. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_13638.map.gz emd_13638.map.gz | 96.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-13638-v30.xml emd-13638-v30.xml emd-13638.xml emd-13638.xml | 26.4 KB 26.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_13638.png emd_13638.png | 140.9 KB | ||
| マスクデータ |  emd_13638_msk_1.map emd_13638_msk_1.map | 125 MB |  マスクマップ マスクマップ | |
| その他 |  emd_13638_additional_1.map.gz emd_13638_additional_1.map.gz emd_13638_half_map_1.map.gz emd_13638_half_map_1.map.gz emd_13638_half_map_2.map.gz emd_13638_half_map_2.map.gz | 116.9 MB 98 MB 98 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-13638 http://ftp.pdbj.org/pub/emdb/structures/EMD-13638 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13638 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13638 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_13638_validation.pdf.gz emd_13638_validation.pdf.gz | 448 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_13638_full_validation.pdf.gz emd_13638_full_validation.pdf.gz | 447.5 KB | 表示 | |
| XML形式データ |  emd_13638_validation.xml.gz emd_13638_validation.xml.gz | 13.2 KB | 表示 | |
| CIF形式データ |  emd_13638_validation.cif.gz emd_13638_validation.cif.gz | 15.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13638 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13638 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13638 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13638 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_13638.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_13638.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Non-postprocessed, non-sharpened full map. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.1 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_13638_msk_1.map emd_13638_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
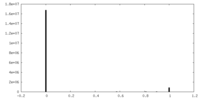
| 投影像・断面図 |
| ||||||||||||
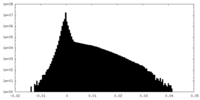
| 密度ヒストグラム |
-追加マップ: Postprocessed, sharpened map with an applied B-factor of -170 A^2
| ファイル | emd_13638_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Postprocessed, sharpened map with an applied B-factor of -170 A^2 | ||||||||||||
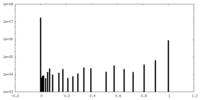
| 投影像・断面図 |
| ||||||||||||
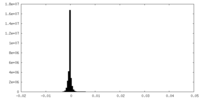
| 密度ヒストグラム |
-ハーフマップ: Half map 1, non-postprocessed, non sharpened.
| ファイル | emd_13638_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 1, non-postprocessed, non sharpened. | ||||||||||||
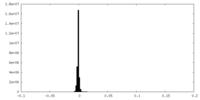
| 投影像・断面図 |
| ||||||||||||
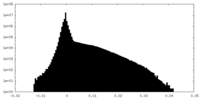
| 密度ヒストグラム |
-ハーフマップ: Half map 2, non-postprocessed, non sharpened.
| ファイル | emd_13638_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 2, non-postprocessed, non sharpened. | ||||||||||||
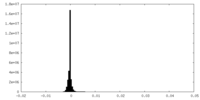
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Structure of pentameric S-layer protein csg
| 全体 | 名称: Structure of pentameric S-layer protein csg |
|---|---|
| 要素 |
|
-超分子 #1: Structure of pentameric S-layer protein csg
| 超分子 | 名称: Structure of pentameric S-layer protein csg / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 / 詳細: Structure of pentameric S-layer protein csg |
|---|---|
| 由来(天然) | 生物種:  Haloferax volcanii DS2 (古細菌) / 細胞中の位置: Cell surface Haloferax volcanii DS2 (古細菌) / 細胞中の位置: Cell surface |
-分子 #1: Cell surface glycoprotein
| 分子 | 名称: Cell surface glycoprotein / タイプ: protein_or_peptide / ID: 1 / コピー数: 5 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Haloferax volcanii DS2 (古細菌) Haloferax volcanii DS2 (古細菌) |
| 分子量 | 理論値: 81.755602 KDa |
| 配列 | 文字列: ERGNLDADSE SFNKTIQSGD RVFLGEEIST DAGLGASNPL LTGTAGNSEG VSLDLSSPIP QTTENQPLGT YDVDGSGSAT TPNVTLLAP RITDSEILTS SGGDVTGSAI SSSDAGNLYV NADYNYESAE KVEVTVEDPS GTDITNEVLS GTDTFVDDGS I GSTSSTGG ...文字列: ERGNLDADSE SFNKTIQSGD RVFLGEEIST DAGLGASNPL LTGTAGNSEG VSLDLSSPIP QTTENQPLGT YDVDGSGSAT TPNVTLLAP RITDSEILTS SGGDVTGSAI SSSDAGNLYV NADYNYESAE KVEVTVEDPS GTDITNEVLS GTDTFVDDGS I GSTSSTGG GVGIDMSDQD AGEYTIILEG AEDLDFGDAT ETMTLTISSQ DEIGIELDSE SVTQGTDVQY TVTNGIDGNE HV VAMDLSD LQNDATTEQA KEVFRNIGDT SEVGIANSSA TNTSGSSTGP TVETADIAYA VVEIDGASAV GGIETQYLDD SEV DLEVYD AGVSATAAVG QDATNDITLT IEEGGTTLSS PTGQYVVGSE VDINGTATSS DSVAIYVRDD GDWQLLEIGG DNEI SVDSD DTFEEEDIAL SGLSGDGSSI LSLTGTYRIG VIDASDADVG GDGSVDDSLT TSEFTSGVSS SNSIRVTDQA LTGQF TTIN GQVAPVETGT VDINGTASGA NSVLVIFVDE RGNVNYQEVS VDSDGTYDED DITVGLTQGR VTAHILSVGR DSAIGD GSL PSGPSNGATL NDLTGYLDTL DQNNNNGEQI NELIASETVD ETASDDLIVT ETFRLAESST SIDSIYPDAA EAAGINP VA TGETMVIAGS TNLKPDDNTI SIEVTNEDGT SVALEDTDEW NNDGQWMVEI DTTDFETGTF TVEADDGDNT DTVNVEVV S EREDTTTSSD NATDTTTTTD GPTETTTTAE PTETTEEPTE ETTTSSNTPG FGIAVALVAL VGAALLALRR EN |
-分子 #2: beta-D-glucopyranose
| 分子 | 名称: beta-D-glucopyranose / タイプ: ligand / ID: 2 / コピー数: 5 / 式: BGC |
|---|---|
| 分子量 | 理論値: 180.156 Da |
| Chemical component information |  ChemComp-BGC: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2.5 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
詳細: Buffer solutions were prepared fresh from sterile filtered concentrated stocksolutions. Solutions were filtered through a 0.22 um filter to avoid microbial contamination and degassed using a ...詳細: Buffer solutions were prepared fresh from sterile filtered concentrated stocksolutions. Solutions were filtered through a 0.22 um filter to avoid microbial contamination and degassed using a vacuum fold pump. The pH of the HEPES stock solution was adjusted with sodium hydroxide at 4 deg C. 1.75 mM holmium chloride was added 2 hours before vitrification. | |||||||||||||||
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER/RHODIUM / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR / 詳細: 20 seconds, 15 mA | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 283.15 K / 装置: FEI VITROBOT MARK IV 詳細: Vitrobot options: Blot time 5 seconds, Blot force -10,1, Wait time 10 seconds, Drain time 0.5 seconds. | |||||||||||||||
| 詳細 | Purified csg protein mixed with 1.75 mM HoCl3 after 2 hour incubation. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 温度 | 最低: 70.0 K / 最高: 70.0 K |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum LS / エネルギーフィルター - スリット幅: 20 eV |
| 詳細 | EPU software with faster acquisition mode AFIS (Aberration Free Image Shift). |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) デジタル化 - サイズ - 横: 5760 pixel / デジタル化 - サイズ - 縦: 4092 pixel / 撮影したグリッド数: 2 / 実像数: 11871 / 平均露光時間: 3.6 sec. / 平均電子線量: 53.9 e/Å2 詳細: Images were collected in two sessions in movie-mode and subjected to 3.6 seconds of exposure where a total dose of 53.45 or 53.9 e-/A2 was applied, and 40 frames were recorded per movie. A ...詳細: Images were collected in two sessions in movie-mode and subjected to 3.6 seconds of exposure where a total dose of 53.45 or 53.9 e-/A2 was applied, and 40 frames were recorded per movie. A total of 11871 movies were collected in two sessions with the same microscope and settings. |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 最大 デフォーカス(補正後): 4.0 µm / 最小 デフォーカス(補正後): 1.0 µm / 倍率(補正後): 81000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 4.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 81000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 詳細 | The initial manual build of D1-D2 was performed independently using the csg pentameric cryo-EM map, which served as an additional validation of the manual building performed in the csg hexamer in the related deposition. The manual building exercise yielded a nearly identical result to the hexamer; thus, the final refined hexameric structures of D1-D2, along with D3-D4 were taken and fitted into the pentameric map (~3.87 angstrom resolution in D1-D3, lower in D4 which is partially resolved). Five copies of these D1-D4 were used for refinement and model building as for the hexamer, except D3 and D4 was restrained in position, due to steadily deteriorating resolution in this part of the map. D5-D6 were not resolved in the pentameric structure and were thus not included in the refinements. Model validation was performed in PHENIX and CCP-EM. |
|---|---|
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL / 温度因子: 179.68 / 当てはまり具合の基準: Best Fit |
| 得られたモデル |  PDB-7ptu: |
 ムービー
ムービー コントローラー
コントローラー

















 Z
Z Y
Y X
X