+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-12819 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Hepatitis B core protein | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Hepatitis B core protein / VIRUS LIKE PARTICLE | |||||||||
| 機能・相同性 | Hepatitis B virus, capsid N-terminal / Hepatitis core protein, putative zinc finger / Hepatitis core antigen / Viral capsid core domain supefamily, Hepatitis B virus / Hepatitis core antigen / virus-mediated perturbation of host defense response / structural molecule activity / extracellular region / External core antigen 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |   Hepatitis B virus (B 型肝炎ウイルス) Hepatitis B virus (B 型肝炎ウイルス) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.8 Å | |||||||||
 データ登録者 データ登録者 | Bottcher B / Makbul C | |||||||||
| 資金援助 |  ドイツ, 1件 ドイツ, 1件
| |||||||||
 引用 引用 |  ジャーナル: Microorganisms / 年: 2021 ジャーナル: Microorganisms / 年: 2021タイトル: Conformational Plasticity of Hepatitis B Core Protein Spikes Promotes Peptide Binding Independent of the Secretion Phenotype. 著者: Cihan Makbul / Vladimir Khayenko / Hans Michael Maric / Bettina Böttcher /  要旨: Hepatitis B virus is a major human pathogen, which forms enveloped virus particles. During viral maturation, membrane-bound hepatitis B surface proteins package hepatitis B core protein capsids. This ...Hepatitis B virus is a major human pathogen, which forms enveloped virus particles. During viral maturation, membrane-bound hepatitis B surface proteins package hepatitis B core protein capsids. This process is intercepted by certain peptides with an "LLGRMKG" motif that binds to the capsids at the tips of dimeric spikes. With microcalorimetry, electron cryo microscopy and peptide microarray-based screens, we have characterized the structural and thermodynamic properties of peptide binding to hepatitis B core protein capsids with different secretion phenotypes. The peptide "GSLLGRMKGA" binds weakly to hepatitis B core protein capsids and mutant capsids with a premature (F97L) or low-secretion phenotype (L60V and P5T). With electron cryo microscopy, we provide novel structures for L60V and P5T and demonstrate that binding occurs at the tips of the spikes at the dimer interface, splaying the helices apart independent of the secretion phenotype. Peptide array screening identifies "SLLGRM" as the core binding motif. This shortened motif binds only to one of the two spikes in the asymmetric unit of the capsid and induces a much smaller conformational change. Altogether, these comprehensive studies suggest that the tips of the spikes act as an autonomous binding platform that is unaffected by mutations that affect secretion phenotypes. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_12819.map.gz emd_12819.map.gz | 228.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-12819-v30.xml emd-12819-v30.xml emd-12819.xml emd-12819.xml | 18.5 KB 18.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_12819_fsc.xml emd_12819_fsc.xml | 15.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_12819.png emd_12819.png | 215.4 KB | ||
| マスクデータ |  emd_12819_msk_1.map emd_12819_msk_1.map | 325 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-12819.cif.gz emd-12819.cif.gz | 5.8 KB | ||
| その他 |  emd_12819_half_map_1.map.gz emd_12819_half_map_1.map.gz emd_12819_half_map_2.map.gz emd_12819_half_map_2.map.gz | 256.9 MB 256.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-12819 http://ftp.pdbj.org/pub/emdb/structures/EMD-12819 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12819 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12819 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_12819_validation.pdf.gz emd_12819_validation.pdf.gz | 1.1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_12819_full_validation.pdf.gz emd_12819_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  emd_12819_validation.xml.gz emd_12819_validation.xml.gz | 23.7 KB | 表示 | |
| CIF形式データ |  emd_12819_validation.cif.gz emd_12819_validation.cif.gz | 30.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12819 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12819 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12819 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12819 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_12819.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_12819.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.0635 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_12819_msk_1.map emd_12819_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
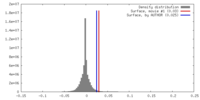
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_12819_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_12819_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Hepatitis B virus
| 全体 | 名称:   Hepatitis B virus (B 型肝炎ウイルス) Hepatitis B virus (B 型肝炎ウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Hepatitis B virus
| 超分子 | 名称: Hepatitis B virus / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all / NCBI-ID: 10407 / 生物種: Hepatitis B virus / Sci species strain: genotype D subtype ayw / ウイルスタイプ: VIRUS-LIKE PARTICLE / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 4.8 MDa |
| ウイルス殻 | Shell ID: 1 / 名称: capsid / 直径: 360.0 Å / T番号(三角分割数): 4 |
-分子 #1: External core antigen
| 分子 | 名称: External core antigen / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Hepatitis B virus (B 型肝炎ウイルス) Hepatitis B virus (B 型肝炎ウイルス) |
| 分子量 | 理論値: 21.146217 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MDIDPYKEFG ATVELLSFLP SDFFPSVRDL LDTASALYRE ALESPEHCSP HHTALRQAIL CWGELMTLAT WVGVNLEDPA SRDLVVSYV NTNMGLKFRQ LLWFHISCLT FGRETVIEYL VSFGVWIRTP PAYRPPNAPI LSTLPETTVV RRRGRSPRRR T PSPRRRRS QSPRRRRSQS RESQC UniProtKB: External core antigen |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 時間: 120 sec. / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 0.029 kPa |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: INTEGRATING / 撮影したグリッド数: 1 / 実像数: 2423 / 平均露光時間: 2.5 sec. / 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.6 µm / 最小 デフォーカス(公称値): 0.6 µm / 倍率(公称値): 75000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル |
| ||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT / 温度因子: 106 | ||||||||||
| 得られたモデル |  PDB-7od4: |
 ムービー
ムービー コントローラー
コントローラー





























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)