+ Open data
Open data
- Basic information
Basic information
| Entry | Database: EMDB / ID: EMD-11429 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Title | bovine ATP synthase dimer state1:state2 | |||||||||
 Map data Map data | ATP synthase dimer state1:state2 main map | |||||||||
 Sample Sample |
| |||||||||
| Function / homology |  Function and homology information Function and homology informationnegative regulation of mitochondrial ATP synthesis coupled proton transport / angiostatin binding / Formation of ATP by chemiosmotic coupling / Cristae formation / negative regulation of hydrolase activity / ATPase inhibitor activity / mitochondrial proton-transporting ATP synthase complex assembly / mitochondrial envelope / proton channel activity / heme biosynthetic process ...negative regulation of mitochondrial ATP synthesis coupled proton transport / angiostatin binding / Formation of ATP by chemiosmotic coupling / Cristae formation / negative regulation of hydrolase activity / ATPase inhibitor activity / mitochondrial proton-transporting ATP synthase complex assembly / mitochondrial envelope / proton channel activity / heme biosynthetic process / Mitochondrial protein degradation / negative regulation of endothelial cell proliferation / proton transmembrane transporter activity / proton motive force-driven ATP synthesis / proton-transporting two-sector ATPase complex, proton-transporting domain / proton motive force-driven mitochondrial ATP synthesis / H+-transporting two-sector ATPase / proton-transporting ATP synthase complex / proton-transporting ATP synthase activity, rotational mechanism / proton transmembrane transport / aerobic respiration / erythrocyte differentiation / ADP binding / mitochondrial membrane / ATPase binding / protein homotetramerization / calmodulin binding / mitochondrial inner membrane / lipid binding / structural molecule activity / cell surface / protein homodimerization activity / ATP hydrolysis activity / protein-containing complex / mitochondrion / ATP binding / identical protein binding / plasma membrane / cytoplasm Similarity search - Function | |||||||||
| Biological species |  | |||||||||
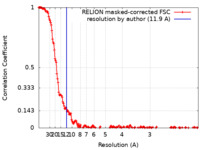
| Method | single particle reconstruction / cryo EM / Resolution: 11.9 Å | |||||||||
 Authors Authors | Spikes TE / Montgomery MG / Walker JE | |||||||||
| Funding support |  United Kingdom, 2 items United Kingdom, 2 items
| |||||||||
 Citation Citation |  Journal: Proc Natl Acad Sci U S A / Year: 2021 Journal: Proc Natl Acad Sci U S A / Year: 2021Title: Interface mobility between monomers in dimeric bovine ATP synthase participates in the ultrastructure of inner mitochondrial membranes. Authors: Tobias E Spikes / Martin G Montgomery / John E Walker /  Abstract: The ATP synthase complexes in mitochondria make the ATP required to sustain life by a rotary mechanism. Their membrane domains are embedded in the inner membranes of the organelle, and they dimerize ...The ATP synthase complexes in mitochondria make the ATP required to sustain life by a rotary mechanism. Their membrane domains are embedded in the inner membranes of the organelle, and they dimerize via interactions between their membrane domains. The dimers form extensive chains along the tips of the cristae with the two rows of monomeric catalytic domains extending into the mitochondrial matrix at an angle to each other. Disruption of the interface between dimers by mutation affects the morphology of the cristae severely. By analysis of particles of purified dimeric bovine ATP synthase by cryo-electron microscopy, we have shown that the angle between the central rotatory axes of the monomeric complexes varies between ca. 76 and 95°. These particles represent active dimeric ATP synthase. Some angular variations arise directly from the catalytic mechanism of the enzyme, and others are independent of catalysis. The monomer-monomer interaction is mediated mainly by j subunits attached to the surface of wedge-shaped protein-lipid structures in the membrane domain of the complex, and the angular variation arises from rotational and translational changes in this interaction, and combinations of both. The structures also suggest how the dimeric ATP synthases might be interacting with each other to form the characteristic rows along the tips of the cristae via other interwedge contacts, molding themselves to the range of oligomeric arrangements observed by tomography of mitochondrial membranes, and at the same time allowing the ATP synthase to operate under the range of physiological conditions that influence the structure of the cristae. | |||||||||
| History |
|
- Structure visualization
Structure visualization
| Movie |
 Movie viewer Movie viewer |
|---|---|
| Structure viewer | EM map:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| Supplemental images |
- Downloads & links
Downloads & links
-EMDB archive
| Map data |  emd_11429.map.gz emd_11429.map.gz | 445.2 MB |  EMDB map data format EMDB map data format | |
|---|---|---|---|---|
| Header (meta data) |  emd-11429-v30.xml emd-11429-v30.xml emd-11429.xml emd-11429.xml | 35.9 KB 35.9 KB | Display Display |  EMDB header EMDB header |
| FSC (resolution estimation) |  emd_11429_fsc.xml emd_11429_fsc.xml | 18 KB | Display |  FSC data file FSC data file |
| Images |  emd_11429.png emd_11429.png | 53.9 KB | ||
| Masks |  emd_11429_msk_1.map emd_11429_msk_1.map | 476.8 MB |  Mask map Mask map | |
| Others |  emd_11429_half_map_1.map.gz emd_11429_half_map_1.map.gz emd_11429_half_map_2.map.gz emd_11429_half_map_2.map.gz | 384.6 MB 384.3 MB | ||
| Archive directory |  http://ftp.pdbj.org/pub/emdb/structures/EMD-11429 http://ftp.pdbj.org/pub/emdb/structures/EMD-11429 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11429 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11429 | HTTPS FTP |
-Related structure data
| Related structure data |  7ajcMC  7ajbC  7ajdC  7ajeC  7ajfC  7ajgC  7ajhC  7ajiC  7ajjC C: citing same article ( M: atomic model generated by this map |
|---|---|
| Similar structure data |
- Links
Links
| EMDB pages |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| Related items in Molecule of the Month |
- Map
Map
| File |  Download / File: emd_11429.map.gz / Format: CCP4 / Size: 476.8 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) Download / File: emd_11429.map.gz / Format: CCP4 / Size: 476.8 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Annotation | ATP synthase dimer state1:state2 main map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Projections & slices | Image control
Images are generated by Spider. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Voxel size | X=Y=Z: 1.048 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Density |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Symmetry | Space group: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Details | EMDB XML:
CCP4 map header:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-Supplemental data
-Mask #1
| File |  emd_11429_msk_1.map emd_11429_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Projections & Slices |
| ||||||||||||
| Density Histograms |
-Half map: ATP synthase dimer state1:state2 half map 2
| File | emd_11429_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Annotation | ATP synthase dimer state1:state2 half map 2 | ||||||||||||
| Projections & Slices |
| ||||||||||||
| Density Histograms |
-Half map: ATP synthase dimer state1:state2 half map 1
| File | emd_11429_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Annotation | ATP synthase dimer state1:state2 half map 1 | ||||||||||||
| Projections & Slices |
| ||||||||||||
| Density Histograms |
- Sample components
Sample components
+Entire : Bovine dimeric ATP synthase
+Supramolecule #1: Bovine dimeric ATP synthase
+Supramolecule #2: monomeric bovine ATP synthase
+Supramolecule #3: ATP synthase
+Macromolecule #1: ATP synthase alpha subunit
+Macromolecule #2: ATP synthase beta subunit
+Macromolecule #3: ATP synthase gamma subunit
+Macromolecule #4: ATP synthase delta subunit
+Macromolecule #5: ATP synthase epsilon subunit
+Macromolecule #6: ATP synthase a subunit
+Macromolecule #7: ATP synthase b subunit
+Macromolecule #8: ATP synthase c subunit
+Macromolecule #9: ATP synthase d subunit
+Macromolecule #10: ATP synthase e subunit
+Macromolecule #11: ATP synthase f subunit
+Macromolecule #12: ATP synthase g subunit
+Macromolecule #13: ATP synthase F6/h subunit
+Macromolecule #14: ATP synthase j subunit (6.8PL)
+Macromolecule #15: ATP synthase k subunit (DAPIT)
+Macromolecule #16: ATP synthase OSCP subunit
+Macromolecule #17: ATP synthase A6L/ATP8 subunit
+Macromolecule #18: ATP synthase inhibitor protein IF1
-Experimental details
-Structure determination
| Method | cryo EM |
|---|---|
 Processing Processing | single particle reconstruction |
| Aggregation state | particle |
- Sample preparation
Sample preparation
| Concentration | 4.5 mg/mL |
|---|---|
| Buffer | pH: 7.4 |
| Vitrification | Cryogen name: ETHANE / Chamber humidity: 100 % / Chamber temperature: 294 K / Instrument: FEI VITROBOT MARK IV Details: The sample was allowed to penetrate through the holey support and to distribute to both sides of the grid surface for ca. 15 sec. Then the grids were blotted with filter paper for 8-10 sec before blotting.. |
| Details | Nickel affinity purified filled by gel filtration |
- Electron microscopy
Electron microscopy
| Microscope | FEI TITAN KRIOS |
|---|---|
| Image recording | Film or detector model: GATAN K2 QUANTUM (4k x 4k) / Detector mode: COUNTING / Average exposure time: 12.0 sec. / Average electron dose: 4.6 e/Å2 |
| Electron beam | Acceleration voltage: 300 kV / Electron source:  FIELD EMISSION GUN FIELD EMISSION GUN |
| Electron optics | Illumination mode: FLOOD BEAM / Imaging mode: BRIGHT FIELD |
| Sample stage | Specimen holder model: FEI TITAN KRIOS AUTOGRID HOLDER / Cooling holder cryogen: NITROGEN |
| Experimental equipment |  Model: Titan Krios / Image courtesy: FEI Company |
+ Image processing
Image processing
-Atomic model buiding 1
| Initial model |
| ||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Space: REAL / Protocol: RIGID BODY FIT | ||||||||||||||||||||||||||||
| Output model |  PDB-7ajc: |
 Movie
Movie Controller
Controller




















































































 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)